利用AI对抗体进行设计文献有很多,让人眼花缭乱,也读不过来。编者喜欢有实验验证的工作或论文,下文会选取几篇优秀的有实验验证的论文进行介绍。纯算法的论文仅简单列在3.5小节。
现如今,许多深度学习算法应用于抗体工程问题。本文首先解释该领域中使用的主要数据集,以及它们包含和对从业者提供的价值。然后,将介绍抗体与深度学习领域中的几类广为人知的模型。每个部分都旨在加深我们对该问题的理解,而不是完全解释每种方法的基础!这个领域发展迅速,而这篇文章在几个月内肯定会过时,但通过阅读这些论文,我们建立的直觉将继续有用。
3.1 数据集
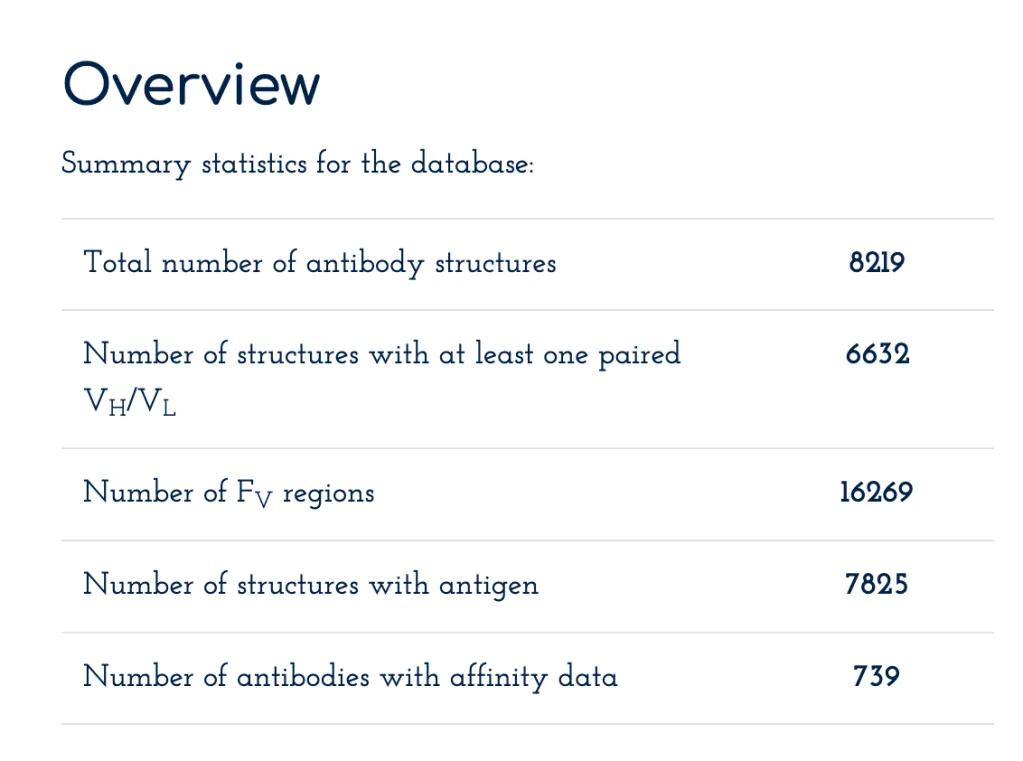
这里只有两个主要数据集OAS和SAbDab。
OAS (Observed Antibody Space)
https://opig.stats.ox.ac.uk/webapps/oas/documentation/
OAS是从下一代测序实验中收集的全面抗体数据。需要注意的是,测序仅限于Fab区域,主要位于重链可变区域(VHC)和轻链可变区域(VLC)。对于数据集的一个显著特点,在于一些通用信息被捕获,允许分配同种型。该数据的主要价值来自可变区域的序列,这有助于描述所有“可能”抗体序列的确切空间,特别是在CDR区域中,这有助于研究人员评估生成的抗体与天然抗体之间的多样性。数据集还包含来自非传统抗体(如纳米抗体)的数据,但占少数。
该数据集分为两部分:不配对的序列和配对的序列。不配对的序列的VHC和VLC序列是分开的,无法相互关联。这是测序技术限制的不幸后果;捕捉相互链接的蛋白质序列(正如在抗体中所发生的那样)在大规模高通量情况下是具有挑战性的!测序技术的进步意味着我们这些天也可以测序配对的序列,从同一抗体中得到VHC和VLC序列相互关联,但规模要小得多。而 OAS有30亿未配对的VHC/VLC序列,只有12万配对的VHC/VLC序列。
关于该数据集的一个不足之处,以及许多类似数据集的不足之处,是它源于“天然”的B细胞BCR,而不是原始抗体!前文已经介绍了BCR,但什么是天然B细胞?我们也讨论了B细胞的活化,即当B细胞遇到与其唯一BCR结合的抗原并转化成“激活”的B细胞时,它就开始产生数十亿的抗体并开始复制。还没有提到的是,该复制过程在CDR区域特意容易出错,在该区域,出错的可能性通常比正常的B细胞分裂高一百万倍。这意味着“激活”B细胞的“子代”产生的抗体异常多样。更重要的是,这些子细胞通常对其应该结合的抗原具有更高的结合亲和力,胜过其“母亲”B细胞。不幸的是,激活的B细胞在您的身体中占少数,这使得通过测序方法对它们进行抽样具有挑战性。这导致了一个抽样问题,OAS主要由所谓的“生殖细胞株” B细胞组成,它们在BCR中预期的多样性和结合亲和力上有一个上限,因为生殖细胞株通常对给定抗原具有低亲和力。在大规模LLM时代,这种数据集偏见可能会导致潜在问题,此问题有一篇论文报道(AbLang2, https://doi.org/10.1101/2024.02.02.578678)。抗体 AI 论文也经常提到这个问题,但目前并不是一个很大的问题。
SAbDab
https://opig.stats.ox.ac.uk/webapps/sabdab-sabpred/sabdab
SAbDab抗体结构数据库,从PDB中整理了经实验证实的抗体结构。与OAS一样,这里也包括更多外形奇特的抗体,如纳米抗体,但这只是少数。以下是一些关于数据集大小的统计数据,它比仅包含序列的数据集要小得多。该数据集主要用于依赖于结构信息的模型,例如抗体-抗原复合物。
3.2 Peter Kim|语言模型与抗体设计
使用蛋白语言模型对蛋白进行改造或突变设计,我会简单介绍下面的两篇Peter Kim团队的两篇文章。
Nature Biotech|pLM进化抗体

第一篇论文基本想法是利用已经通过优化过程的FDA批准的抗体,并通过LLM推荐的突变体来重新优化它们。作者这样做的方式是计算每个VHC和VLC的每个单氨基酸替换的对数似然率(log-likelihood)。这个log-likelihood打分怎么理解呢,比如将VHC上的甘氨酸替换为半胱氨酸,然后询问某一个pLM,突变体本是否“更有可能存在于自然界”。这是pLM非常擅长回答的问题,因为它们已经在所有曾经进化过的蛋白质上进行了训练。该论文总共使用了六个pLM(ESM-1b和ESM-1v的五个模型),并取得了所有6个模型的共识,即6个模型的打分都一致认为该突变更可能存在于自然界。他们进行了两轮优化,第一轮只突变一个残基,第二轮在前一轮的基础上再突变另一个残基。重要的是,没有对这些网络进行额外的训练,作者使用的预训练权重。本文的策略总结一下如下:
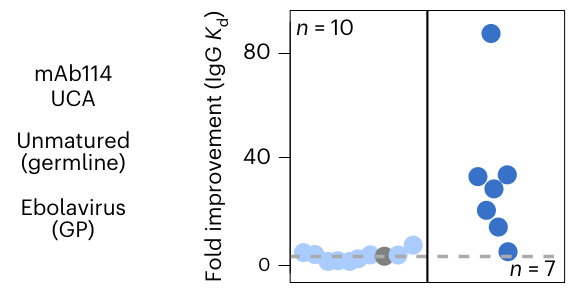
可见,本文的方法如此简单纯粹,但实验上却取得了非凡的结果,一些结果令人印象深刻:
作者从已经在临床使用中的七种抗体开始,因此这些抗体已经经过了为了它们的结合亲和力和物理特性进行优化的阶段。即使是这些抗体,pLM建议的突变,也具有更好的热稳定性,较低的免疫原性,而且在每一种情况下都具有更好的中和效力。这是在每种情况下为每种情况得分少于20种变体,以及经过两轮传统实验室进化之后的情况。对于这种情况来说,这个工作量比理性设计、定向进化的工作量要少得多。
这说明,在生命科学的深度学习中,一个相当普遍的经验是,模型在优化已经具有期望特性的事物方面非常擅长,但在首次(de novo)找到这些事物方面却非常糟糕。类似的现象也在其他领域出现,如Binder生成,小分子生成等。
这项研究的最后一个有趣部分是,绝大多数有益的突变不在CDR区域!尽管一直在讨论CDR的重要性,但利用CDR区域之外的突变可能会有相当大的价值。
Science|ESM-IF1进化抗体
接下来我们来看Peter Kim团队最新发表在Science的后续工作,同样是对抗体进行突变进化。这篇Science论文处理方式基本和Nature Biotech基本上是一模一样,只是将蛋白语言模型pLM换成了蕴含结构信息的语言模型(structure-informed language model),实质上是ESM-IF1.

这篇Science文章有几个重要的结论或经验,值得强调。
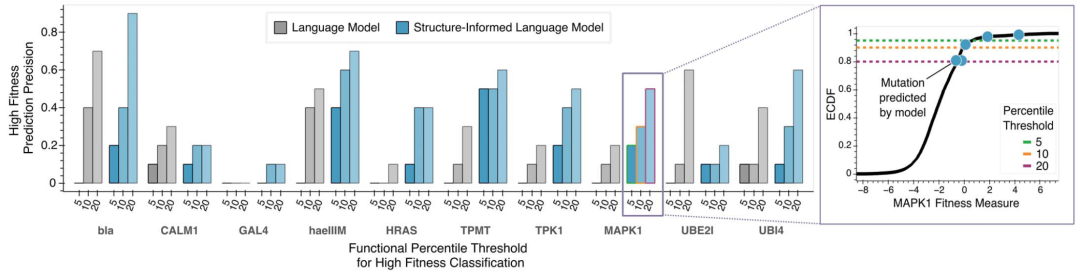
第一个结论,是蛋白的多模态(序列+结构)输入,i.e., ESM-IF1,能够更好的预测蛋白的fitness变化,作者并做了实验验证。见下图,在5/10/20点位突变替换的场景下,蓝色的ESM-IF1,要比仅用序列进行预训练的模型表现更好。这个结论也和最新ProteinGym的基准测试的结果一致,融合结构的语言模型SaProt表现很好。
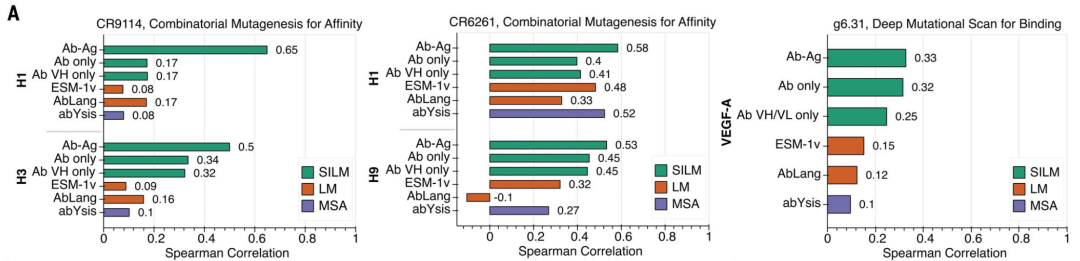
第二个有趣点是,ESM-IF1模型采用多条链作为输入,能够更准确的预测抗原抗体突变亲和力的变化。见下图,Ag-Ab作为模型的输入,抗原抗体突变亲和力变化预测上PR最高。
第三个点,ESM-IF1模型的log-likelihood与突变亲和力表现出强相关性(下图),能自然地想到用这类模型进行抗体突变体的筛选或设计,得到高Fitness的变体。
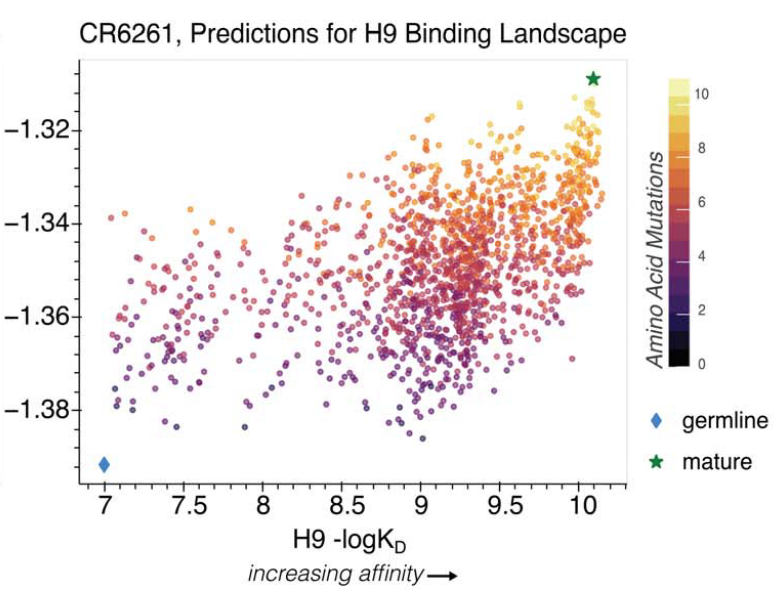
但编者也有些担忧之处:
除了上面指出的点之外,作者也是进行两轮迭代,选取单位点突变,如下图:
3.3 AbSci|抗体CDR的从头设计
AbSciBio公司的这篇CDR从头设计的论文,23年就上传Arxiv但好像一致未见刊,此论文针对HER2做了大量的实验,实验数据也开源到了Github,https://github.com/AbSciBio/unlocking-de-novo-antibody-design

只能够提供一种抗原结构,并让模型设计一种专门定制、能够与该抗原高亲和力和特异性结合的抗体。2023年初的AbSci的论文探讨了这个确切的问题,他们设计了完全能够结合到特定抗原的抗体,都是通过使用一个从未见过结合到该特定抗原的抗体的模型!这就是“de-novo”。有许多抗原有初始结合的抗体,所以一个不需要这一点的模型具有非常强大的能力。
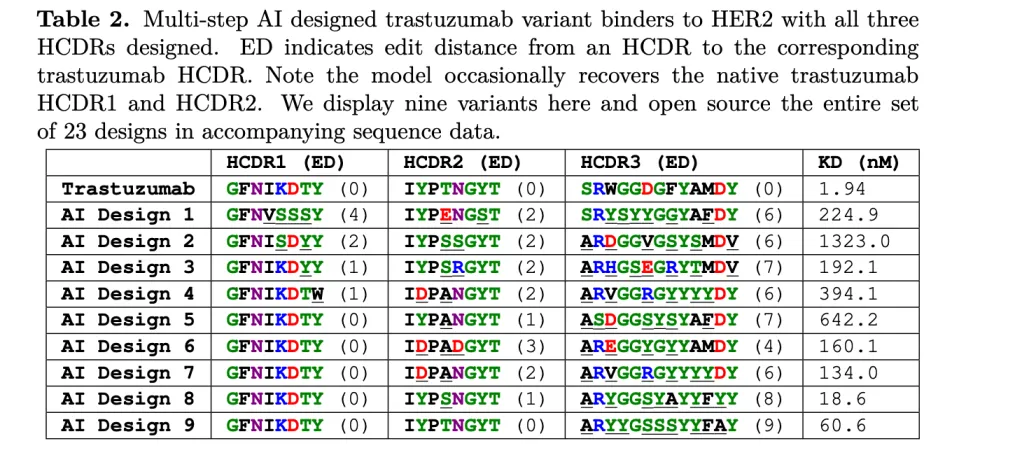
作者重新设计了一个著名抗体(曲妥珠单抗)结合到一个叫做HER2抗原蛋白的HCDR3区域。他们仅给模型输入HER2氨基酸序列,然后让模型填充曲妥珠单抗的HCDR3区域。那么这个“模型”到底是什么呢?遗憾的是,论文中根本没有讨论这个问题,也没讲算法技术的细节,可能是出于商业原因,因为一家抗体设计公司发布了这篇论文。
考虑到HCDR3区域之外的结合也被认为是重要的,并且曲妥珠单抗在HCDR3之外也有突变,可能想象AbSci这里的建模策略,可能在曲妥珠单抗非HCDR3区域已知的良好序列空间上实现“坐享其成”。此外,如果是从一个经过验证的抗体出发,这到底算不算是“de-novo”呢?还值得商榷!!!
作者的模型,生成了40万种可能的抗体,并以高通量实验方式测试它们与HER2抗原的结合情况。其中有4千个显示出结合性,其中有421个被选中进行后续高精度的实验验证,通过一些分子动力学进行预过滤,作者在SI补充材料中解释了这些。其中71个具有小于10nM的Kd值(记住,
但是当该模型被扩展到重新设计所有HCDR的3个区域时,情况就没那么乐观了……当然,尽管这些完全重新设计的抗体的Kd值并不糟糕,但与已已有的药物相比,它们与之仍有很大差距。实现全面抗体设计的梦想还远远未实现,即使是重新设计已建立抗体的HCDR区域(而不是HCDR,甚至非CDR区域)仍然具有相当大的挑战性!
所以划个重点,此论文结果说明,仅设计抗体HCDR3区域很多时候就能获取高亲和力;同时设计所有CDR区域,甚至整个抗体蛋白,只会增加难度。
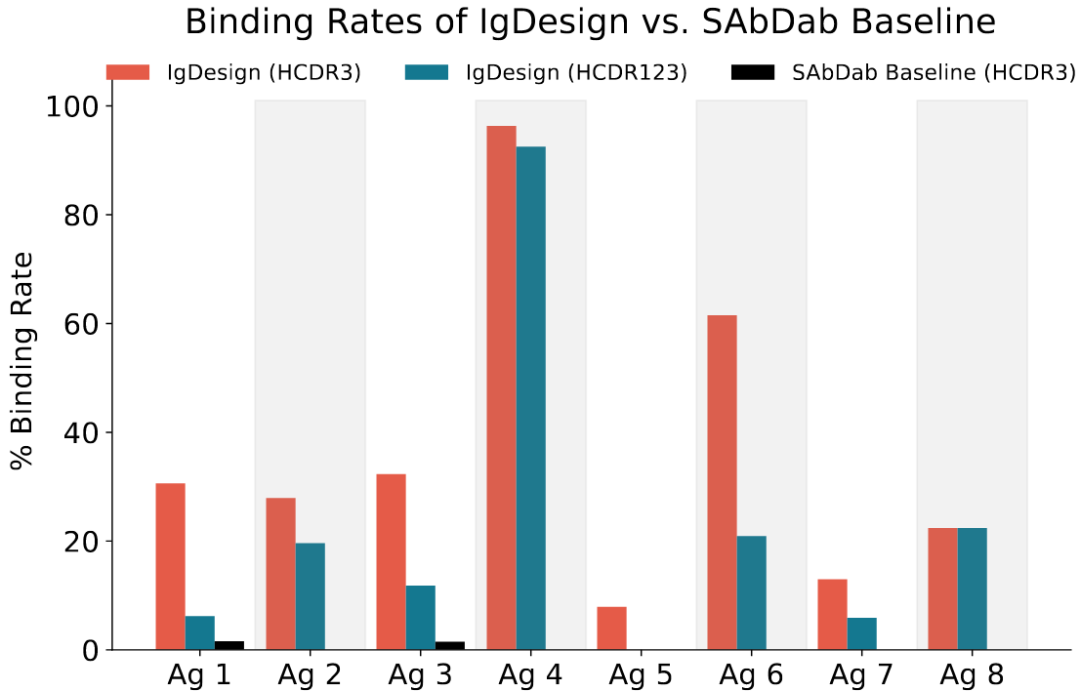
这篇论文的公司发布了一篇后续工作,使用一种称为‘IgDesign’的方法,该方法生成根据抗原而定的全新抗体。

这里不会过多讨论这篇论文,篇幅有限,实际上编者想偷懒。但我强烈建议读者自行阅读这篇论文!这篇论文的方法涉及重新设计HCDR1-3,以及对仅设计HCDR3进行了比较。实验结果表明,仅设计HCDR3就已经足够,见下图。还关注了各种不同的抗原,尽管对于其他部分抗体仍然使用经验证的抗体抗原!该文章取得了一些有趣的结果:对于8个抗原中的5个(抗原1、2、6、7和8),IgDesign生成了与参考抗体相等或更高亲和力的结合物。对于抗原5,却表现的很难。
3.4 David Baker|纳米抗体从头设计
David Baker这篇论文已有很多公众号写了优秀的报道,编者仅简单扼要介绍下

贝克实验室当然是解决蛋白设计难题的传奇人物,他们对此有一套方法。该方法最接近我们完全设计抗体的梦想,只需提供抗原作为输入。他们使用重新训练的RFDiffusion模型作为主要方法来实现这一目标。
但是,他们设计的并不完全是抗体,而是‘纳米抗体’或VHH。在这篇文章中,设计完整抗体并不特别重要,因为这些抗体的更小、更紧凑版本基本上具有相同的功效,甚至可能还有一些优势。我们在此之前已经讨论过这种抗体形式,但作为提醒,纳米抗体只由抗体的一个VHC组成;所以,只有3个CDR,它们都是重链,没有FWR区域。
那么,结果如何?最好由他们用自己的话来陈述:
已经针对RSV第三位点和流感血凝素对9000个设计的VHH进行了酵母表面显示筛选,然后在大肠杆菌中表达出顶级物种。表面等离子共振(SPR)表明,对RSV第三位点和流感血凝素具有最高亲和力的VHH分别与其各自的目标结合,亲和力分别为1.4μM和78nM。
对SARS-CoV-2受体结合结构域(RBD)进行了9000个VHH设计的测试,在可溶性表达后,SPR确认与目标之间的亲和力为5.5μM。重要的是,结合是到预期表位,通过与结构确定的从头开始的结合蛋白(AHB2,PDB:7UHB)竞争来确认。95个VHH设计针对TcdB进行了测试。具有最高亲和力的VHH具有262nM的亲和力。
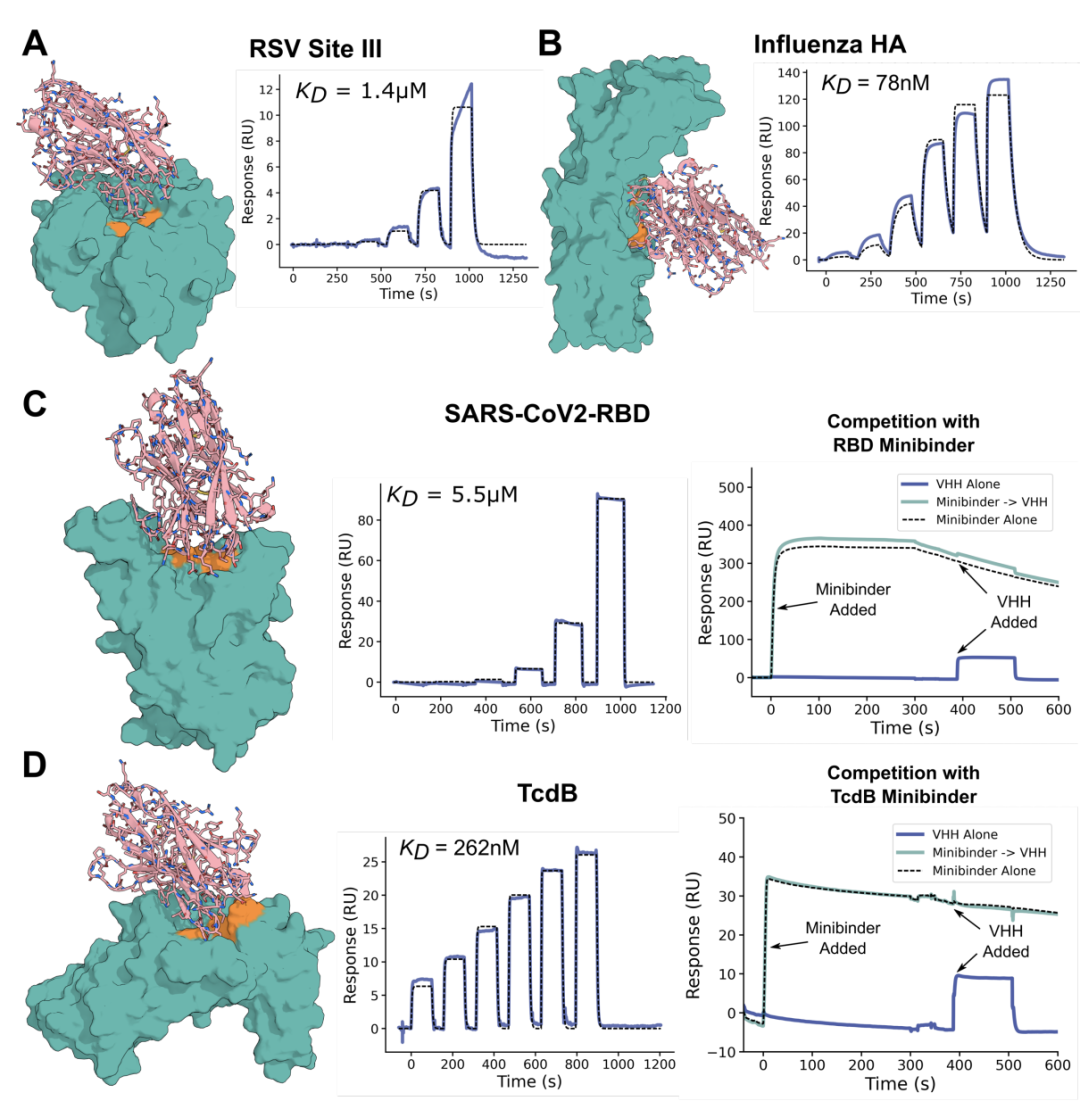
因此,虽然他们设计的纳米抗体,不能合理地被描述为对其抗原的‘强’结合体(Kd
整体来说,结合能力结果中等。但将抗体设计技术应用到抗体的最小化形式上取得了巨大进展(这比通常情况下的抗体设计更有潜力),但这些工程纳米抗体的结合能力,与临床级抗体中期望的亲和力相比还是不足的。然而,这是最接近真正的从头设计。鉴于RFDiffusion-AA已经发布,如果将本文的RFdiffusion替换,这可能会显著提高准确性和成功率。
关于本论文一些更优质的公众号报道,推荐读者拓展阅读:
3.5 其他抗体设计AI算法
除了上面介绍的几篇论文之外,还有很多算法或论文对抗体进行设计,但大多通常无湿实验的验证。在抗体骨架生成场景,IgDiff生成整个骨架,LoopGen只生成CDR骨架。在序列设计层面,有的算法设计整个CDR的6个区域,有的只针对设计CDRH1/2/3区域,有的只设计CDRH3。常用的逆向折叠方法,也微调到了抗原抗体场景,如ESM-IF1微调后的AntiFold,ProteinMPNN微调后的AbMPNN。在优化设计的抗体指标方面,大多数生成算法只管设计序列的恢复率、可设计性等,但也有些算法结合亲和力预测进行设计,仅有少数几个算法考虑抗体可开发性多种性质进行设计。还有针对抗体的语言模型,IgLM/AbLang等。文章之多很难一时讲清楚,编者简单归纳一下列在下表:
| 模型名称 | 文章链接 | 开源链接 | 场景 | 备注 |
|---|---|---|---|---|
| IgDesign | https://doi.org/10.1101/2023.12.08.570889 | N/A | 序列设计HCDR1/2/3 | 有实验验证 |
| Sculptor | https://doi.org/10.1101/2022.12.22.521698 | https://tinyurl.com/sculptormb | 抗原Binder设计 | 有实验验证,Po-Ssu Huang |
| AbLang2 | https://doi.org/10.1101/2024.02.02.578678 | https://github.com/oxpig/AbLang2 | 抗体语言模型 | |
| IgLM | https://doi.org/10.1016/j.cels.2023.10.001 | https://github.com/Graylab/IgLM | 抗体语言模型 | |
| BALM | https://doi.org/10.1016/j.patter.2024.100967 | https://github.com/brineylab/BALM-paper | 抗体语言模型 | |
| IgDiff | https://arxiv.org/abs/2405.07622 | https://zenodo.org/records/11184374 | 抗体骨架生成 | Charlotte M. Deane |
| PLAN | https://doi.org/10.1101/2023.09.04.556278 | 抗体人源化 | 许锦波 | |
| AbDiffuser | http://arxiv.org/abs/2308.05027 | |||
| AntBO | https://doi.org/10.1016/j.crmeth.2022.100374 | https://github.com/huawei-noah/HEBO/tree/master/AntBO | 逆向折叠 | 华为 |
| VcAb | https://doi.org/10.1101/2024.06.05.597540 | https://github.com/Fraternalilab/VCAb | 抗体工程网页工具 | |
| AbMPNN | https://arxiv.org/abs/2310.19513 | https://zenodo.org/record/8164693 | 逆向折叠 | Charlotte M. Deane |
| GeoAB | http://biorxiv.org/lookup/doi/10.1101/2024.05.15.594274 | https://github.com/BIRD-TAO/GeoAB | CDR骨架生成 | 李子青 |
| DIffAB | http://biorxiv.org/lookup/doi/10.1101/2022.07.10.499510 | https://github.com/luost26/diffab | CDR结构/序列共设计 | 彭建 |
| ATUE | http://arxiv.org/abs/2301.12112 | https://github.com/dqwang122/EATLM | Benchmark | |
| dyMEAN | http://arxiv.org/abs/2302.00203 | 全原子CDR设计 | 刘洋 | |
| DWJS | https://arxiv.org/abs/2306.12360 | https://github.com/prescient-design/walk-jump | ||
| AntiFold | https://arxiv.org/abs/2405.03370 | https://opig.stats.ox.ac.uk/data/downloads/AntiFold | 逆向折叠 | Charlotte M. Deane |
| AGN | http://arxiv.org/abs/2402.05982 | |||
| https://doi.org/10.1101/2023.11.22.568230 | https://github.com/amelvim/antibody-diffusion-properties | 可开发性优化设计 | ||
| http://arxiv.org/abs/2401.05341 | 序列设计CDRH3 | 强化学习 |
?上表肯定有遗漏或不全之处,欢迎读者在留言区推荐好的算法或模型。

