DRUGAI
今天为大家介绍的是来自英国剑桥大学Roser Vento-Tormo团队的一篇论文。细胞间通讯对组织发育、功能和再生至关重要。单细胞基因组学技术的革命性发展提供了前所未有的机会,使我们能够揭示细胞如何在其组织微环境中进行体内通讯,以及这些微环境的破坏如何导致疾病和发育异常。CellPhoneDB是一种生物信息学工具包,通过结合经过验证的配体-受体相互作用的精选库与整合这些相互作用和单细胞基因组学数据的方法,设计用于推断细胞间通讯。本文介绍了CellPhoneDB最新版本(v5)的使用方案,该版本提供了多项新功能。首先,数据库扩展了三分之一,增加了新的相互作用,包括约1,000个由非多肽配体介导的相互作用,如类固醇激素、神经递质和小G蛋白偶联受体(GPCR)结合配体。其次,作者概述了一种使用数据库的新方式,允许用户根据实验设计定制查询。第三,此更新纳入了优先考虑特定细胞间相互作用的新策略,利用来自其他模态的信息,如源自空间转录组学技术的组织微环境或源自单细胞转座酶可及染色质检测的转录因子活性。最后,作者描述了新的CellPhoneDBViz模块,用于交互式可视化和共享结果。总之,CellPhoneDB v5提高了细胞间通讯推断的精确性,为生理微环境中的组织生物学提供了新的见解。原论文需要约15分钟阅读,并且需要基本的python知识。此处受篇幅限制,仅展示部分章节内容。

从单细胞基因组图谱中模拟旁分泌、近邻分泌和内分泌细胞间通讯提供了一个独特的机会,使我们能够理解细胞在其生理微环境中的功能。为了从单细胞转录组数据中重建细胞通讯,作者开发了CellPhoneDB,它通过将单细胞基因表达数据与经过精选的受体-配体对数据库相结合,估计配体和受体的丰度。研究人员已经使用CellPhoneDB更好地理解人类疾病或感染的病因学,以及指导治疗策略的设计和改进体外模型。自首次推出以来,CellPhoneDB经历了几次更新,增加了新功能。在此,作者提供了CellPhoneDB最新版本(v5)包含的更新和扩展:(1)数据库,(2)推断和优先考虑细胞间相互作用的方法,以及(3)可视化结果的策略。
数据库更新部分
细胞间通讯推断的一个基本方面是存储先验知识的底层资源的准确性和范围。无论使用什么方法用转录组数据查询数据库,不准确或不完整的分子相互作用收集都会对预测产生负面影响。CellPhone v1从科学文献和同行评审库(Reactome和UniProt)手动精选,这种模式存储的相互作用覆盖范围仍然有限。在CellPhoneDB v5中,作者扩展了库,包括由非蛋白质配体介导的细胞间相互作用,如类固醇激素(例如,雌激素)、神经递质(例如,谷氨酸)和其他小非肽分子(例如,组胺)。大多数这些非蛋白质配体结合GPCR(是最大的膜受体家族,也是已批准药物的靶点)或核受体(是由能通过细胞膜的小疏水配体调节的最大转录因子超家族)。这些非蛋白质配体是一系列酶反应的产物,将原始底物转化为最终信使代谢物。为了考虑这种细胞间通讯模式并估计这些不直接由基因编码的代谢物的丰度,CellPhoneDB v5使用生物合成途径中最后一个已确认酶的表达水平和配体的转运蛋白(如已知)。酶和转运蛋白是从文献或同行评审的数据库(Reactome、HMRbase、GPCRdb)手动提取的。为了帮助跨库对齐,作者将这些非蛋白分子链接到其ChEBI标识符(专注于小化学化合物的分子实体字典)(如有)。相关的酶反应已尽可能链接到Rhea(专家精选的生化反应知识库)或REACTOME反应标识符。
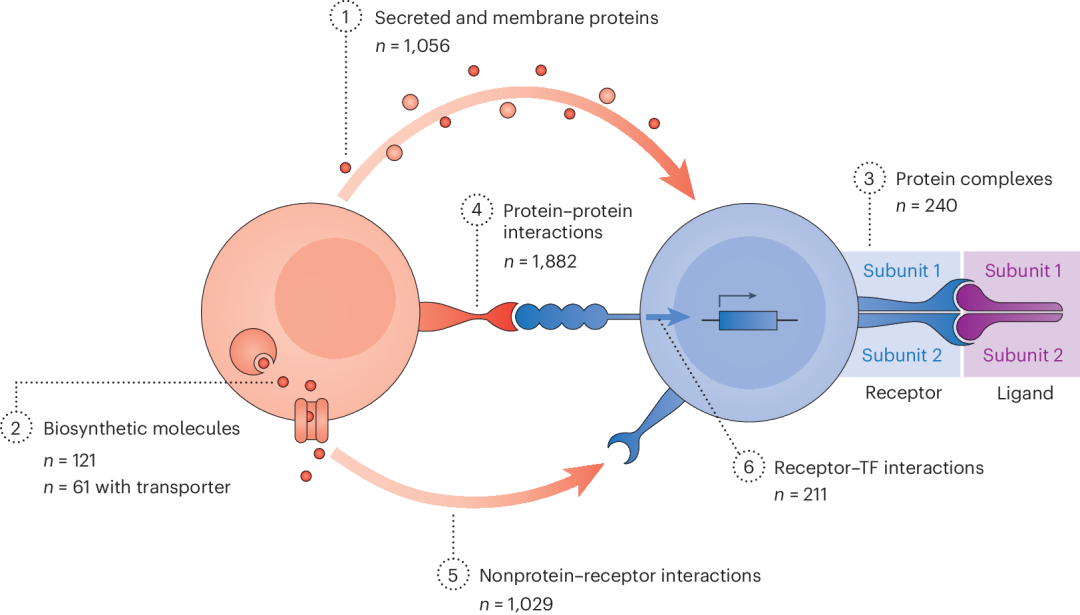
图 1
此外,CellPhoneDB v5通过(1)对文献进行额外的手动精选和(2)整合最近一项考虑了蛋白质亲和力和天然计量比的高通量表面受体筛选所识别的相互作用,加入了更多蛋白质介导的相互作用。CellPhoneDB v5库包括240个异源多聚复合物、1,056个同源多聚蛋白和120个非蛋白配体,其中61个依赖于多种蛋白质(即酶和转运蛋白;图1)。CellPhoneDB v5库有2,911个手动精选的相互作用,其中1,882个完全由蛋白质介导,1,029个由非蛋白配体介导。在所有精选的相互作用中,约50%涉及至少一个异源多聚复合物。与以前版本一样,用户可以修改内部数据库以包含新的相互作用,便于对数据集进行更有针对性的查询。
此外,在第5版中,作者通过从Uniprot数据库获取蛋白质位置(例如,用于识别分泌蛋白)和功能,并进行手动修订,组织了所有相互作用,以正确反映信号的方向性(从发送细胞到接收细胞)。方向性对于追踪信号级联和将受体连接到接收细胞中的下游转录因子测量至关重要(参见下文描述的CellSign模块)。在数据库的这个版本中,2,508个相互作用被分类为定向的配体-受体相互作用,而403个是双向相互作用,主要涉及细胞外基质蛋白(如胶原蛋白和整合素,n = 242)以及配体-配体、受体-受体或间隙连接相互作用(n = 161)。最后,为了帮助对这些相互作用进行功能解释,作者利用来自Reactome的信息和对文献的手动精选,将功能相关的相互作用分组为约150个信号通路。
细胞间相互作用的计算方法更新部分
一旦定义了细胞可以通过其进行通讯的潜在相互作用集合,下一步就是利用这些先验知识与注释的单细胞转录组数据相结合,基于配体和受体的表达来预测细胞间相互作用。没有适用于所有研究场景的从单细胞转录组数据集推断细胞间通讯的通用方法。相反,所选择的方法应根据实验设计(例如,考虑生物或技术协变量)和研究问题进行定制。CellPhoneDB提供了三种方法(方法1-3)将相互作用库与单细胞转录组数据集整合(图2)。
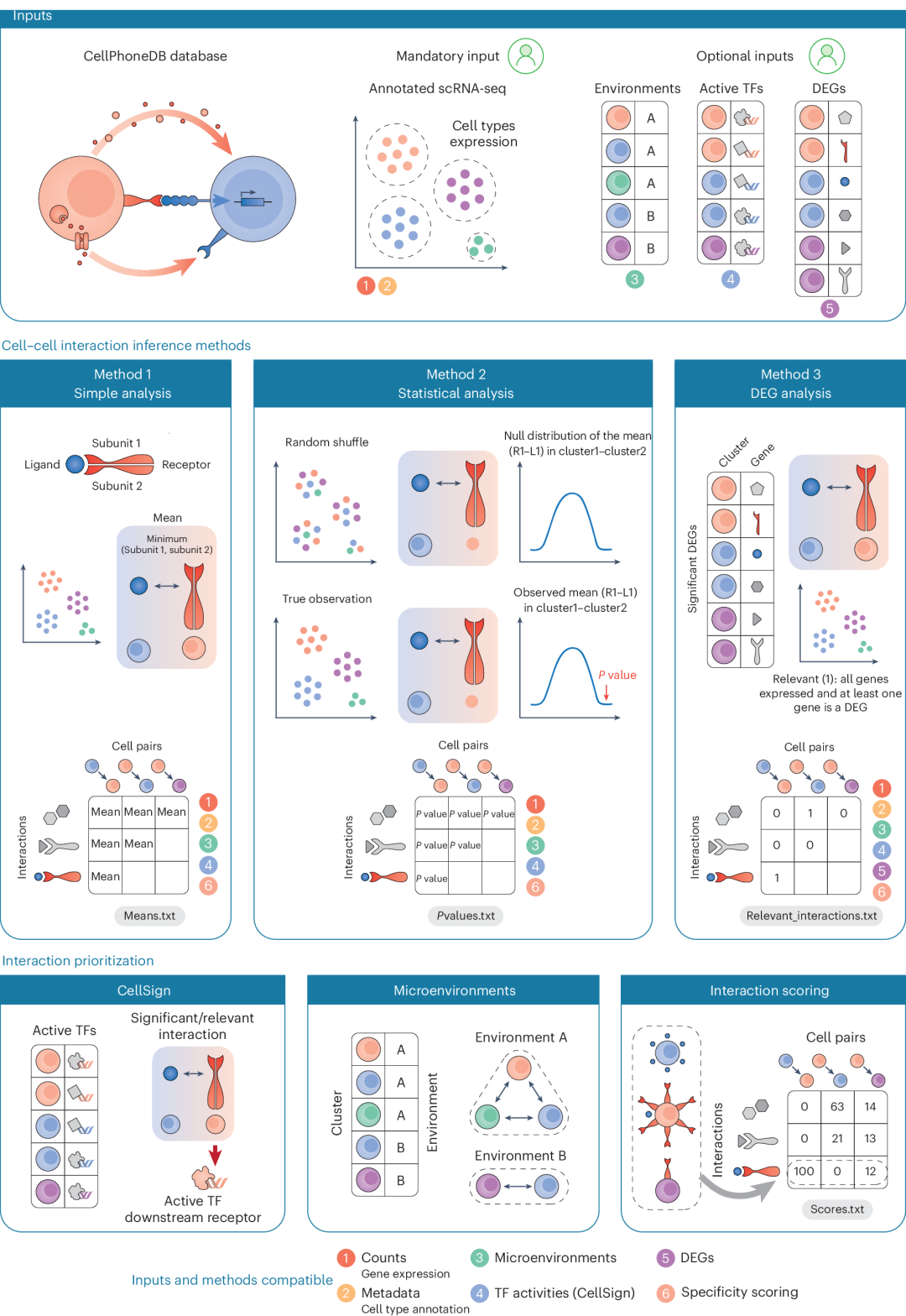
图 2
所有方法都依赖于由用户预先注释的单细胞转录组数据,其中细胞被分类为离散组,代表细胞类型或用户定义的任何其他功能单位。方法1-2是作为CellPhoneDB v1的一部分提出的。为了增加CellPhoneDB对更多定制实验设计的多功能性或限制预测,作者已经包含了一种新的计算方法,从差异表达分析中推断细胞间相互作用(方法3)。用户可以根据实验设计和需要解决的研究问题选择方法。
方法1(又称简单分析)为每个细胞类型对中的每个相互作用返回相互作用伙伴的平均表达值。对于多亚基异源多聚复合物,该方法将表达最低的亚基值分配给整个复合物。该方法排除了至少一个基因伙伴不表达或在细胞类型中表达的细胞比例低于阈值(由用户定义)的相互作用。因此,方法1返回所有伙伴表达高于阈值的相互作用,不包含统计数据;这些结果可以用作初步探索分析,提取所有潜在的相互作用,或用于生成针对数据集定制的细胞间相互作用数据库,用于下游分析。
方法2(又称统计分析),也在CellPhoneDB v1中实施,通过评估平均相互作用表达偶然发生的概率来扩展方法1。方法2通过随机置换整个数据集中所有细胞的细胞类型标签,为每个相互作用估计平均表达的空分布,这意味着输入数据集中的所有细胞类型都用于估计这个背景分布。细胞类型对之间每个相互作用的P值被计算为空分布中等于或高于观察到的平均值的比例。因此,方法2旨在识别在整个数据集中被特定细胞类型对上调的相互作用,使其适用于用户寻求执行"一对多"比较的一般探索性分析。换句话说,它试图回答问题"与数据集中所有其他细胞类型对相比,特定相互作用在给定细胞类型对中的表达是否显著更高?"。为了减少方法2中置换大型数据集所需的计算时间,CellPhoneDB可以使用原始数据的几何草图对原始数据进行子采样,减少原始数据集的大小同时保留稀有细胞状态。
方法3(又称差异表达分析,aka differential expression analysis)提供了与"一对多"方法2不同的方法。它允许用户设计更有针对性的查询,以识别特定细胞类型或条件的相关相互作用。这对于识别以下情况特别有用:(1)特定于细胞类型或谱系内的个别细胞状态的相互作用(图3a),(2)特定于特定条件的细胞的相互作用(即通过比较疾病与对照或扰动与稳态),(3)在分化过程中产生的相互作用,或(4)沿着空间轴差异表达的相互作用。方法3需要用户提供感兴趣细胞类型中差异表达基因的列表,这由用户通过比较离散细胞类型(应用1和2)或通过沿轨迹建模表达动态(应用3和4)来估计。因此,差异表达分析由用户使用其首选方法预先计算,以适应其实验设计(例如,建模混杂协变量和技术批次)和研究问题(例如,通过定义零假设或将表达的背景分布限制在特定细胞谱系或条件,而不是数据集中的所有细胞)。使用这种方法,CellPhoneDB检索那些至少一个伙伴在细胞类型中差异表达(根据用户提供的差异表达列表),而另一个伙伴由数据集中的任何其他细胞类型表达(高于用户指定的阈值)或者,如果已定义,在微环境中表达的相互作用。
计算方法优先考虑细胞间相互作用
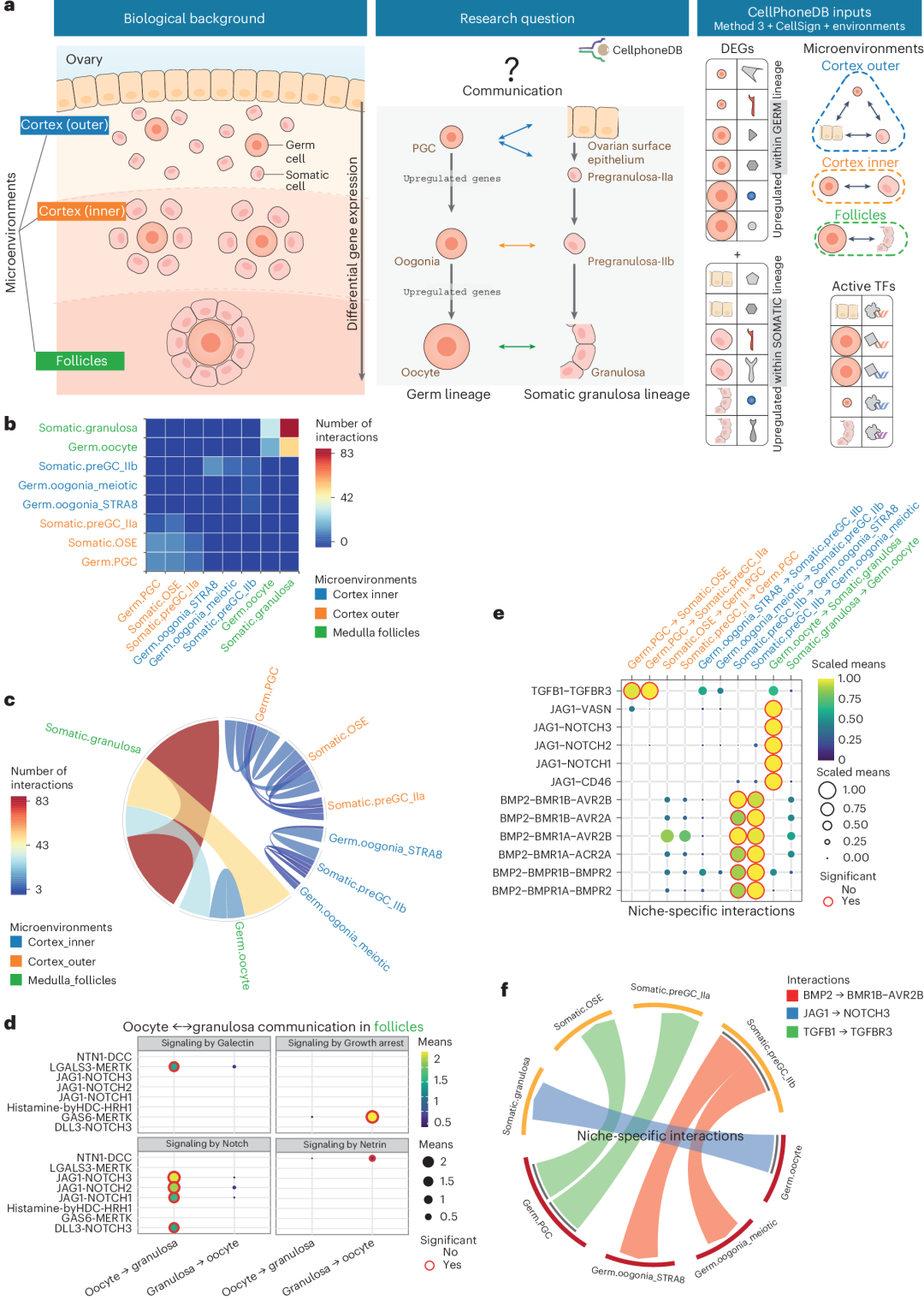
图 3
由于CellPhoneDB方法预测的细胞间相互作用数量通常很大,CellPhoneDB v5包括了新的可选方法,以优先考虑由多层证据支持的细胞间相互作用,例如包括空间转录组学或单细胞染色质可及性测定等其他组学模态。CellPhoneDB v5可以使用空间转录组学将分析限制在那些在组织微环境中在时间和空间上共存的细胞类型对,从而将预测的相互作用减少到发生在相邻细胞之间的相互作用(图3a)。空间微环境可以从多种数据模态中定义,这些模态可以识别共定位细胞,包括来自空间转录组学(例如,通过使用cell2location、stereoscope或类似工具对10x Visium空间转录组学进行解卷积)或基于成像的技术(例如,原位杂交或细胞类型标记物的原位测序)。或者,用户可以依靠组织的先验生物学知识(例如,血管中周细胞和内皮细胞的共定位)。从空间转录组学数据集系统推断空间细胞生态位的其他方法包括SpatialDE2、Giotto、SPARK或更新的工具如NicheCompass或COVET。微环境组成也可以从发育过程中细胞类型的时间动态或对比条件(如扰动与对照或疾病与对照)中定义,这些信息可用于排除在时间上不共存的细胞类型。
此外,CellPhoneDB v5包括CellSign模块,通过利用下游信号依赖性转录因子的活性来优先考虑高可信度的相互作用。这个优先模块基于这样的假设:某些转录因子直接被细胞表面的配体结合受体激活(或在核受体超家族的情况下在细胞质中)。该方法依赖于一个经过精选的受体链接到其假定下游转录因子的数据库,这些转录因子通过手动文献回顾检索。这个数据库仅关注对上游受体具有高特异性的转录因子,包括作为转录因子的核受体。目前,CellSign数据库包含总共212个高度特异的受体到转录因子关系,作为CellPhoneDB数据库的一部分存储。CellPhoneDB使用CellSign优先考虑那些得到下游直接调控转录因子激活支持的配体-受体相互作用,从而为细胞间相互作用的可能性增加额外的证据层。通过仅包含直接的受体到转录因子关系,该方法减少了噪音和假阳性,帮助研究人员优先考虑最有意义的相互作用进行进一步验证。该模块需要用户提供每种细胞类型中活跃的转录因子列表,理想情况下从高通量表观遗传学分析数据(如单细胞染色质可及性测定,使用ChromVar或SCENIC)或利用转录因子靶基因表达的单细胞转录组数据集(使用DoRothEA或DecoupleR中的CollecTRI)以数据驱动的方式估计。用户可以更新受体-转录因子数据库,以包括原始数据库中没有的额外关系。
最后,CellPhoneDB v5包括一种评分方法,该方法基于相互作用伙伴的细胞类型特异性基因表达来优先考虑细胞间相互作用,这可用于减少生成的相互作用数量。这些策略之前由其他团队提出,当没有其他信息(如空间共定位或转录因子活动)来优先考虑相互作用时特别有用。评分方法的目标是作为一种排名方法,与其他方法结合使用,便于选择一组最特异的相互作用。虽然方法1和方法2分别提取所有伙伴表达或过表达的相互作用,但评分方法旨在根据表达对细胞类型对的特异性来对相互作用进行排名。
可视化策略的更新
CellPhoneDBViz是一个新的基于网络的界面,通过用户的最少配置,提供任何CellPhoneDB分析结果的交互式可视化和搜索。它可以用于与没有计算机科学背景的其他研究人员共享CellPhoneDB结果,并允许他们执行交互式搜索,显示整体视图以发现全局相互作用模式,或缩放到特定的细胞类型对或微环境以执行更具体的查询。具体来说,它以桑基图、热图、弦图和点图的形式为CellPhoneDB输入和输出提供各种不同的可视化(图3b,c)。这些图可以通过基因、相互作用对或细胞类型以及通过微环境进行筛选,以执行定制查询。最后,图可以下载为pdf格式以供重用。CellPhoneDBViz最初是为了可视化人类子宫内膜细胞图谱中的细胞交叉对话而实施的。
除了使用CellPhoneDBViz,CellPhoneDB结果的可视化还可以使用ktplots(R)和ktplotspy(python)包(图3d-f)。每个包都包含接受CellPhoneDB输出的函数,包括相应于相互作用均值、P值(或相关相互作用)、解卷积值的数据框架,以及来自相互作用得分和CellSign模块的新输出。两个包还需要用于CellPhoneDB的相应单细胞对象(例如,Seurat、SingleCellExperiment和AnnData)。主要函数'plot_cpdb'将结果绘制为点图,其中每个细胞类型对的每个相互作用的大小和颜色梯度分别按照显著性和相互作用伙伴的平均值进行缩放。如果提供了来自相互作用得分和CellSign模块的结果,'plot_cpdb'使用不同级别的不透明度来区分按等级的相互作用或突出显示由CellSign支持的相互作用。'plot_cpdb'还允许用户按细胞类型对或基因过滤相互作用(图3d,e),这些可以进一步按信号通路或任何其他分类分组(图3d)。其他相关函数使用从'plot_cpdb'生成的长格式数据框生成弦图和各种其他可视化(图3f)。所有图都是可定制的,因为它们主要使用ggplot2(R)和/或plotnine和matplotlib语法(python)创建。Ktplots还实现了原始CellPhoneDB可视化,展示每个细胞类型对的总相互作用数的热图。这种可视化可以使用pheatmap(R)或seaborn(python)实现,并提供对称和非对称输出,以反映细胞间相互作用从发送者到接收者的定向性质。
编译|黄海涛
审稿|王梓旭
参考资料
Troulé, K., Petryszak, R., Cakir, B., Cranley, J., Harasty, A., Prete, M., ... & Vento-Tormo, R. (2025). CellPhoneDB v5: inferring cell–cell communication from single-cell multiomics data. Nature Protocols, 1-29.
内容中包含的图片若涉及版权问题,请及时与我们联系删除

