DRUGAI
今天为大家介绍的是来自Jennifer A. Doudna团队的一篇论文。病毒的快速进化产生了对感染性和复制至关重要的蛋白质,但由于序列的极端分化,这些蛋白质的功能尚不明确。本文中,作者利用一个包含4,463种真核病毒物种的67,715个新预测的蛋白质结构数据库发现,62%的病毒蛋白在结构上是独特的,并且在AlphaFold数据库中没有同源蛋白。剩下的38%的病毒蛋白中,许多具有非病毒的结构类比,揭示了人类病原体与其真核宿主之间令人惊讶的相似性。结构比较为多达25%的未注释病毒蛋白提供了推测功能,其中包括在逃避先天免疫中的作用。特别是,RNA连接酶T样磷酸二酯酶被发现与噬菌体编码的蛋白质相似,这些蛋白质能够水解宿主免疫激活的环状二核苷酸3′,3′-和2′,3′-环状GMP-AMP (cGAMP)。实验分析表明,由禽痘病毒编码的RNA连接酶T同源物同样能够水解cGAMP,显示RNA连接酶T介导的cGAMP靶向是存在于噬菌体和真核病毒中的一种进化保守的免疫逃逸机制。总之,这里展示的病毒蛋白结构数据库和分析为识别跨病毒组普遍存在的病毒-宿主相互作用机制提供了新的机会。

病毒蛋白在感染过程中发挥关键作用。有些蛋白或它们的组成结构域在病毒家族内外广泛保守,包括不同巴尔的摩分类的病毒之间,以及感染不同生命界的病毒中。这些保守的蛋白包括“病毒标志基因”,如病毒衣壳蛋白的果冻卷折叠结构以及与RNA和DNA依赖的RNA聚合酶相关的折叠。然而,理解病毒感染机制和进化的一个主要挑战是大量病毒蛋白的功能未知。病毒蛋白与其他病毒或非病毒蛋白之间的序列相似性有时可以推测蛋白功能,但病毒快速进化和基因的新生使得许多蛋白没有注释的序列同源,这就迫切需要通过其他方法来识别蛋白质类比。
即使在同一个病毒家族内,病毒蛋白也表现出高度的分化,当氨基酸序列相似性低于30%时,基于序列的相似性搜索的实用性受限。相比之下,病毒之间以及病毒与细胞之间的水平基因转移会产生结构关系,如果能够检测到这些关系,就可以揭示蛋白功能。然而,病毒蛋白在蛋白质数据库(PDB)中实验确定的结构中代表性有限,并且在AlphaFold数据库中缺乏预测的蛋白结构。
为了弥补这一空白并系统性预测病毒蛋白的功能,作者建立了一个包含67,715个由4,463种真核病毒编码的蛋白质的预测结构数据库。作者通过序列和结构将这些蛋白分为5,770个多成员簇和12,422个单一成员簇。结构相似性搜索大大扩展了蛋白簇的分类多样性,通过将未注释的病毒蛋白与已注释的类比蛋白连接,揭示了潜在的蛋白功能。病毒与非病毒蛋白的结构比较识别出由人类病原体编码的蛋白质的潜在功能。特别是,RNA连接酶T(LigT)样磷酸二酯酶(PDEs)在分析中被发现为一种广泛存在的酶类,在细菌和真核病毒组中高度保守。结合病毒蛋白结构数据库中的保守性以及细胞实验中验证的酶活性,揭示了这些蛋白在病毒抗免疫途径中古老而重要的作用。
真核病毒的蛋白质组
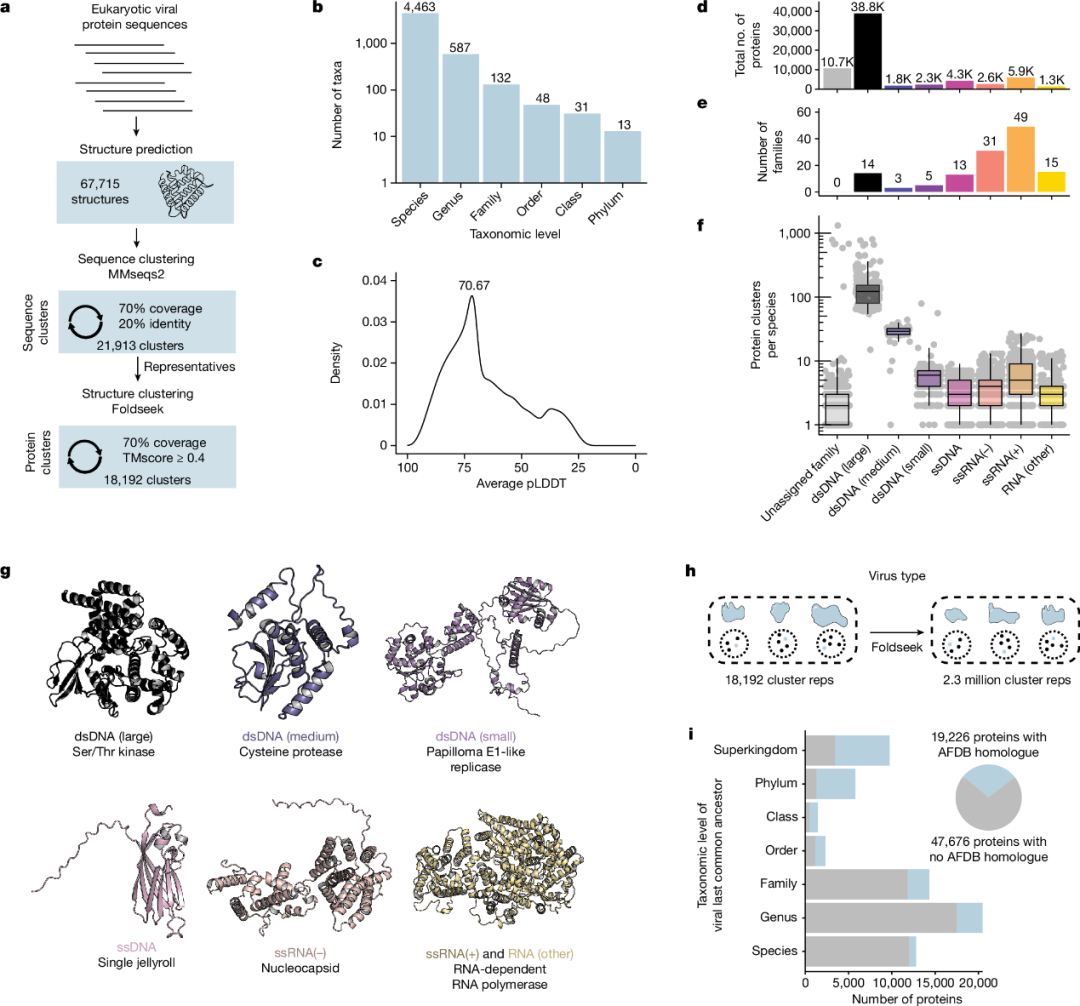
图 1
为分析真核病毒中存在的蛋白质结构多样性,作者使用ColabFold根据病毒多序列比对(MSAs)预测了RefSeq中67,715个真核病毒的蛋白质结构(方法部分)。随后,作者采用两步法对这些蛋白进行聚类,结合了基于序列和结构的聚类方法(图1a)。首先,使用MMseqs2将蛋白序列聚类至70%覆盖率和20%同一性,产生了21,913个序列簇。接着,利用Foldseek的对齐速度,对每个序列簇的代表序列进行结构对齐,并筛选对齐覆盖率至少为70%、TM分数不低于0.4且E值低于0.001的对齐结果。最终,结构对齐结果的中位TM score为0.52,反映了较强的结构相似性。70%的对齐覆盖率门槛丰富了簇中在大多数蛋白序列中相似的成员。最终结果为18,192个蛋白簇,其中12,422个为单一成员簇(扩展数据图1b)。这一数据集包含了大量多样化的病毒,包括来自132个不同病毒家族的4,463种病毒(图1b)。聚类具有结构一致性,因为对具有至少100个成员的簇,使用DALI对簇代表与每个成员进行对齐,得到的簇平均DALI z分数中位数为13.1。DALI z分数高于8表示两个蛋白质可能是同源的。单成员簇中的蛋白相比于非单成员簇,具有明显较低的预测局部距离差异测试(pLDDT)值,这表明结构预测质量对检测结构相似性能力有重要影响。作者测试了针对更大参考数据库的MSA生成是否对预测质量有影响。结果发现,尽管单成员簇的平均MSA深度较低且与较低的pLDDT相关,但这一替代MSA生成对结构预测质量的影响微乎其微。
作者研究了该数据库对病毒多样性的代表性以及是否能够重现核心的病毒标志基因。作者根据巴尔的摩分类并稍作修改,将病毒家族按基因组类型进行分组:DNA病毒根据其平均基因组长度分为大型、中型和小型,RNA病毒中没有单链正义或负义基因组的归入RNA(其他)类别。大型双链DNA(dsDNA)病毒每个物种的蛋白簇最多,尽管它们在数据集中只占132个病毒家族中的14个,却包含了大多数的病毒蛋白(图1d, f)。如预期,蛋白簇的数量与基因组大小密切相关。由于基因组较大,dsDNA病毒有能力编码更多辅助基因而不影响基因组稳定性。RNA病毒在数据集中占据了大量家族,但在总蛋白质中占比较小(图1e, f)。具有相似基因组类型的病毒家族之间常见结构相似性,大型dsDNA病毒共享许多蛋白质折叠。
如预期,数据集中整体和各基因组类型内(图1g)的主要蛋白簇大多参与病毒生命周期的基本方面。这些包括单果冻卷折叠,组成病毒衣壳,并存在于多种基因组类型的病毒中。双果冻卷折叠也组成病毒衣壳,但仅限于dsDNA病毒。RNA病毒家族通常编码核衣壳,负责包装病毒RNA,以及负责基因组复制的RNA依赖的RNA聚合酶。尽管RNA依赖的RNA聚合酶在RNA病毒中普遍保守,但由于蛋白质长度的变化,它被分为多个蛋白簇。相比之下,小型dsDNA病毒如乳头瘤病毒和多瘤病毒编码具有保守起始结合和解旋酶结构域的病毒复制酶。总的来说,作者发现他们的结构数据库成功地重现了在不同病毒亚型中保守的病毒蛋白。
接下来,作者研究了病毒蛋白簇的分类分布。作者将病毒蛋白簇的代表与整个AlphaFold数据库中的230万个簇代表进行了结构比对(图1h)。对于每个病毒蛋白簇,作者确定了编码簇成员的病毒的最近共同祖先。结果发现,29%的蛋白簇存在于多个病毒家族中,其中大多数在AlphaFold数据库中有记录,表明它们具有古老的进化历史(图1i)。此外,作者发现62%的病毒蛋白(或来自非单一成员簇的55%蛋白)仅限于单一病毒家族,并且在AlphaFold数据库中没有对应的类比蛋白(图1i)。这表明病毒的进化产生了大量在现有结构数据库中缺失的新蛋白。
病毒蛋白之间的相似性
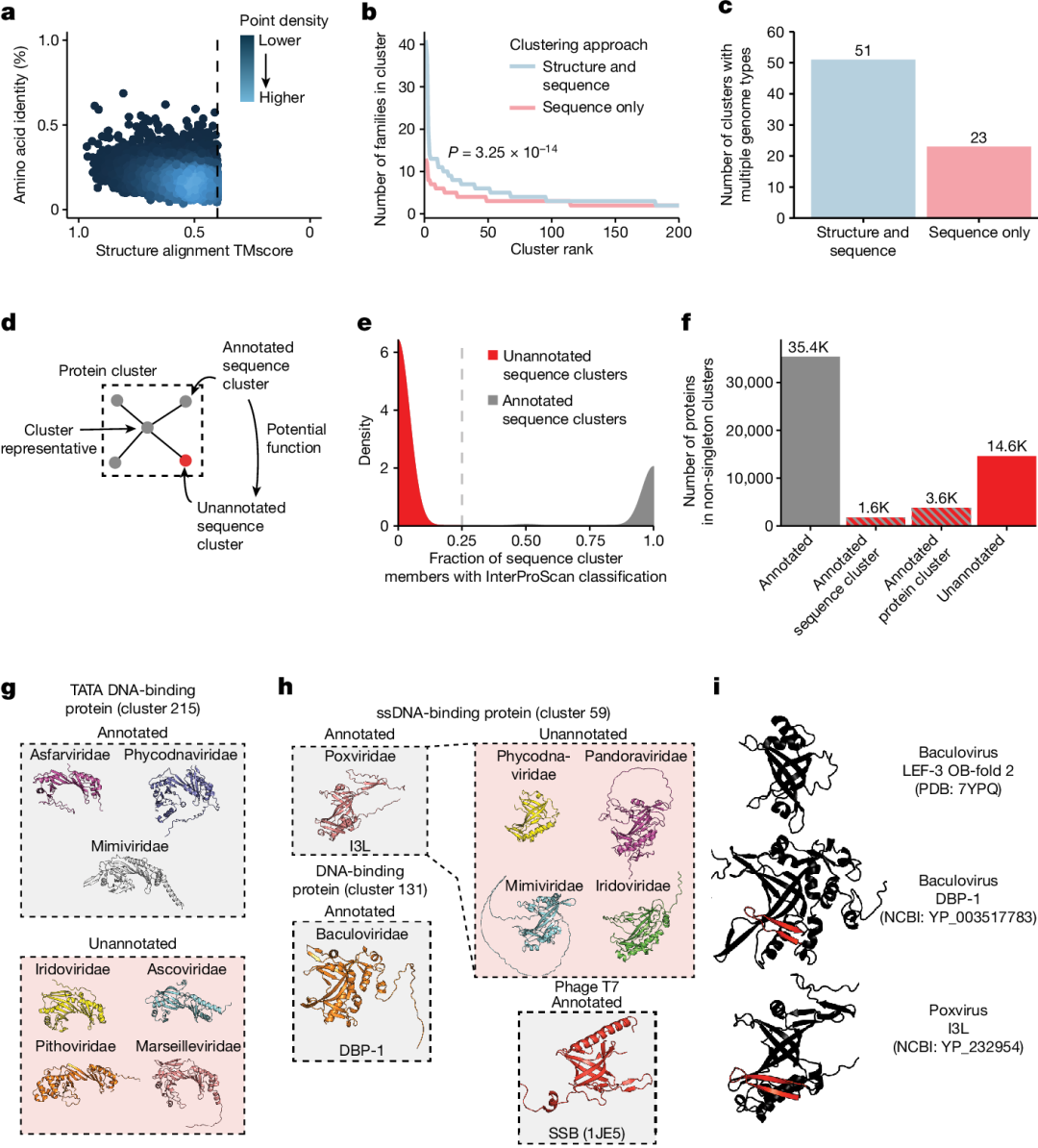
图 2
作者研究了结构比对在识别仅通过蛋白质序列无法发现的关系的能力。发现许多序列簇的代表尽管序列相似性较低,但在结构上却很相似(图2a)。将结构信息加入蛋白聚类的过程中,可以生成更多分类多样的蛋白簇,每个簇包含的病毒家族数量显著增加(图2b)。这对于发现来自不同病毒的蛋白质之间的相似性尤为重要,显著增加了包含不同基因组类型病毒蛋白的蛋白簇数量(图2c)。
作者还探索了结构比对是否能够将注释较少的序列簇与注释较多的序列簇关联起来(图2d)。作者使用基于序列的分类工具InterProScan为所有蛋白分配了Pfam、保守域数据库(CDD)和TIGRFAM分类。序列簇几乎完全由InterProScan注释的成员或完全未注释的成员组成,导致序列簇呈现出双峰分布(图2e)。在拥有多个成员的蛋白簇中,超过25%的未注释蛋白位于一个注释的序列簇中或位于包含注释序列簇的蛋白簇中(图2f)。
许多蛋白簇包含了注释和未注释序列簇的混合体。作者发现,这些序列簇之间的关联有助于推测病毒组中那些特性不明确的蛋白质的潜在功能。例如,尽管单果冻卷折叠(single jellyroll fold)是最丰富的蛋白簇,但其中许多成员并没有被正确注释。其他许多蛋白簇也包括了注释和未注释的序列簇,包括编码酶的簇,如核苷酸-磷酸激酶、NUDIX水解酶、DNA连接酶和核酸酶。
接下来,作者研究了具有生物技术应用的DNA结合蛋白,包括在诊断和基因组编辑中的应用。首先,作者研究了TATA结合蛋白(TBPs),这些蛋白与真核生物启动子中的TATA盒基序结合。许多DNA病毒会靶向人类TBP以促进病毒基因表达或调节宿主基因表达。目前,已知有三个大型dsDNA病毒家族编码了病毒TBPs。作者在另外四个大型dsDNA病毒家族中发现了这些蛋白的证据(图2g),显著扩大了病毒编码TBPs的多样性。接着,作者研究了由痘病毒编码的单链DNA(ssDNA)结合蛋白I3L家族(图2h)。I3L能够强效且特异性地结合ssDNA,据信它是一种与病毒DNA复制或修复相关的DNA结合蛋白。目前尚无I3L的实验结构,并且它与其他蛋白折叠和家族的联系尚不明确。作者发现I3L包含一种寡核苷酸结合折叠(OB折叠),与杆状病毒DNA结合蛋白DBP和噬菌体T7单链结合蛋白(SSB)相似,这与这些蛋白共享的ssDNA结合行为一致。作者确认在另外四个dsDNA病毒家族中存在类似的OB折叠蛋白,表明痘病毒I3L代表了一类广泛存在的ssDNA结合蛋白。这些真核dsDNA病毒的OB折叠在N端含有一个独特的β折叠,这在其他由杆状病毒编码的OB折叠蛋白LEF-3中是缺失的(图2i)。总的来说,这些结果表明,基于序列和预测结构的大规模聚类可以为功能未明的病毒蛋白提供功能推断。
与非病毒蛋白的相似性
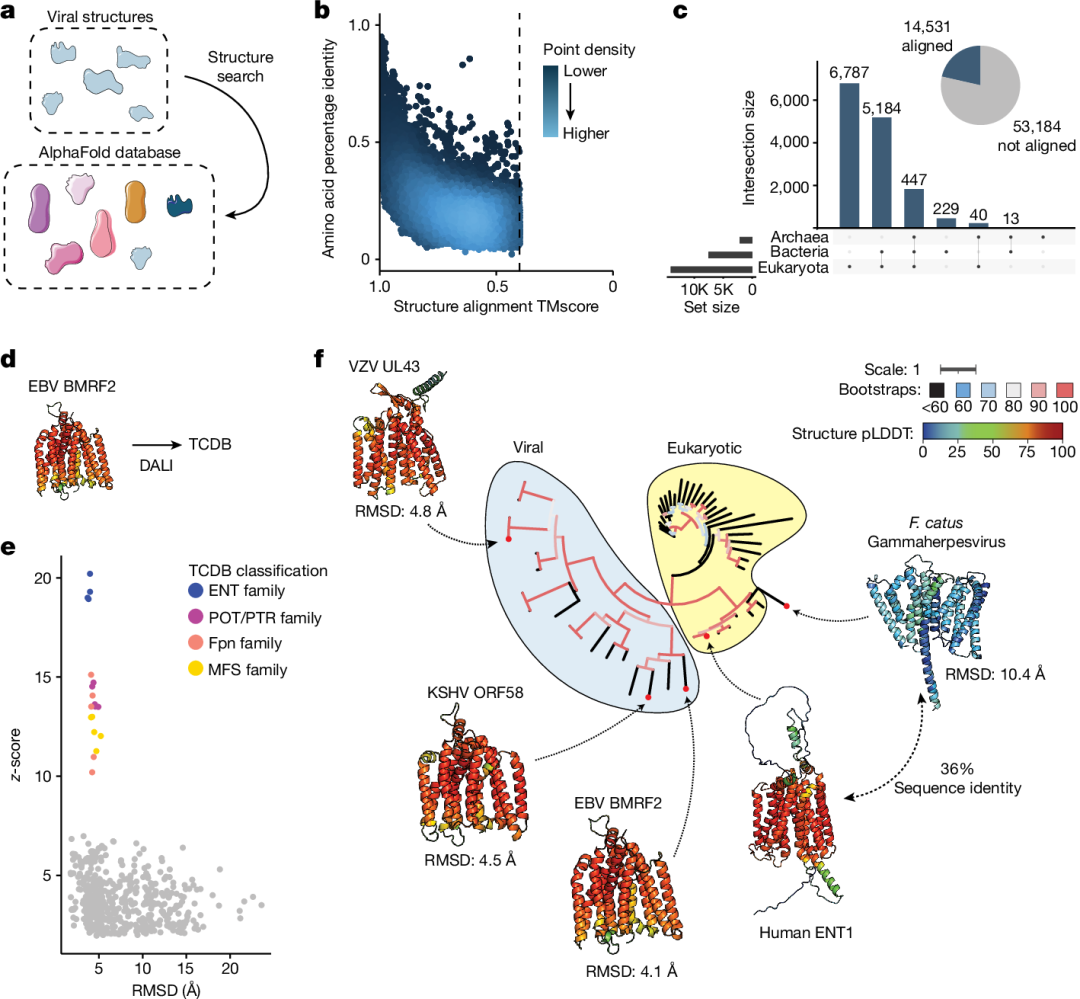
图 3
与核苷酸或蛋白质序列不同,结构特征常在较长的进化时间尺度上保持保守。因此,作者研究了预测的病毒和非病毒蛋白结构之间的比对是否能为由人类病原体编码的注释较少的蛋白质功能提供见解。为此,作者使用Foldseek将病毒蛋白结构数据库与AlphaFold数据库的初始版本进行比对,该数据库包含来自真核生物、细菌和古细菌的21个生物的30多万种蛋白质(图3a)。结果显示,病毒与非病毒蛋白之间存在广泛的结构相似性,即使氨基酸同一性较低,也表现出高度的结构相似性(图3b)。
最终,共有14531个预测的病毒蛋白与AlphaFold数据库中的成员进行了比对,大多数比对对象是由真核生物编码的蛋白(图3c)。这些比对包括未注释但由人类病原体编码的蛋白质。为了减少假阴性的发生率,作者使用了DALI进行了一系列比对,尽管DALI比Foldseek慢,但其敏感度更高。首先,作者发现一组由痘病毒编码的蛋白在结构上与哺乳动物气体诱导蛋白的自抑制结构域相似。同样,几种痘病毒蛋白在结构上与人类半乳糖转移酶COLGALT1相似,后者被认为在病毒入侵时能够与表面糖胺聚糖结合。此外,作者观察到痘病毒C4样蛋白与真核双加氧酶在结构上的相似性,这与先前研究发现痘病毒频繁利用失活的宿主酶一致。天花病毒C4因对抗多条先天免疫通路而引人注目。C4直接与模式识别受体DNA依赖蛋白激酶(DNA-PK)结合,阻止DNA结合并通过该通路阻断免疫信号传导。此外,C4在IκB激酶(IKK)复合物或其下游抑制NF-κB信号传导,但其抑制机制尚不明确。进一步的研究需要确定其类似双加氧酶的折叠是否参与了其对先天免疫的对抗。
接着,作者发现人类疱疹病毒UL43样蛋白,包括来自EB病毒(Epstein–Barr herpesvirus)的BMRF2蛋白和水痘带状疱疹病毒(VZV),在结构上与人类平衡核苷转运蛋白ENT4相似。作者使用DALI对EBV BMRF2与转运蛋白分类数据库(TCDB)中的蛋白进行了结构比对(图3d)。结果显示,BMRF2与人类平衡核苷转运蛋白(ENT)家族的转运蛋白具有很强的结构相似性,并且与相关的转运蛋白家族也有较弱的相似性(图3e)。作者还构建了疱疹病毒UL43样蛋白与相关真核蛋白的系统发育树,显示这些蛋白广泛分布于疱疹病毒中(图3f)。值得注意的是,作者鉴定出一种由猫伽玛疱疹病毒编码的变体,其与人类ENT1有36%的序列相似性,这支持了这些疱疹病毒蛋白与ENT蛋白之间的结构关联。EB病毒在感染过程中显著重塑宿主细胞代谢,这一发现表明,除了BMRF2在病毒附着中的作用外,还可能具有潜在的代谢功能。此外,抗病毒核苷类似物如伐昔洛韦的转运是由核苷转运蛋白介导的,这引发了关于该蛋白在VZV感染期间与伐昔洛韦相互作用的疑问。这些蛋白属于与疱疹病毒后期蛋白UL43家族相似的蛋白簇,其中一些尚未注释。进一步的实验表征需要确认这些假定的转运蛋白所转运的底物。总体而言,这些发现展示了病毒与非病毒蛋白之间结构相似性的普遍性,并表明这种相似性可以用来预测功能尚不明确的病毒蛋白的潜在功能。
共享结构域的识别
作者构建了蛋白质簇,并设定了严格的70%覆盖率要求,这样可以通过结构比较识别出单个结构域。作者认为,出现在多个蛋白质簇中的蛋白质结构域可能具有特殊的生物学重要性。作者使用DALI对所有包含多个成员的蛋白质簇代表进行了全对全比对,结果显示许多比对的z分数大于8,显示出显著的蛋白质相似性。最终,蛋白质簇形成了一个共享结构域的网络。作者发现,不同的结构域常常在不同的蛋白质簇中以各种组合形式共享,这在与细胞骨架相互作用的结构域和参与代谢的结构域中尤为明显,这些结构域存在于真核病毒和噬菌体中。
发现cGAMP磷酸二酯酶
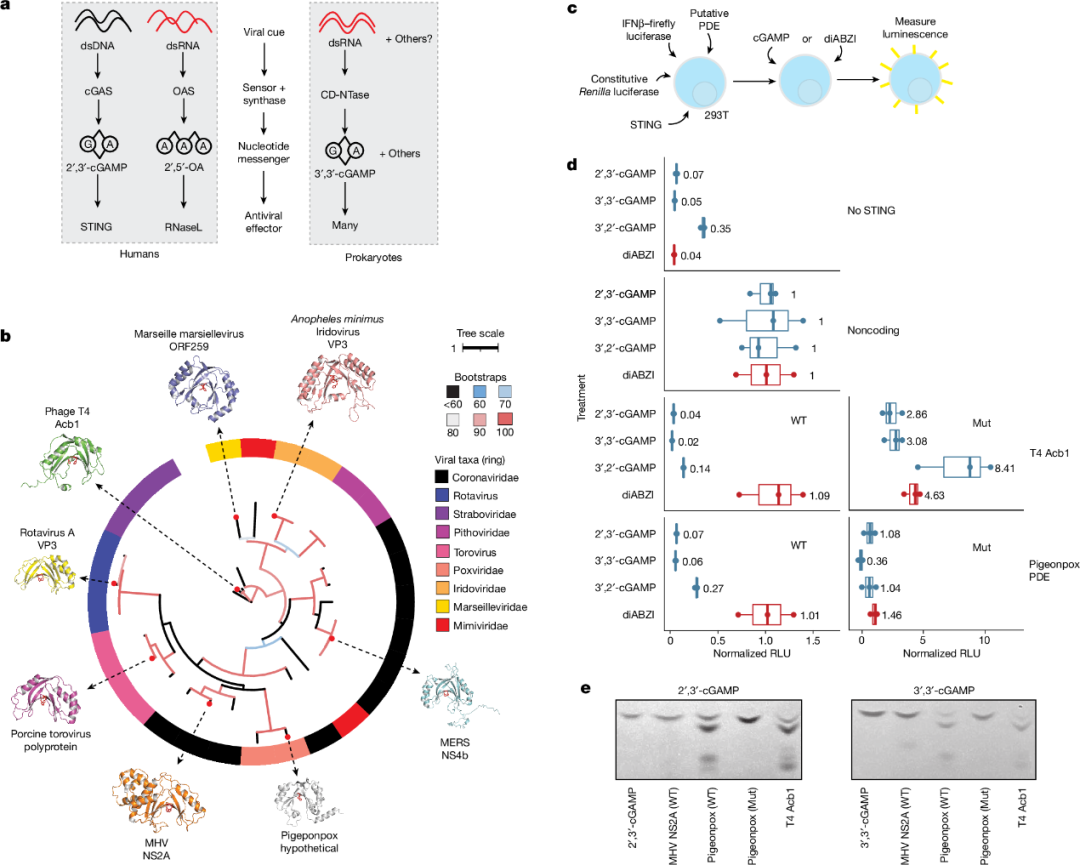
图 4
真核和原核生物的免疫系统有许多方面具有共同的起源。一组相关的途径是哺乳动物环状GMP-AMP合酶(cGAS)-STING和寡腺苷酸合酶(OAS)途径,以及原核生物基于环状寡核苷酸的抗噬菌体信号系统(CBASS)。在这两种情况下,蛋白传感器检测到病毒信号后会生成一个核苷酸二级信使,激活下游的抗病毒效应器(图4a)。在cGAS途径中,cGAS识别细胞质中的双链DNA并生成2′,3′-cGAMP。原核生物的CBASS系统中的许多cGAS/DncV样核苷酸转移酶(CD-NTases)在响应病毒信号时会生成类似的二级信使3′,3′-cGAMP。相比之下,OAS识别双链RNA(dsRNA)并生成线性2′,5′-寡腺苷酸。在原核生物中,噬菌体T4编码了类似LigT的PDE抗CBASS蛋白1(Acb1),该蛋白可以降解3′,3′-cGAMP以及其他多种环状核苷酸底物,包括2′,3′-cGAMP。
在真核生物中,一些RNA病毒编码能降解2′,5′-寡腺苷酸的PDEs。值得注意的是,作者发现这些PDEs具有与Acb1相似的LigT样折叠。鉴于LigT样PDE在病毒抗免疫中的保守使用,作者研究了它们的分布和系统发育。结构搜索显示,许多不同分支的LigT样PDEs存在于真核病毒中(图4b)。特别是,RNA病毒中存在多个独立的LigT样PDE分支。A系的冠状病毒和吐温病毒共享一支与轮状病毒中的PDE相似的分支。令人惊讶的是,C系冠状病毒包含一个不同的PDE分支,这表明在冠状病毒属内发生了两次独立的PDE获得事件,显示出冠状病毒在逃避OAS途径方面的强大选择压力。作者还发现一些大型DNA病毒也含有LigT样PDE。尽管在LigT样PDE树上氨基酸变化极大,但病毒LigT样PDE中的两个催化组氨酸几乎完全保守。
大型DNA病毒中存在LigT样PDE引发了它们是否具有抗免疫功能的疑问。尽管RNA病毒的LigT样PDE常常靶向RNA感应的OAS途径,但大型DNA病毒对OAS的靶向压力可能较小。因此,作者测试了大型DNA病毒编码的LigT样PDE对2′,3′-cGAMP的活性。首先,作者克隆并测试了一组LigT样PDE的表达,发现其中一些能够在哺乳动物细胞中良好表达。接着,作者在293T细胞中构建了一个合成的STING回路(图4c)。在这个系统中,STING可以通过cGAMP或非核苷酸STING激动剂diABZI处理来激活,从而以STING依赖的方式诱导萤火虫荧光素酶的表达。作者预期,靶向cGAMP的病毒LigT应该能够抑制cGAMP介导的STING活性,而不影响diABZI介导的STING活性。对表达良好的LigTs进行测试显示,由禽痘病毒编码的LigT样PDE对2′,3′-cGAMP介导的STING信号具有很强的活性,但对diABZI介导的STING信号活性有限。此外,催化组氨酸的突变显著降低了LigT样PDE的活性。接下来,作者测试了鸽痘LigT对一组cGAMP异构体(包括2′,3′-、3′,3′-和3′,2′-cGAMP)的活性。结果显示,与T4 Acb1类似,鸽痘LigT对多种cGAMP变体具有广泛的活性(图4d)。
为了确认鸽痘LigT能够降解cGAMP变体,作者纯化了野生型和突变型(H72A/H167R)鸽痘PDE,并通过薄层色谱(TLC)可视化2′,3′-和3′,3′-cGAMP的切割。结果显示,类似于噬菌体T4 Acb1,而不同于靶向2′,5′-寡腺苷酸的鼠肝炎病毒(MHV)LigT样PDE NS2a,鸽痘PDE能够切割2′,3′-和3′,3′-cGAMP(图4e)。
编译 | 黄海涛
审稿 | 曾全晨
参考资料
Nomburg, J., Doherty, E. E., Price, N., Bellieny-Rabelo, D., Zhu, Y. K., & Doudna, J. A. (2024). Birth of protein folds and functions in the virome. Nature, 1-8.

