DRUGAI
今天为大家介绍的是来自Matteo Dal Peraro团队的一篇论文。利用深度学习的进步,蛋白质设计和工程学正以前所未有的速度发展。然而,目前的模型在设计过程中无法原生考虑非蛋白质实体。这里,作者介绍了一种基于几何transformer的深度学习方法,该方法仅依赖于原子坐标和元素名称,能够在考虑多样分子环境约束的情况下,从骨架支架预测蛋白质序列。为了验证该方法,作者展示了它可以高成功率地生成高度耐热且具有催化活性的酶。这一概念预计将提高蛋白质设计流程的多功能性,以创造所需的功能。

从头设计蛋白质以工程化其功能特性是一个重大挑战,直接影响生物学、医学、生物技术和材料科学。一个关键应用领域是蛋白质疗法的工程化。它涉及创造专门针对特定疾病或状况的蛋白质,具有高度的精准性。这样的方法已被证明是小分子药物的有力替代方案。这为我们治疗许多健康问题打开了新的可能性,从自身免疫性疾病到癌症,相较于传统药物,提供了潜在的更有效和个性化的治疗方案。
此外,工程化酶的功能也是蛋白质设计的另一个有前景的挑战任务。酶作为天然催化剂,在各种生物过程中起着关键作用。通过设计新酶或修改现有酶,可以创造出能够促进自然界中稀有或不存在的反应的催化剂。这对许多行业都有深远影响,包括制药行业,定制酶可以更高效地合成复杂的药物分子;在环境技术中,设计的酶可能更高效地分解污染物或塑料。因此,蛋白质工程不仅在工业过程和环境可持续性方面展示了巨大的潜力,还为科学研究和生物技术创新开辟了新途径。
尽管基于物理的方法在蛋白质工程的进步中做出了贡献,深度学习方法最近通过提高蛋白质设计流程的成功率和多样性,带来了显著的加速。在最新的显著例子中,基于编码器-解码器神经网络的ProteinMPNN能够生成实验验证能按预期折叠的蛋白质序列。最近,结合了去噪扩散概率模型生成蛋白质骨架,ProteinMPNN和RFdiffusion显示出显著的成功。此外,基于混合蛋白质语言模型和结构模型的ESM-IF1能够生成超出已知天然序列范围的高度多样化蛋白质。该模型最近也通过实验验证,报告了非常高的成功率。更广泛地说,深度学习方法在该领域无处不在,广泛应用于多种蛋白质设计任务。例如,MaSIF专注于通过学习的蛋白质表面指纹设计蛋白质相互作用,或Chroma能够在任意约束条件下使用扩散方法生成蛋白质骨架和序列。
当前的蛋白质设计模型可以原生处理输入中的多个蛋白质链,从而设计相互作用蛋白质的序列。然而,这些模型在设计过程中处理非蛋白质实体的能力较差,限制了它们的多样性和适用范围。作者最近介绍了一种深度学习模型,旨在缓解这些限制,即蛋白质结构Transformer (PeSTo),这是一种几何transformer架构,操作于原子点云。它集成了深度学习中的不同进展,如transformer注意力机制,并使用标量和矢量状态来表示原子。通过元素名称和坐标唯一地表示分子,PeSTo可以应用于并预测与几乎任何种类分子(如其他蛋白质、核酸、脂类、离子、小配体、辅因子或碳水化合物)相互作用的蛋白质界面。
模型部分
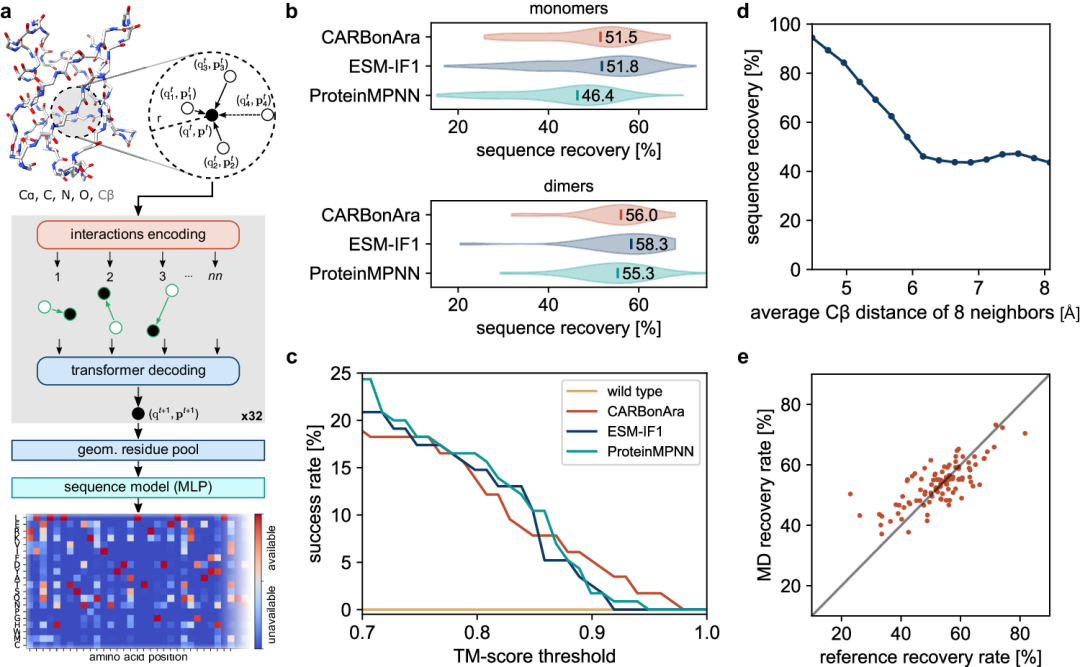
图 1
作者提出的CARBonAra基于PeSTo模型改进而来,CARBonAra使用由几何transformer组成的深度学习模型,预测在输入骨架支架的每个位置上出现特定氨基酸的可能性(图1a)。CARBonAra将骨架原子(Cα、C、N、O)的坐标和元素作为输入,并通过理想的键角和键长添加虚拟的Cβ原子。几何信息通过每个原子之间的距离和归一化的相对位移向量来描述。CARBonAra的核心是几何transformer操作,逐步处理从8到64个最近邻居的局部邻域信息。几何信息从矢量状态等变地编码,而标量状态表示在原子点云全局旋转下几何的不变量。几何transformer操作编码所有最近邻居的相互作用,并使用transformer处理标量和矢量信息,更新每个原子的状态。最后,通过将原子状态汇集到残基水平,作者训练模型以预测蛋白质序列每个位置的氨基酸置信度,以位置特异评分矩阵(PSSM,图1a)的形式表示。实际上,这些置信度可以通过表征每种氨基酸类型的预测置信度来解释并映射为概率。像其他模型如ProteinMPNN一样,CARBonAra支持自回归预测,通过使用独热编码将特定氨基酸的先前序列信息嵌入骨架原子。
最重要的是,CARBonAra继承了PeSTo的能力,仅使用元素名称和原子坐标工作,消除了对广泛参数化的需求,从而能够轻松适应各种场景。因此,CARBonAra可以解析和处理蛋白质骨架附近的任何分子实体,包括其他蛋白质、小分子、核酸、脂类、离子和水分子。利用CARBonAra的这种固有灵活性,作者将所有来自RCSB PDB的生物组装体纳入训练数据集,详见“方法”。这包括与其他分子实体(如离子、配体、核酸等)复合的蛋白质。训练数据集由大约37万个亚基组成,验证数据集中使用了额外的10万个亚基,所有这些都来自RCSB PDB中标注为最可能的生物组装体。测试数据集由大约7万个亚基组成,这些亚基与训练集没有共享的CATH域,并且序列同一性低于30%。这种选择标准确保了测试的稳健性,因为它排除了训练数据集中存在的相似折叠和序列。
与当前sota方法的性能比较
在没有非蛋白质分子背景下,针对骨架结构进行的独立蛋白质或蛋白质复合物的序列设计中,CARBonAra在序列预测方面表现与ProteinMPNN和ESM-IF1等最先进的方法相当(图1b),且计算成本具有竞争力(在GPU上的计算速度约为ProteinMPNN的3倍和ESM-IF1的10倍)。在从骨架结构重构蛋白质序列时,作者的方法在蛋白质单体设计中达到了51.3%的中位序列恢复率,而二聚体设计中达到了56.0%。尽管恢复率相似,三种方法的最佳序列之间的中位序列同一性范围为54%到58%。此外,作者观察到,在使用AlphaFold的单序列模式预测时,CARBonAra可以生成高质量的序列,按预期折叠,TM-score超过0.9(图1c)。作者还发现,CARBonAra学习了蛋白质核心更紧密的氨基酸包装,因此在蛋白质核心的恢复率较高,反映了典型的埋藏氨基酸对替换的较低耐受性(图1d),而在蛋白质表面则允许更高的变异性,除非提供额外的功能或结构约束。
从骨架支架预测序列的方法主要在具有理想骨架几何形状的实验数据上训练,这可能会在应用于生成的骨架时导致性能下降。在训练过程中向几何结构添加噪声可以缓解这个问题。作者通过将CARBonAra应用于分子动力学(MD)模拟的结构轨迹,来表征该方法的鲁棒性。作者观察到,由于骨架构象变化,序列恢复率从共识预测的54 ± 7%降至53 ± 10%,但没有显著下降,且在之前显示恢复率较低的情况下有所增加(图1e)。同时,作者观察到每个位置预测的可能氨基酸数量总体减少,这表明探索构象空间限制了序列空间,从而使设计目标结构构象成为可能。
序列空间采样
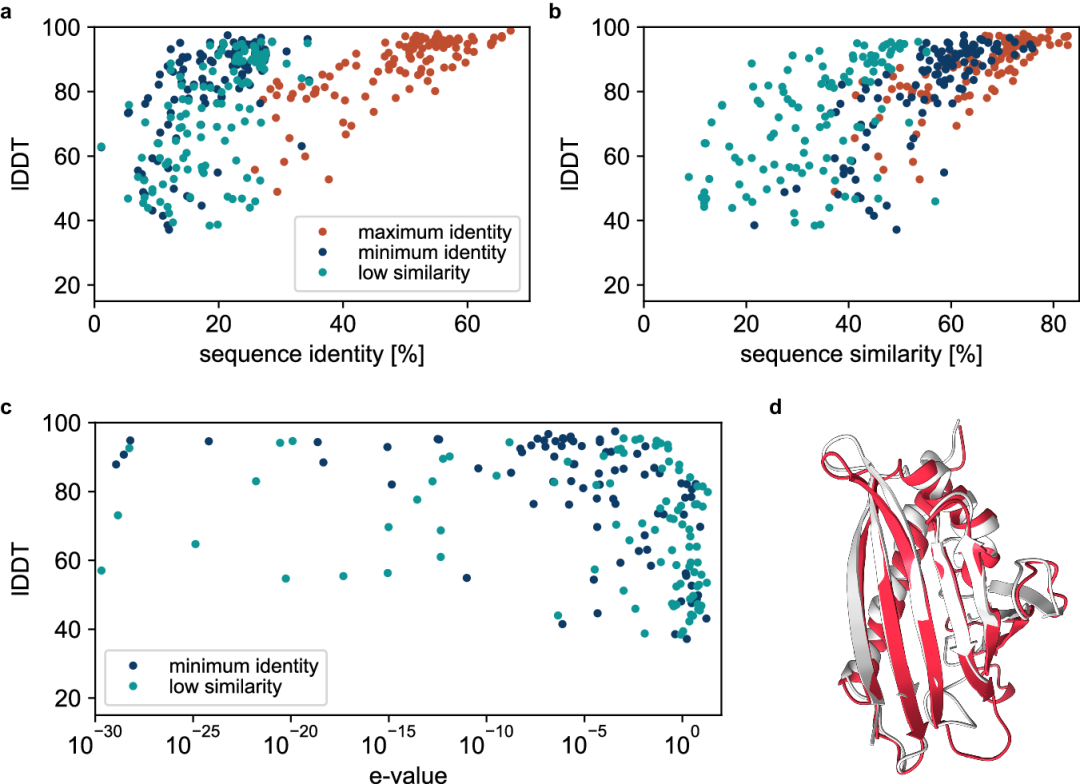
图 2
与其他蛋白质序列预测方法一样,特定位点的氨基酸置信度/概率需要通过某种方式采样,以生成实际蛋白质的序列。ProteinMPNN和ESM-IF1使用模型的logit输出作为玻尔兹曼分布的能量,来描述用户定义采样温度下的氨基酸概率。相比之下,CARBonAra使用多类氨基酸预测,生成一个潜在序列空间,为序列采样提供了多种可能性。例如,可以根据特定目标定制序列,如实现最小的序列同一性或低序列相似性,以设计具有特定折叠的独特序列。作者展示了可以生成同一性低至约10%和相似性低至20%的序列,同时仍能恢复与骨架结构接近的AlphaFold预测结构(lDDT > 80)(图2a, b)。一些生成的序列不仅与骨架蛋白不同,还与任何已知蛋白不同(图2c)。例如,其中一个案例使用桦树花粉过敏原Bet v 1蛋白(PDB ID: 6R3C)作为骨架。作者生成了一个与原始骨架具有7%同一性和13%相似性的序列,极限地推进了序列相似性。BLAST搜索显示没有显著匹配,生成序列的AlphaFold预测结构的lDDT为70(图2d)。
上下文感知的序列预测
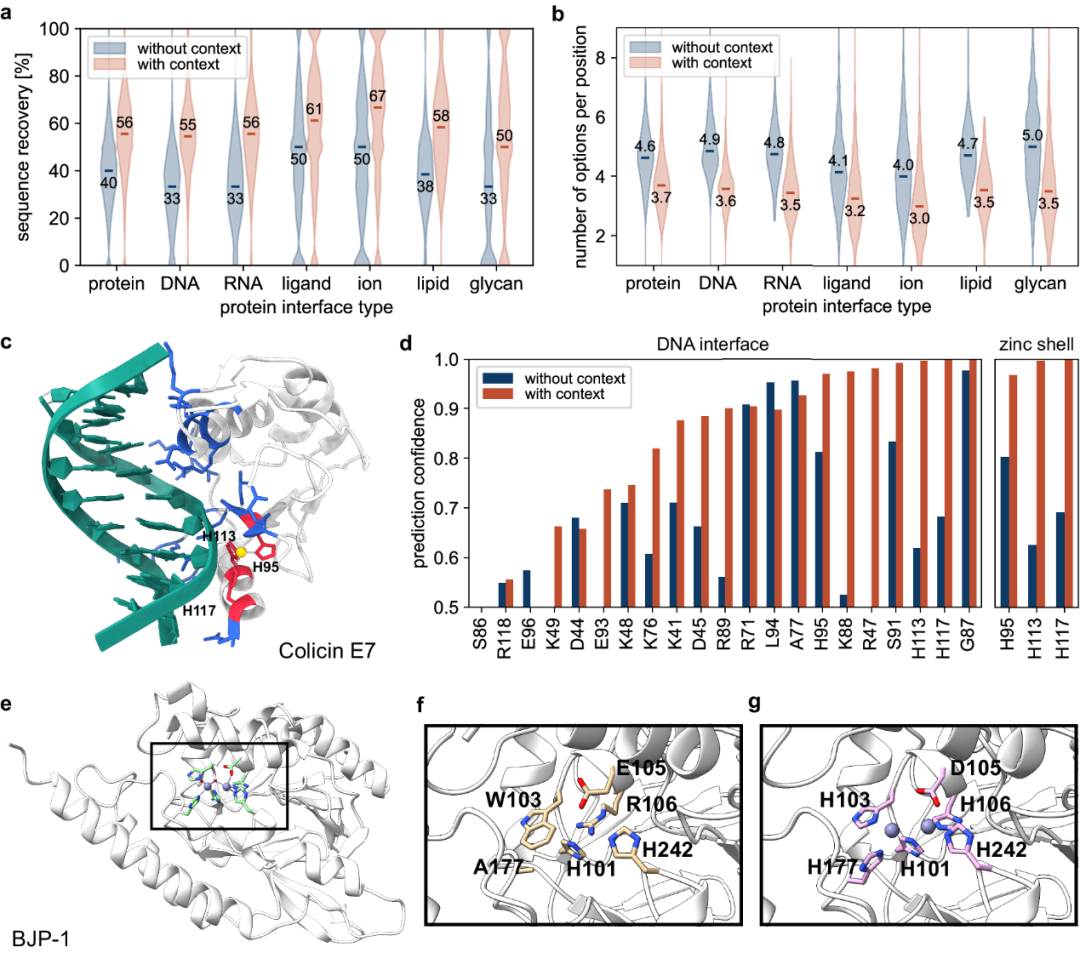
图 3
更重要的是,利用PeSTo的架构,CARBonAra能够在特定非蛋白质分子背景下进行蛋白质序列预测。在由与训练集不同折叠结构组成的测试集中,作者展示了当提供额外的分子背景时,总体结构的中位序列恢复率从54%提高到58%。特别是,CARBonAra在蛋白质相互作用界面上的中位序列恢复率达到了56%,在与核酸相互作用界面上达到了55%,即相较于无背景预测有显著提高(图3a)。同样,如果包含小分子实体如离子(67%)、脂类(57%)、配体(61%)和糖链(50%),蛋白质界面的恢复率也显著提高。这与PeSTo的最新发展相一致,作者在其中探讨了重新训练特定模型以解决碳水化合物-蛋白质界面的问题(PeSToCarbs)。包含这些分子不仅提升了它们周围的序列恢复率,还减少了需要采样的氨基酸种类(图3b)。
一个说明这种方法威力的示例是ColE7的核酸内切酶结构域,它以锌依赖方式与双链DNA相互作用(图3c)。当包含锌离子或12个碱基对的DNA双链并在原始结构中解析时,CARBonAra在金属和DNA界面的序列恢复率显著提高,从29%提高到52%(图3d)。因此,施加非蛋白质相互作用界面的存在可以显著提升序列恢复率,相对于ProteinMPNN(24%)和ESM-IF1(43%)的预测也是如此。有趣的是,当提供非原生分子背景(例如更大的离子,如钙)时,序列恢复率会下降。因此,离子口袋中预测的氨基酸置信度很大程度上依赖于给定的背景,这在BJP-1的案例中也得到了说明,BJP-1是一种锌依赖的金属β-内酰胺酶(图3e)。在BJP-1活性位点没有锌离子的情况下,CARBonAra的预测未能完全恢复锌配位残基(图3f)。通过保持结构中的锌离子,CARBonAra的背景感知能力使得在金属β-内酰胺酶活性位点的正确锌配位残基得以完全恢复(图3g)。这表明,不同原子的存在或缺失导致不同的预测,显示出CARBonAra对背景的高度敏感性。
蛋白质工程化一个β-内酰胺酶
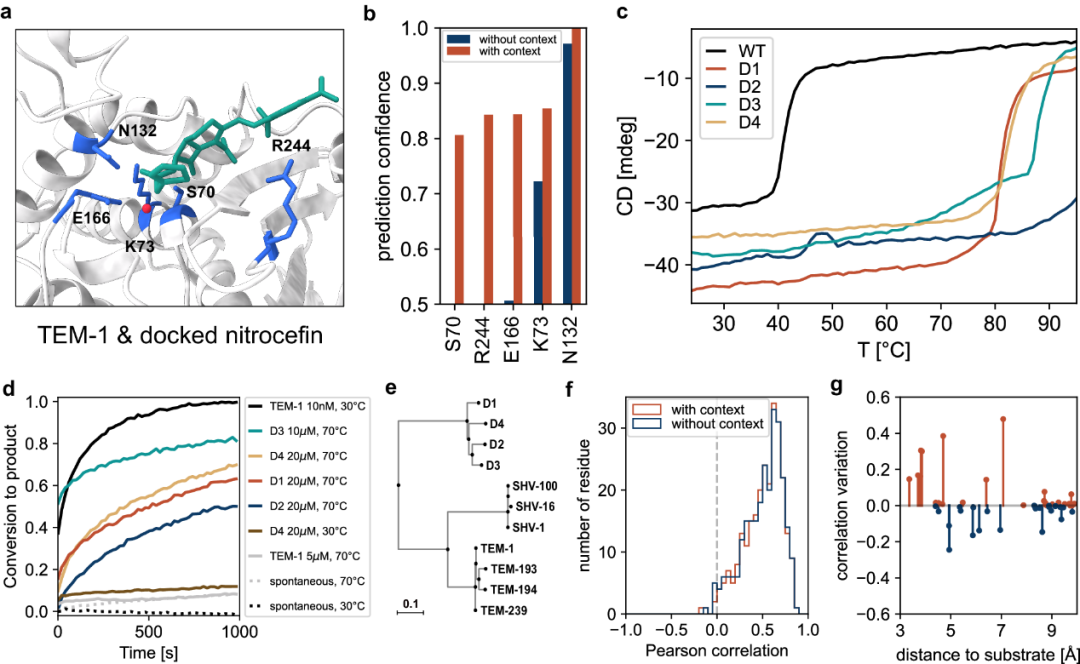
图 4
接下来,作者测试了CARBonAra的预测结果,通过设计酶的变体并在体外研究它们的结构和功能特征。对于酶设计而言,能够在所需底物或高亲和力配体提供的约束下设计序列是非常重要的。因此,作者使用了与β-内酰胺底物(即硝基头孢素)复合的TEM-1 β-内酰胺酶骨架支架来生成具有β-内酰胺酶活性的潜在新序列(图4a)。在没有背景的情况下,催化性S70和底物结合位点R244没有被预测出来,置信度分别为0.39和0.11(图4b)。然而,当在催化口袋中对接硝基头孢素后进行预测时,催化三联体S70、K73和E166,以及β-内酰胺结合所需的关键残基(即N132,R244)都具有较高的预测置信度(> 0.8)和低排名(前2名)。重要的是,在这种情况下,当考虑催化水时,序列恢复率达到最大,表明对分子背景具有非常高的敏感性。
为了测试设计的TEM样酶,作者使用带有对接硝基头孢素的印记法采样CARBonAra的预测结果。在CARBonAra中,印记法(imprinting)允许将任意序列信息识别为预测的先验信息,并应用于骨架支架中的任何位置。通过随机印记先前预测的氨基酸,该协议允许在采样序列中生成多样性,同时使用最大置信度预测,确保高质量序列。使用这种方法,作者生成了900个序列,并使用AlphaFold在单序列模式下提供的预测lDDT(pLDDT)对它们进行排名。单序列预测已被证明与实验成功相关,提供了一种评估新设计的折叠性和功能潜力的度量标准。
作者对pLDDT最高的前10个序列进行了体外验证。虽然这10个变体在大肠杆菌中都表现出高产量,但有4个变体在高浓度下也能溶解,并显示出良好折叠的蛋白质特征:远紫外圆二色谱特征与TEM-1相似,热变性曲线显示其稳定性明显更高(熔点约80°C或更高,而野生型TEM-1约为42°C),并呈现出两态展开行为(图4c);尺寸排阻色谱和SEC-MALS推导的分子量与单体状态一致,核磁共振谱中有良好分布的共振峰。所有4个设计的突变体在30°C时对硝基头孢素表现出弱到无的酶活性,但在70°C时表现出显著活性,而天然β-内酰胺酶TEM-1在该温度下完全失活(图4d)。从TEM-1在30°C和设计的β-内酰胺酶在70°C时达到相似活性所需的浓度,作者估算后者在该温度下的催化效率较低。在这种情况下,需要强调的是,相对于TEM-1的大幅度热稳定性(高出40°C或更多)赋予了设计的突变体广阔的空间来进化其催化特性。与其他A类β-内酰胺酶相比,这些新设计的序列与TEM-1 β-内酰胺酶的同一性最多约为55%,与同家族的其他蛋白(如SHV、KPC或CTX-M)的同一性约为30%到50%。它们在自身谱系内的多样性比其他家族更大(图4e),有效地在A类β-内酰胺酶内聚集成一个新的家族级别的群体。
编译|黄海涛
审稿|曾全晨
参考资料
Krapp, L. F., Meireles, F. A., Abriata, L. A., Devillard, J., Vacle, S., Marcaida, M. J., & Dal Peraro, M. (2024). Context-aware geometric deep learning for protein sequence design. Nature Communications, 15(1), 6273.

