DRUGAI
今天为大家介绍的是来自E. A. Huerta团队的一篇论文。蛋白质三维结构从氨基酸序列的预测是生物物理学中的一项计算性重大挑战,并在稳健的蛋白质结构预测算法中起着关键作用,应用范围从药物发现到基因组解读。随着AlphaFold等人工智能模型的出现,依赖于稳健蛋白质结构预测算法的应用正在发生革命性变化。为了最大化这些AI工具的影响力,并简化其使用,作者推出了APACE(AlphaFold2与超级计算相结合),这是一个有效处理该AI模型及其TB级数据库的计算框架,能够在现代超级计算环境中加速进行蛋白质结构预测分析。作者将APACE部署在Delta和Polaris超级计算机上,并使用四种典型蛋白质(6AWO、6OAN、7MEZ和6D6U)量化了其在准确蛋白质结构预测中的性能。利用多达300个组合,分布在200个NVIDIA A100 GPU上,作者发现APACE的速度比现成的AlphaFold2实现快了多达两个数量级,将解决时间从几周缩短到了几分钟。这种计算方法可以很容易地与机器人实验室结合,以实现科学发现的自动化和加速。

人工智能与超级计算相结合的创新正在推动科学和工程领域的突破性进展。GPT-4、AlphaFold等AI模型的崛起,为加速和自动化科学发现提供了新的能力。然而,这些模型中有一些并未向公众开放,打破了AI社区内的一项重要传统。据称,这些AI模型的庞大规模使得许多潜在用户难以使用。
为了解决这一问题,作者展示了如何将大型AI模型与高性能计算平台结合起来,以帮助广泛的用户群体充分利用AI的能力进行科学发现。作者选择了AlphaFold2作为本研究的科学驱动因素,因为这一AI模型正在革新生物物理学领域的发现,并且其在准确且快速的蛋白质结构预测(PSP)中的应用需要最优利用现代超级计算环境。在此,作者展示了如何优化AlphaFold2及其超过2.6 TB数据存储的数据库,以将准确PSP所需的时间从几周缩短到几分钟。
AlphaFold2的特点:AlphaFold2利用中央处理器(CPU)计算关键的输入特征:多序列比对(MSA)和结构模板。MSA代表与查询蛋白相关的蛋白序列同源体的集合。MSA捕捉了不同蛋白质之间的进化关系,例如保守的和变异的氨基酸残基。MSA使用基于CPU的序列比对算法计算,例如Jackhmmer,它将查询蛋白质序列与从数据库(如Uniclust)中获取的已知序列同源体进行比对。AlphaFold2可以从MSA中提取关键的残基相互作用。此外,结构模板是指与查询蛋白质具有显著序列相似性的实验已知的蛋白同源体结构。这些模板结构用于提高AlphaFold2预测的准确性。基于CPU的算法,如HHsearch(用于单体)和Hmmsearch(用于多聚体),在公共蛋白质结构数据库如蛋白质数据库(PDB)中进行搜索。然后,AlphaFold2从模板中提取与查询蛋白相关的空间信息。
在图形处理单元(GPU)阶段,AlphaFold2利用从MSA和模板生成的特征,通过evoformer网络进行处理。Evoformer通过交叉方式迭代地在MSA和配对交互之间交换信息,以提取氨基酸残基关系。更新后的表示进入结构模块,其中对每个残基的位置进行旋转和翻译的预测。
生成的预测3D结构通过分子动力学(MD)引擎的最小化过程进行松弛以提高准确性。生成最终结构后,信息循环回到evoformer模块的起点,以进一步优化结构预测。总的来说,AlphaFold2是端到端训练的,具有卓越的准确性和可靠性,能够预测蛋白质的三维结构。
APACE对AlphaFold2的改进:作者引入了APACE,即AlphaFold2和超级计算结合服务,这是一种通过CPU和GPU优化以及在超级计算环境中的分布式计算来加速AlphaFold2的计算框架。该方法的关键特点包括:
数据管理:首先,APACE通过在Delta和Polaris超级计算机上托管AlphaFold2的2.6 TB AI模型和数据库,来简化其使用。AlphaFold2的神经网络可以通过利用固态硬盘(SSD)数据存储和无限内存引擎(IME)数据分层,轻松访问数据。
CPU优化:APACE使用Ray库进行CPU优化,以并行化CPU密集型的MSA和模板计算。作为CPU优化的一部分,APACE为MSA/模板搜索工具分配了更多的CPU核心,而不是默认的4或8个核心,这在实验中显示出启发式的速度提升。此外,作者还实现了一个检查点,如果存在`features.pkl`文件(即存储MSA/模板搜索结果的中间文件),可以跳过冗余的MSA/模板步骤。
GPU优化:第三,APACE使用Ray库进行GPU优化,以并行化GPU密集型的神经网络蛋白质结构预测步骤。与ParaFold的GPU加速不同的是,ParaFold仅使用model_1(一个基于模板的预训练模型)对肽序列(例如,平均长度小于100个氨基酸残基)进行一个构象预测,而不是对蛋白质序列(即大约400个氨基酸残基或更多)进行预测。与此形成鲜明对比的是,APACE可以为每个蛋白质序列预测多个蛋白质构象,并在必要时为肽序列预测,使用所有五个预训练模型并行进行,这对计算要求更高。
新功能:第四,APACE可以为每个预训练神经网络模型(共五个模型)预测多个单体构象,这在原始AlphaFold2模型中仅在多聚体预测中存在。APACE还包括其他功能,如在结构预测期间启用dropout、更改Evoformer的循环次数或对MSA选项进行子采样。
作者完成了三项计算实验,以详细比较APACE与原始AlphaFold2模型的性能。作者将逐一描述每个实验,并提供相应的结果。
实验1:预测四种基准蛋白质的结构
作者选择了四种蛋白质作为基准,以评估APACE的有效性和操作性能。为了使用APACE预测蛋白质结构,作者开发了科学软件,使用户能够在sbatch脚本中提供合适的headers,并加载适当的环境和模块,从而成功地在Delta和Polaris超级计算机上提交并完成模拟。
单体蛋白质:作者使用单体蛋白质6AWO(血清素转运蛋白)作为基本结构,利用五个模型测试基线预测的准确性和构象多样性。因此,作者创建了一个由八个NVIDIA A100/A40 GPU(相当于Delta和Polaris中的2个A100/A40 GPU节点)组成的Ray集群,以便为所有五个模型(即每个模型一个结构)促进CPU和GPU的并行执行和松弛优化。
多聚体蛋白质:对于多聚体蛋白质,作者测试了6OAN(与单链可变片段抗体结合的Duffy结合蛋白),7MEZ(磷脂酰肌醇3-激酶),以及6D6U(一种具有三个不同链的五聚体GABA转运蛋白),这些蛋白质代表了多聚体预测中的更具挑战性的案例。对于每种蛋白质,作者为每个模型进行了八次结构预测,总共生成了40个预测(五种集合模式 × 每个模型八次预测)。为了同时执行和松弛全部40个模型,使用了Ray集群并调动了40个NVIDIA A100和40个A40 GPU(10个A100/A40 GPU节点)。
CPU加速:通过实施并行优化技术,APACE在Delta超级计算机上实现了1.8倍的平均CPU加速,在Polaris超级计算机上实现了1.78倍的平均CPU加速。这些结果与计算节点的数量无关。
GPU加速:APACE在GPU加速方面也表现出显著的提升。以下结果是在6AWO使用8个GPU,以及在6OAN、7MEZ和6D6U上使用40个GPU时获得的:
1. 6AWO:在Delta超级计算机上,A40和A100 GPU分别实现了4.4倍的加速;在Polaris上实现了4.98倍的加速。
2. 6OAN:在Delta超级计算机上,A40和A100 GPU分别实现了34倍和32.4倍的加速;在Polaris上实现了40.1倍的加速。
3. 7MEZ:在Delta超级计算机上,A40和A100 GPU分别实现了37.5倍和37.1倍的加速;在Polaris上实现了40倍的加速。
4. 6D6U:在Delta超级计算机上,A40和A100 GPU分别实现了21.2倍和22倍的加速;在Polaris上实现了40倍的加速。
表1:在现成的AlphaFold2和APACE CPU&GPU优化框架之间的性能基准测试
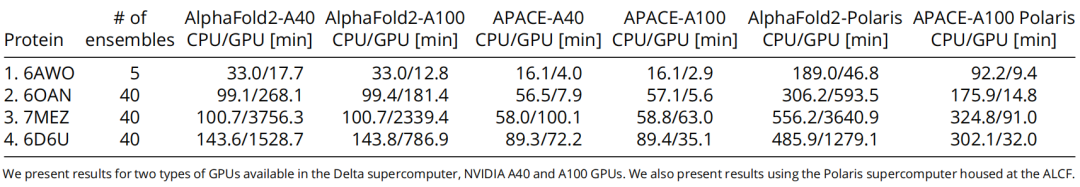
作者在表1中总结了这些结果。值得注意的是,使用NVIDIA A100 GPU时,预测时间始终较短。简而言之,APACE为基础和复杂结构提供了显著的加速,同时保留了原始AlphaFold2模型的准确性和稳健性。
实验2:使用100和200个NVIDIA A100 GPU预测蛋白质7MEZ
为了量化APACE在Delta和Polaris超级计算机上的性能和可扩展性,作者使用大量计算节点进行了蛋白质7MEZ的预测。具体来说,作者利用了100个NVIDIA A100 GPU,相当于25个A100 GPU计算节点来生成预测(每个模型20次预测)。同样,作者利用了200个NVIDIA A100 GPU的计算能力,相当于50个A100计算节点,总共生成了200次预测(每个模型40次预测)。
表2:在现成的AlphaFold2和APACE对于蛋白质7MEZ的性能基准测试
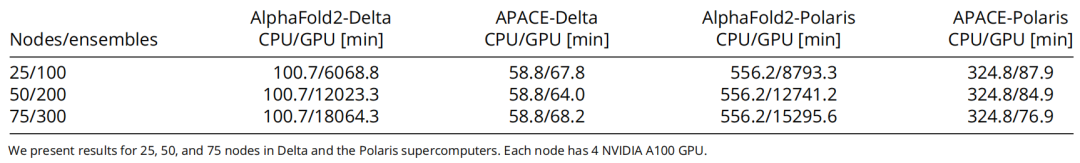
如表2所示,APACE实现了显著的加速效果。当作者在100个GPU上计算7MEZ的100个集合时,APACE在67.8分钟内完成了所需的计算,而在Delta超级计算机上使用AlphaFold2则需要6068.8分钟(101.1小时/4.2天)。在Polaris超级计算机上,APACE将解决时间从8793.3分钟(146.5小时/6.1天)减少到87.9分钟。
类似地,如果作者需要为同一蛋白质计算200个集合,APACE在Delta超级计算机上在64分钟内完成了所有预测,而使用原始AlphaFold2方法则需要12023.3分钟(200.4小时/8.3天)。在Polaris超级计算机上,APACE将解决时间从12741.2分钟(212.4小时/8.8天)减少到仅84.9分钟。
最后,当使用300个集合进行7MEZ蛋白质的预测时,APACE在Delta超级计算机上在68.2分钟内完成了所有预测,而使用原始AlphaFold2方法则需要18064.3分钟(301.1小时/12.5天)。在Polaris超级计算机上,APACE将解决时间从15295.6分钟(254.9小时/10.6天)减少到仅76.9分钟。
实验3:APACE的集合多样性
AlphaFold2的固有限制使作者只能为每个单体生成五个预测,例如每个模型一个预测,从而限制了蛋白质构象的多样性。此外,诸如dropout之类的参数微调也无法实现。然而,作者通过调整ColabFold的代码成功解决了这一限制。
APACE允许用户选择以下选项:
1. 使用`-num_ensemble`设置结构模块的集合数量,
2. 使用`--num_recycles`控制循环次数,
3. 使用`--max_seq`和`--max_extra_seq`对MSA进行子采样,
4. 使用`--use_fuse`进行Evoformer融合,
5. 使用`--use_bfloat16`进行Bfloat16混合精度计算,
6. 使用`--use_dropout`进行基于伯努利掩码的多样化构象采样。
APACE的蛋白质结构预测和构象多样性。作者修改了AlphaFold2的代码,以反映ColabFold灵活的蛋白质结构预测参数定制。通过这些改进,作者成功地扩展了预测的范围,从而增强了预测结构的整体可靠性。虽然蛋白质结构预测具有重要意义,但作者希望扩展APACE以预测构象多样性,因为蛋白质并非静态,而是可塑且灵活的结构。这对广泛的构象集合进行采样对于药物发现至关重要。
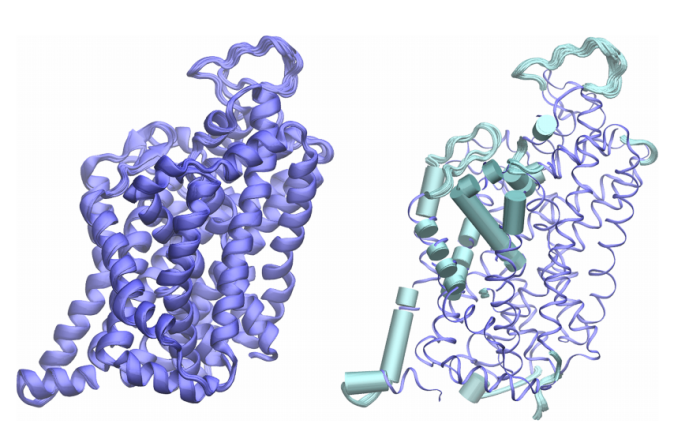
图 1
在6AWO(约500个氨基酸残基)的情况下,如图1所示,作者使用了参数定制增强(选择了`--use_dropout=True`选项),并预测了血清素转运蛋白(SERT)的100个结构。作者发现APACE预测的结构与真实结构相当。当作者可视化变异较大的跨膜域α螺旋时(右图中的青色部分),可以看到TM2、TM6、TM10和TM12突出显示。其中,TM6、TM10和TM12负责构象变化或配体从外向内的结合,这表明APACE学会了预测SERT的广泛构象范围。APACE对SERT的预测是准确的,SERT是一个完整的膜蛋白。即使在没有膜的情况下,APACE也能准确预测跨膜域,展示了APACE在药物发现研究中的潜力。
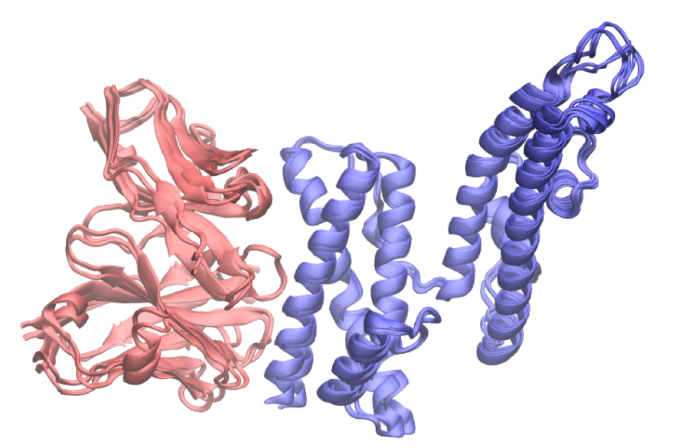
图 2
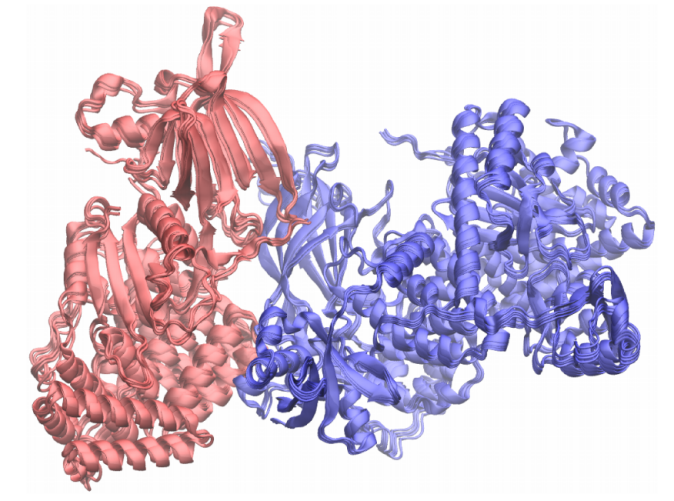
图 3
对于6OAN、7MEZ和6D6U(分别约600、2000和1800个氨基酸残基),作者进行了多聚体预测。图2和图3显示,6OAN和7MEZ都高精度地预测了异二聚体结构的构象集合。尤其是界面结合姿态的预测与真实结构相当,尽管在预测的次级结构中可能存在与界面结合无关的轻微错误,但通过非键合相互作用预测的蛋白质之间的正确界面结合姿态更为重要。这些轻微的错误可以通过分子动力学、蒙特卡罗方法和蛋白质设计工具来解决。
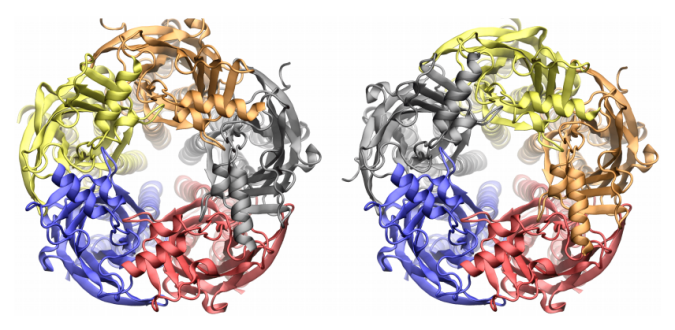
图 4
在图4中,对于6D6U,可以观察到与真实结构相当的预测结构(左图),以及由于同二聚体位置预测错误而导致的错误结构(右图)。由于6D6U是一个膜五聚体异源蛋白,预测每个单体的正确结构以及交替的链模式是一个具有挑战性的任务。因此,跨膜螺旋的预测可能不准确,但整体结构仍然与真实结构相当。
总之,作者展示了APACE在预测蛋白质结构方面的能力,反映了AlphaFold2的稳健性和准确性,并提供了显著的加速效果,将解决时间从数天缩短至数分钟。然而,在预测跨膜蛋白质和/或多链多聚体时,APACE可能会受到限制,这些限制是从AlphaFold2继承而来的。
结论
作者引入了APACE,这一框架保留了AlphaFold2的稳健性和准确性,并利用超级计算技术将从数据到洞察的时间从几天缩短到几分钟。作者通过以下方式实现了这一目标:a) 有效利用Delta和Polaris超级计算机系统的数据存储和数据分层;b) 优化了CPU和GPU的计算性能;c) 开发了科学软件,使得能够预测蛋白质结构的构象集合。这些工具与本文一起发布,旨在为研究人员提供一个计算框架,可以方便地与机器人实验室相结合,以实现科学发现的自动化和加速。
编译 | 于洲
审稿 | 曾全晨
参考资料
Park H, Patel P, Haas R, et al. APACE: AlphaFold2 and advanced computing as a service for accelerated discovery in biophysics[J]. Proceedings of the National Academy of Sciences, 2024, 121(27): e2311888121.

