DRUGAI
今天给大家介绍的是来自湖南大学信息科学与工程学院曾湘祥教授团队发表在 IJCAI 2024 上的工作,题目为“An Image-enhanced Molecular Graph Representation Learning Framework”。该研究提出了一种基于图像的跨膜态知识蒸馏框架并成功改善了基于分子图方法(但不限于该类方法)的性能。该方法是一种通用的、即插即用的框架并有望拓展到更多的领域。

摘要
提取丰富的分子表征是精确药物发现的重要前提。近来,分子表征学习方法取得了令人印象深刻的进展,但是从单一模态学习的范式逐渐遇到了表征能力有限的瓶颈。在本文中,作者充分考虑了3D构象分子图像中包含的丰富视觉信息(即纹理、阴影、颜色和平面空间信息),并提炼基于图的模型以进行更具判别力的药物发现。具体来说,作者提出了一个图像增强的分子图表示学习框架(称为IEM),它利用从3D构象中提取的多视图分子图像来增强分子图表征。为了从多视角图像中提取有用的辅助知识,本文设计了一个教师模型,通过五个精心设计的预训练任务对具有3D构象的200万个分子进行预训练。同时,为了将知识从教师模型迁移到基于图的学生模型,本文提出了一种有效的跨模态知识蒸馏策略,包括知识增强器和任务增强器。值得注意的是,IEM的蒸馏架构可以直接集成到现有的基于图的模型中,并显著提高了这些模型的分子表征学习能力。特别是,配备IEM的GraphMVP和MoleBERT在MoleculeNet基准上实现了新的最先进的性能。
背景介绍
目前,分子表征学习在高精度药物发现(如分子性质预测、靶标活性预测)中发挥着重要作用。许多研究者考虑到单一模态的限制,提出基于多模态学习的方法用于药物发现,如 GraphMVP、3D InfoMax。然而,这些多模态学习方法所获得的性能增益依然有限。作者认为原因有以下两点:(1)相似的模态(2D graph 和3D graph)和编码方式(基于gnn的方法) ;(2)在分子的基本属性上进行特征提取的能力较弱,导致模态之间的互补信息不足。图像是一种在模态特征和编码方式上与图有着明显差异性的模态。我们通过经验证明,2D图与3D图具有较高的皮尔逊相关性,与图像的皮尔逊相关性明显较低。
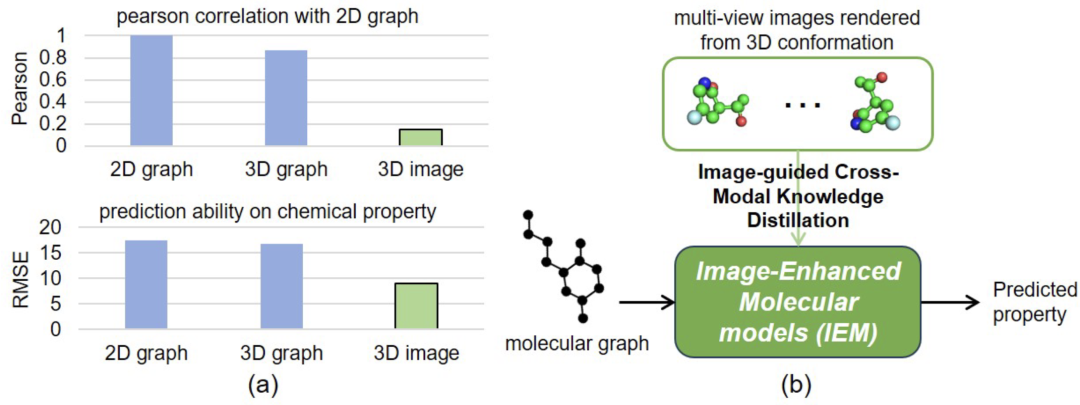
图 1
如图1(a)所示,2D图与3D图具有高度的皮尔逊相关性与图像具有显著的低皮尔逊相关性。与图不同,图像从视觉角度理解分子,其中包含分子丰富的纹理信息,并允许直接可视化空间排列,而不引入任何构象,如分子的手性变化。受到知识蒸馏(KD)的启发,作者将信息传递的过程描述为如何用一个知识渊博的老师(图像)来教一个优秀的学生(graph)。KD的成功取决于好的老师模型和好的KD策略。为了获得知识渊博的基于图像的教师模型,作者提出了一种具有 3D 构象的多视图分子图像表征学习方法并通过 5 种预训练策略来获取丰富的特征表示。考虑到跨模态特征之间简单的特征对齐会导致有限的增益甚至负迁移在跨模态 KD(CMKD)阶段,作者设计了一种新颖的 CMKD 框架(称为 IEM)来丰富图的特征,其减轻了知识增强器和任务增强器在 logit 空间中对齐图和图像的问题,从而避免了特征空间中的模态差距。如图1(b),展示了IEM将图像中知识蒸馏到图中的示意图。
模型方法
流程
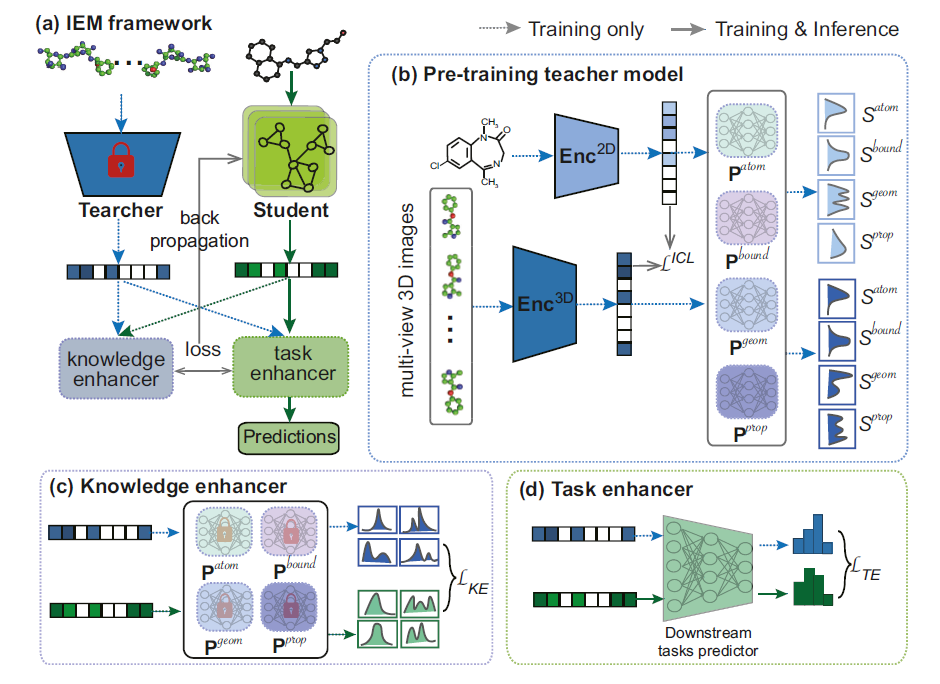
图 2
IEM的整体概述如图2所示,主要分为三个部分:
如图2(b)所示,使用5个预训练任务来训练知识渊博的基于图像的老师模型;
如图2(c)和图2(d)所示,定义了2个用于知识蒸馏的增强器(知识增强器和任务增强器)。
如图2(a)所示,利用基于图像的老师和2个增强器来迁移图像中的知识到基于图的学生中。
接下来,将介绍所提出方法的细节。
如图2(b)所示,作者将由RDKit生成的低计算成本的2D图像和由PyMol渲染的信息丰富的3D图像结合起来,两者都可以用来增强图表征。本文中使用3D图像编码器作为老师模型,因为它更具判别力。同时,为了更好的处理3D构象信息,作者选择了4个固定的视角来生成多视角图像。由于下游任务的不确定性,教师应具有较强的知识泛化能力。因此,作者基于泛化知识应反映分子基本属性信息的原则,设计了5个预训练任务,包括(1) 2D和3D图像之间的对比学习(ICL);(2)原子分布预测任务(ADP);(3)化学键分布预测任务(BDP);(4)几何分布预测任务(GDP);(5)属性分布预测任务(PDP)。
其中ICL的主要目的是提取分子图像中的固有信息。同时,二维图像编码器具有对三维信息的感知,三维图像编码器具有对全局信息的感知,以缓解视图遮挡问题。具体而言,作者表示FI = {F2D, F3D}。在InfoNCE之后,2D和3D图像之间的对比学习损失被形式化为如下:
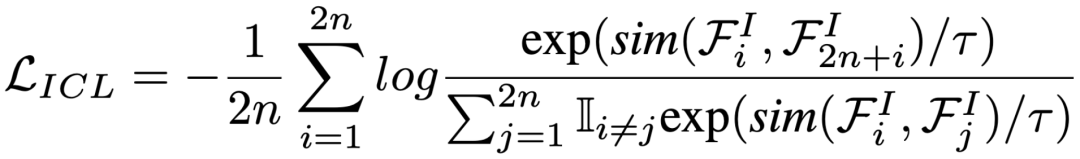
ADP、BDP、GDP和PDP任务的动机是让教师获得泛化的知识。这4个预测任务中的2D图像编码器和3D图像编码器的损失可表述如下:
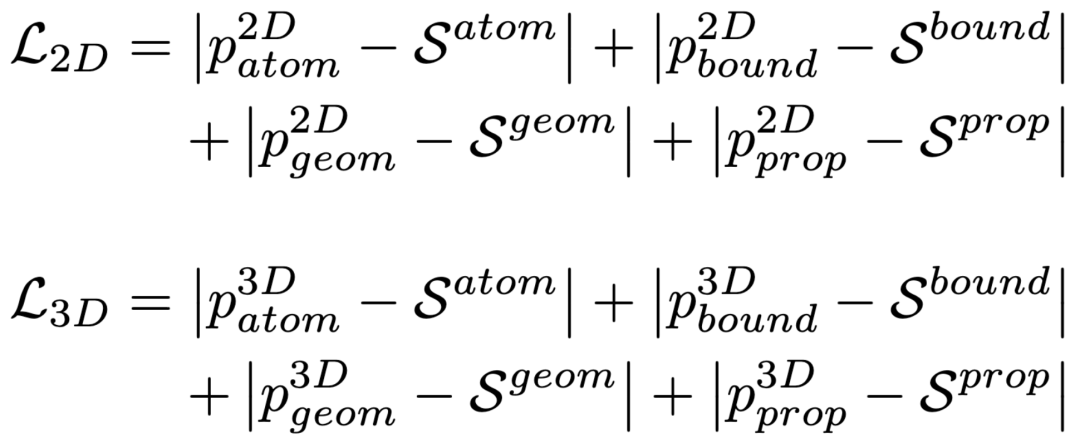
最后,2D图像编码器和3D图像编码器可以用以下总损失进行预训练:
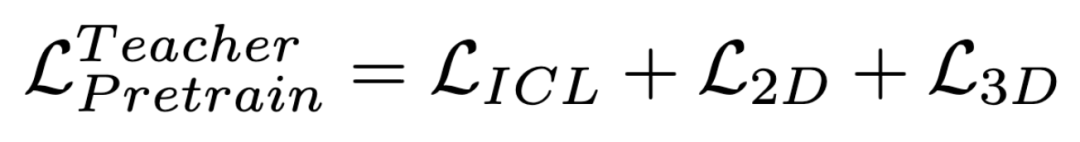
如图2(c)所示,知识增强器的目的是从教师那里提炼出与任务无关的泛化知识。知识增强器接受F3D和Fg作为输入,使用4个冻结的预测器Patom、Pbound、Pgeom、Pprop来预测不同知识水平的分子分布,可以表示为{pvatom,pvbound, pvgeom, pvprop}和{pgatom, pgbound, pggeom, pgprop}。作者将老师的预测标签作为ground truth并利用 L1 损失来监督学生,损失函数可以被形式化为如下:
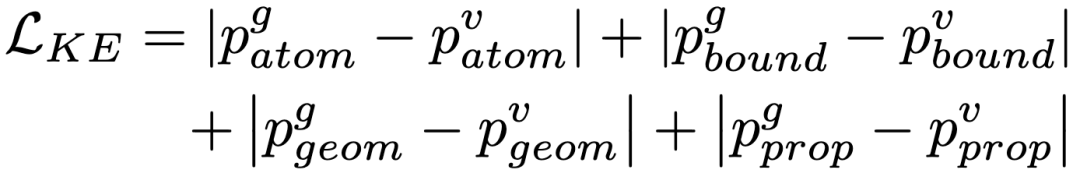
如图2(d)所示,任务增强器的目的是从教师那里提取与下游任务相关的知识,可以形式化为:
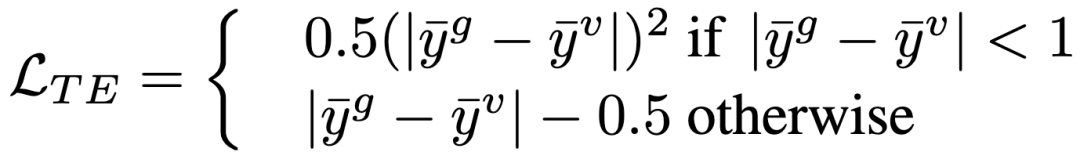
为了提高效率,通常希望在推断时使用单一模态而不是多个模态。为此,作者进一步利用来自下游任务的真实的ground-truth 来训练学生,损失形式化为:
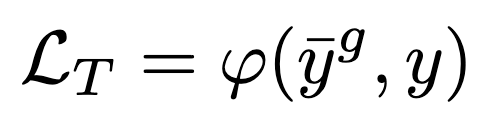
最终,总体的损失可以形式化为:
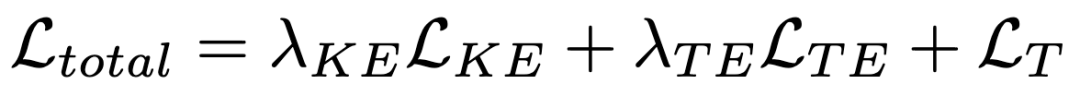
在推理过程中,通过将图数据输入到学生中,就可以得到预测结果。
实验结果
数据集和评估指标:对于预训练教师模型,作者从PCQM4Mv2数据库中采样了200万个具有3D构象的未标记分子。在评估阶段,作者使用来自MoleculeNet的8个广泛使用的二元分类数据集并根据ROC-AUC指标对模型进行评估。为了进一步更广泛的评估模型性能,作者还使用均方根误差(RMSE)指标评估了IEM在GraphMVP中包含的4个回归数据集上的有效性。
实现细节:在预训练教师模型阶段,作者使用2个独立的ResNet-18作为2D和3D图像编码器的架构。2D图像编码器直接从单个2D分子图像中提取512维视觉特征,而3D图像编码器通过对多视图3D图像的特征进行逐视角平均池化(view-wise mean-pooling)获得512维视觉特征。
主要结果
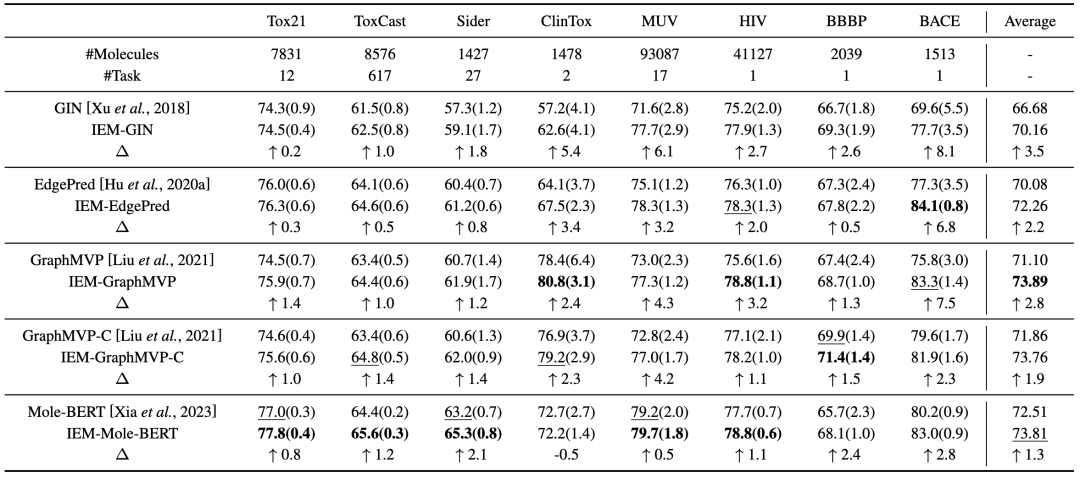
表 1
在表1中,用5个基线模型(GIN、EdgePred、GraphMVP、GraphMVP-C和Mole-BERT)评估了IEM在8个分子性质预测数据集上的性能并报告了主要结果。从上述实验结果表1中可以观察到,配备IEM的基线达到了最先进的性能。值得注意的是,在配备IEM后,所有基线的性能都得到了一致的改善。平均ROC-AUC的绝对改善幅度在1.3%至3.5%之间。为了验证IEM的性能提高不是由基线的标准差引起的,作者统计了高于基线的α=平均值+标准差的结果,发现70%(40个中的28个)的结果优于基线的α。特别是GIN,除Tox21外,其他改善均优于α基线。
随后,作者在更广泛的药物发现任务进行进一步评估,包括4个回归基准。如表2所示,发现与分类任务相同的结论,即IEM综合改善了所有基线,最大相对改善率为8.56%,均达到了最先进的性能。因此,IEM有望作为一种通用插件来改进任何基于图的模型。
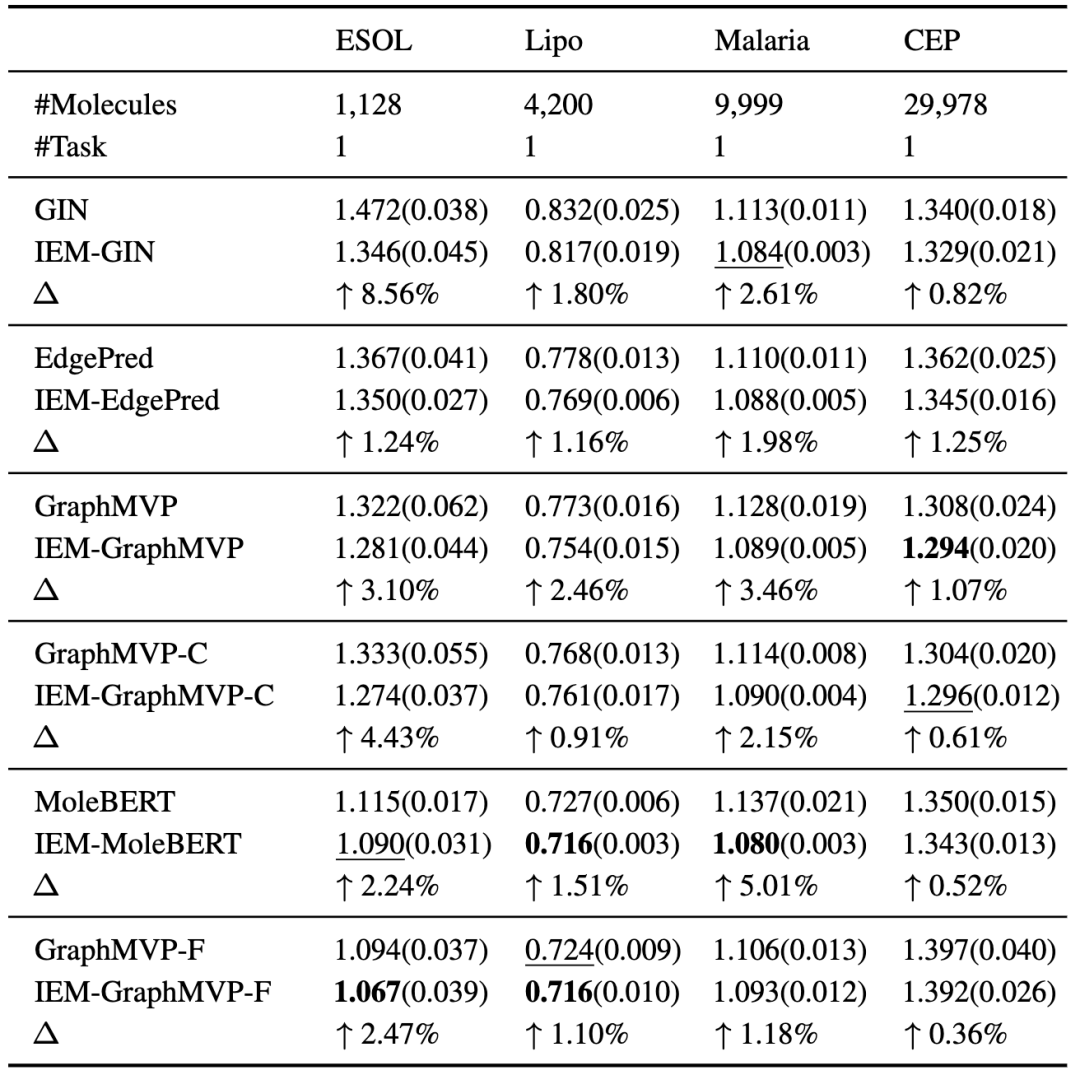
表 2
不同的GNN架构
为了验证IEM在不同GNN体系结构上的有效性,使用了4种不同的GNN体系结构,包括GCN、GIN、GAT和GraphSAGE。表3给出了不同GNN架构在8个分类数据集上的平均ROC-AUC结果。发现IEM可以显著改善不同的GNN架构的性能, 相对性能提高3.92%至5.23%。特别是,观察到配备IEM的GIN与InfoGraph、EdgePred和3DInfoMax等预训练模型相比具有一定竞争力。这表明IEM具有较强的泛化能力,即使不进行任何预训练,也能达到与预训练相当的性能。
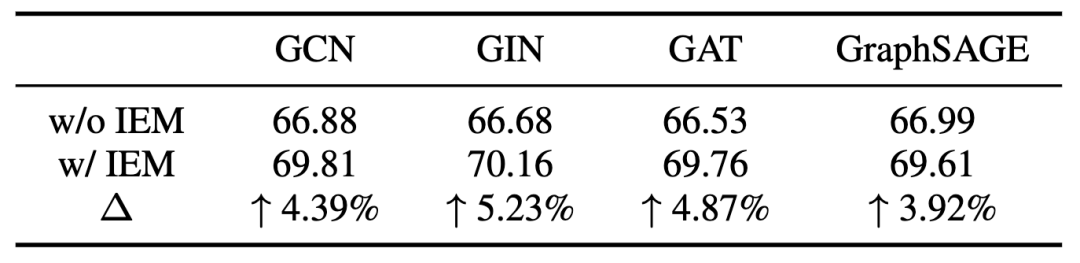
表 3
不同的图像渲染策略
为了探索 IEM 对无构象分子的性能,作者讨论了不同图像渲染策略的影响,包括由 RDKit 和 PyMol 渲染的无构象 2D 图像。如表4所示。作者发现IEM可以提高EdgePred和GraphMVP的性能,在所有的渲染策略中平均性能提升超过2%,这表明IEM可以成功兼容无构象的2D图像。作者还发现,IEM 在 RDKit 和 PyMol 渲染的 2D 图像上的性能相似(72.21% vs. 72.00% 和 73.34% vs. 73.41%),这表明在有限的计算资源条件下,使用更经济的 RDKit 来改进基于图的模型是一种较好的选择。
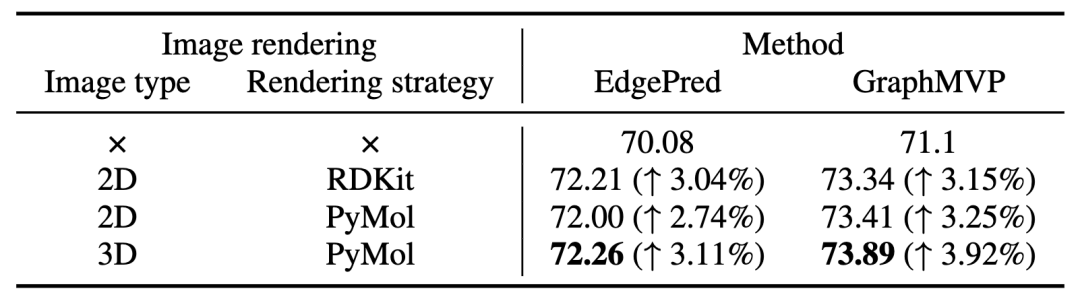
表 4
图像效率
为了验证IEM的图像效率,作者使用了不同数量的图像来增强GraphMVP。表5给出了不同图像数量下的平均ROC-AUC。发现随着图像样本数量的增加,IEM的性能从1.55% 逐渐提升到 3.92%。特别是,在仅使用5%图像数量的情况下,IEM仍然能够提高基线的性能,其性能相对提高1.55%,实验结果证明IEM对图像是高效的,可以在节省计算资源的条件下来保证性能的提升。

表 5
消融研究
如表6所示,展现了作者使用4种不同GNN对知识增强器和任务增强器进行消融研究的结果。根据实验结果显示,单个增强可以提升基线的性能,绝对提升幅度高达2.06%,可见两种增强的有效性。同时实验结果也显示,组合两种增强可以实现更多的性能提升,绝对性能提升幅度在2.62%到3.48%之间。此外,作者还注意到,KE的性能改进总是优于TE,这表明教师的基础先验知识(原子,化学键,几何和属性)可以很好地转移到学生身上,从而提升更多的性能。
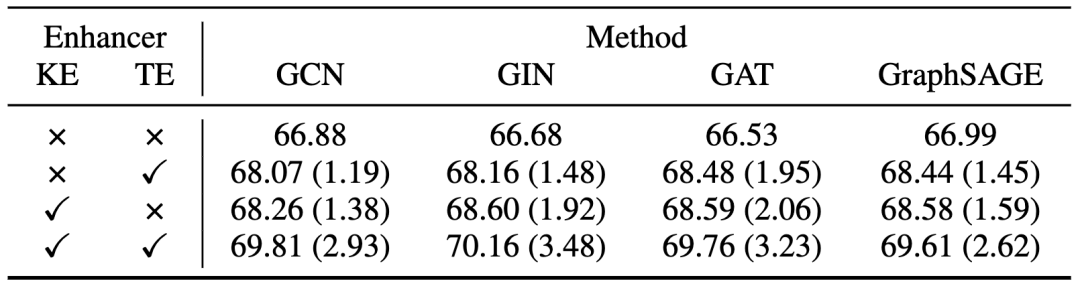
表 6
结论
在这项工作中,作者提出了一种新的图像增强分子图表征学习框架(称为IEM),这是第一次尝试使用图像来提高图的性能。配备了知识丰富的基于图像的教师和2个增强器(知识增强器和任务增强器),提出的IEM可以在大量药物发现基准上显著提高基于图的方法的性能,而无需进行任何架构修改。因此,IEM有潜力利用图像来支持更广泛的图表征学习领域,例如网格表示学习和骨架表示学习。特别是,通过实验证明,对于难以获得图像数据的微观实体,通过廉价的图像渲染可以提高性能,这将鼓励IEM用于生命科学中更多的生物实体,如蛋白质和核糖核酸。
参考资料
论文:https://www.ijcai.org/proceedings/2024/675
代码:https://github.com/HongxinXiang/IEM

