DRUGAI
今天为大家介绍的是来自武汉大学的田天和南京大学的张洁撰写的一篇论文。空间分辨转录组学(Spatially Resolved Transcriptomics, SRT)技术显著推动了生物医学研究的发展,但由于数据的离散性、高噪声水平以及复杂的空间依赖性,使得数据分析仍然充满挑战。在此,作者提出了一种名为spaVAE的感知依赖深度生成空间变分自编码器模型,该模型能够以概率方式表征计数数据,同时捕捉空间相关性。spaVAE引入了一种混合嵌入方法,将高斯过程先验与高斯先验结合,明确捕捉不同点位之间的空间相关性。然后,该模型通过优化深度神经网络的参数来逼近SRT数据的底层分布。利用这些逼近的分布,spaVAE可以完成多个SRT数据分析中的关键任务,包括降维、可视化、聚类、批次整合、去噪、差异表达、空间插值、分辨率增强和空间变异基因的识别。此外,作者还将spaVAE扩展到spaPeakVAE和spaMultiVAE,分别用于表征空间ATAC-seq数据和空间多组学数据。

SRT技术实现了基因表达的高通量分析,同时保留了空间信息,显著推动了多个生物医学研究领域的发展。尽管技术上与单细胞RNA测序(scRNA-seq)有差异,SRT测得的表达通常是离散的、过度分散且噪声较大。此外,SRT还测量了组织中细胞或点位的相对位置。如果能准确捕捉这些自然空间依赖性,将会提供极具价值的信息。现有的scRNA-seq数据分析方法在空间域检测方面可能效果不佳,因为它们未能利用宝贵的空间信息,简单地假设细胞或点位是独立的。
为了应对这些挑战,多种深度学习方法被提出来建模SRT数据的空间信息,但这些方法需要在模型训练前显式建立空间位置之间的依赖关系,可能会将偏差传递到后续的分析任务中。此外,一些统计方法也被提出用于SRT数据建模,但这些方法通常需要强假设,且在数据多样性和复杂性面前性能会下降。
具有空间依赖性感知能力的深度生成模型
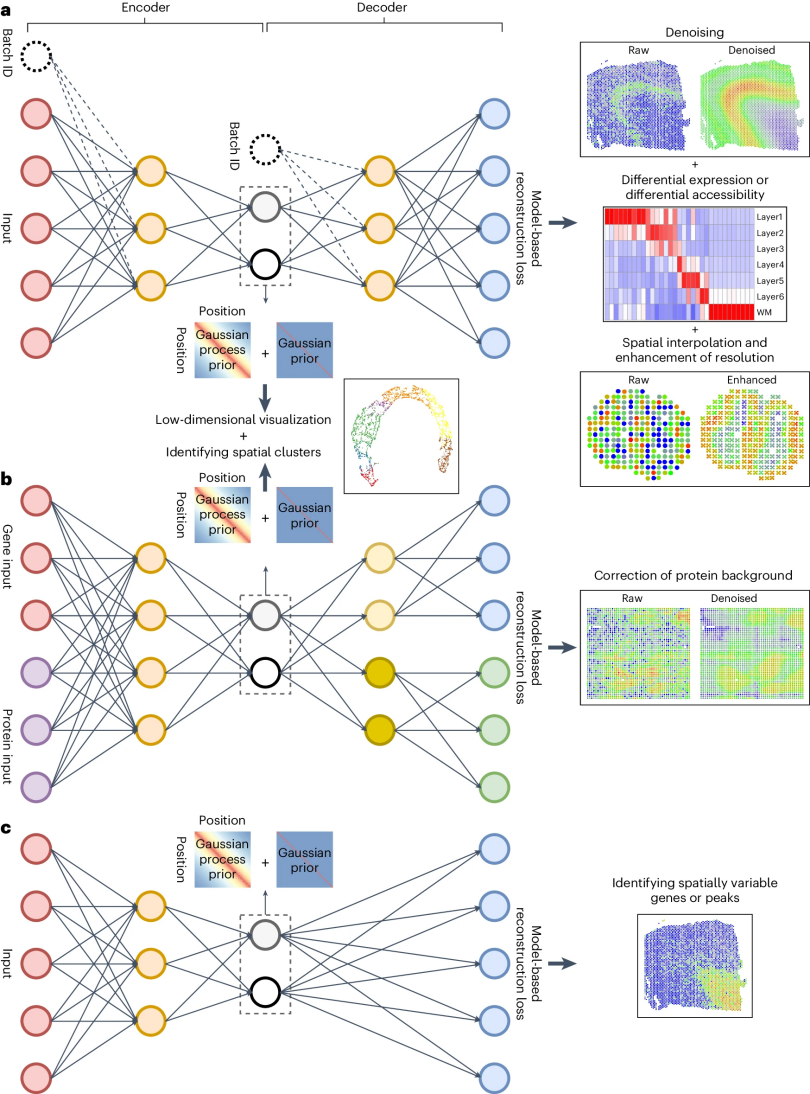
图 1
图1a展示了用于SRT数据的空间变分自编码器模型(spaVAE)和用于空间ATAC-seq数据的模型(spaPeakVAE)的示意图。spaVAE模型基于变分自编码器(VAE),以概率方式建模SRT数据。与典型的标准高斯先验VAE模型不同,作者通过引入高斯过程先验来建模空间依赖性。首先,瓶颈层学到的潜在表示可以用于可视化和聚类分析。其次,基于负二项式模型的解码器输出提供了观测到的SRT计数数据的底层分布估计,可用于去噪和差异表达分析。最后,由于其生成特性,高斯过程先验能够插值未观测位置的观测值,这一特性使得插值未观测点和提高空间分辨率成为可能,进而提供原始数据中不可检测的生物学洞察。理论上,高斯过程先验可以在任何合适的位置插值潜在表示,使模型能够灵活地在用户期望的分辨率下增强空间分辨率。
spaPeakVAE模型是spaVAE的扩展,专门用于分析空间ATAC-seq数据。空间ATAC-seq数据描绘了具有位置信息的染色质可接近性,观测数据非常稀疏且是二值的(即峰区是否可接近)。根据先前的研究,作者在spaPeakVAE中使用了伯努利模型解码器以适应ATAC-seq数据的二值特性。此外,spaPeakVAE还考虑了点位和峰的测序偏差。如图1b所示,作者还可以将深度生成模型扩展到空间多组学数据(spaMultiVAE)。在空间多组学数据中,基因和表面蛋白的表达被描绘,表面蛋白的强度通过条形码抗体测量。然而,这种技术会引入不同的技术偏差,如由于环境或非特异性抗体结合导致的不良背景。为了解决这个问题,作者引入了负二项式混合模型解码器来区分背景和前景蛋白信号,这一方法已在单细胞CITE-seq数据的totalVI分析中成功使用。spaPeakVAE和spaMultiVAE都与spaVAE共享相似的框架,因此spaVAE中适用的多种分析也可以使用这两种变体模型进行。
图1c展示了spaVAE和spaPeakVAE模型的变体,涉及空间线性解码器变分自编码器(即spaLDVAE和spaPeakLDVAE)。与spaVAE模型一样,该模型中的潜在嵌入也包含两部分:一部分遵循高斯过程先验,另一部分遵循高斯先验。该模型使用线性解码器,并设定其潜在嵌入和权重非负。spaLDVAE和spaPeakLDVAE可以看作非负因子分析模型。线性解码器的权重可用于衡量两种先验对重建的贡献,这使得基于高斯过程嵌入部分的贡献评分对SVGs或空间可变峰进行优先排序成为可能。
spaVAE用于对人类背外侧前额叶数据的多重分析
作者将spaVAE应用于LIBD人类背外侧前额叶皮层(DLPFC)数据集,以展示其在各种分析中的性能。该数据集测序了来自三个大脑的12个组织切片,覆盖了六个神经层和白质。首先,作者在12个样本上获取了spaVAE的低维嵌入,并进行了k-means聚类以识别空间域。作者提供了手动标注的层标签以评估嵌入质量和聚类性能。
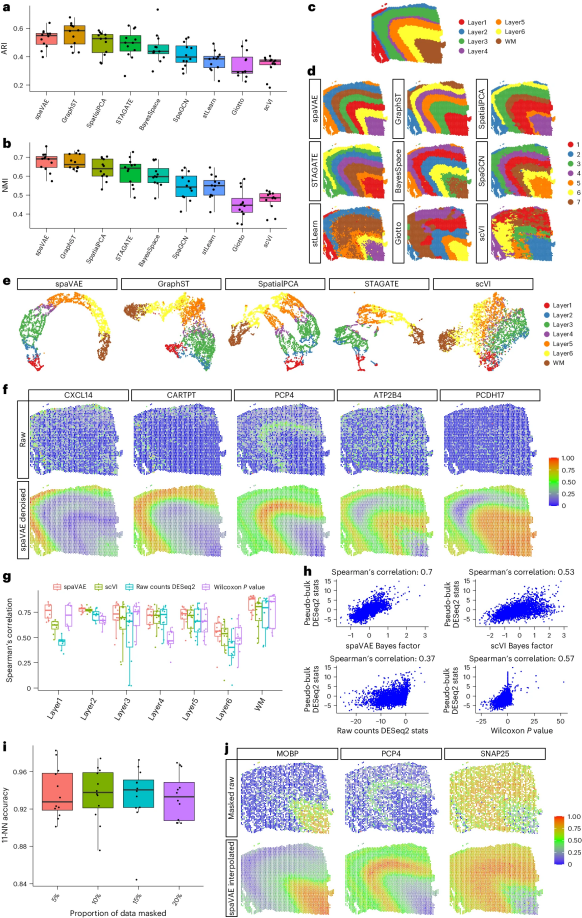
图 2
图2a和2b显示了用调整后的Rand指数(ARI)和归一化互信息(NMI)量化的聚类准确性。结果表明,spaVAE和GraphST在所有竞争方法中表现最佳,这其中包括专为SRT数据聚类分析设计的方法如STAGATE、BayesSpace、SpaGCN和Giotto。如图2c和2d所示,作者展示了DLPFC切片151673的各种方法的聚类结果。spaVAE推断的聚类标签具有与真实标签一致的清晰层次结构,而scVI的结果空间模式非常模糊。这些结果说明了在深度学习模型中整合位置信息以表征SRT数据的重要性。如图2e所示,作者展示了使用UMAP(统一流形近似和投影)对DLPFC切片151673的不同方法的潜在表示。
其次,作者评估了去噪和差异表达分析的性能。图2f展示了不同层标记基因的去噪计数。spaVAE去噪后,标记基因表达呈现清晰的层次结构,而原始计数则噪声较大且空间模式不明显。图2g显示了差异表达分析的结果。作者首先汇总了12个切片中相同神经层的计数,并构建了伪批量RNA-seq数据。然后,作者使用DESeq2软件包对伪批量RNA-seq数据进行差异表达分析。随后根据Lopez等人的方法,将DESeq2的Wald统计量作为伪批量RNA-seq数据的真实差异表达结果。接下来,作者计算了不同方法推断的每个组织切片计数的差异表达统计量与所有12个切片层间真实差异表达统计量之间的Spearman相关性。值得注意的是,spaVAE生成的贝叶斯因子显著优于其他方法(如图2h所示)。这一结果强调了结合空间信息的重要性,以及spaVAE利用整个数据集进行推断的能力。
最后,作者展示了高斯过程先验的空间插值功能,图2i展示了遮掩实验的结果。作者遮掩了不同比例(5-20%)的数据,并使用剩余点位作为训练数据。训练spaVAE模型后,作者使用这些点位的空间位置插值遮掩点位的潜在表示。作者使用基于训练点位的11最近邻(11-NN)预测器预测遮掩点位的标签,并评估其准确性。结果显示12个切片的准确性非常高,并且在遮掩位置比例增加的情况下具有鲁棒性(>90%)。这表明高斯过程先验可以进行精确的插值分析。图2j展示了DLPFC切片151673中遮掩20%点位时层标记基因MOBP、PCP4和SNAP25的插值计数,该图表明插值可以恢复基因表达的潜在空间模式。
复杂空间转录组学数据的spaVAE
鼠海马体Slide-seq V2数据集包含超过50,000个点位和20,000个基因。作者筛选了基因,并选择了前3,000个空间变异基因(SVG)进行分析。作者在该数据集上训练了spaVAE模型,并对学到的嵌入进行了k-means聚类。
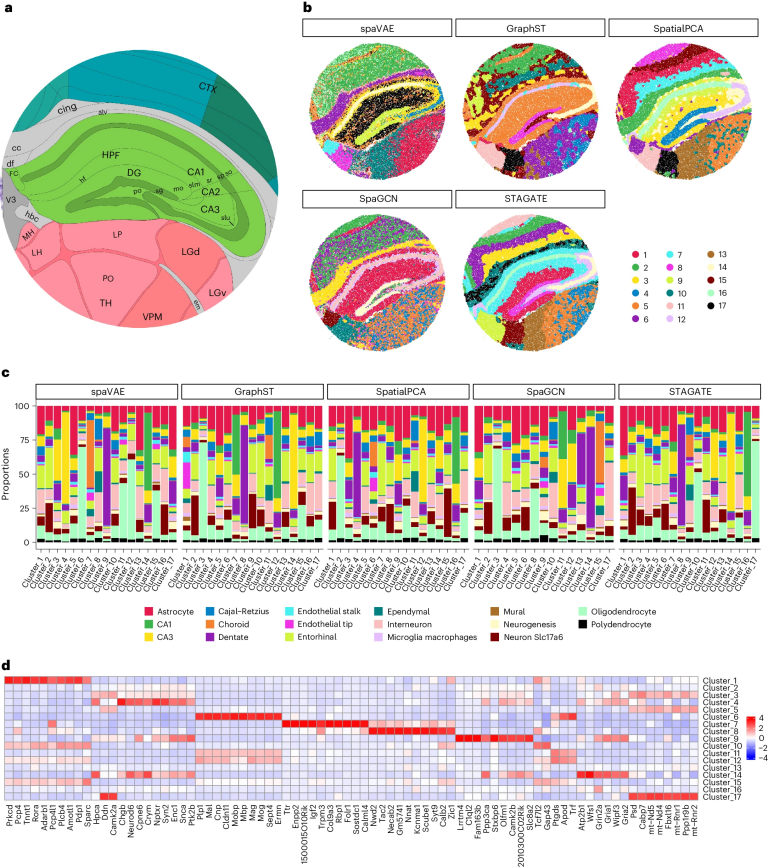
图 3
图3a显示了使用Allen Brain Atlas注释的鼠海马体结构。在图3b中,作者比较了spaVAE与其他能够处理大型数据集的方法的聚类结果。spaVAE预测的聚类标签识别了大多数空间域,包括皮质层(clusters 2, 16)、胼胝体(cluster 6)、CA1(cornu ammonis 1; cluster 14)、CA3(cluster 4)、齿状回(cluster 9)、海马区域(clusters 3, 5, 17)、丘脑区域(clusters 1, 8, 10)和第三脑室(cluster 7)。相比之下,GraphST和STAGATE未能识别出第三脑室,而SpaGCN未能正确识别CA1和CA3。接下来,作者应用RCTD(鲁棒细胞类型分解)对数据集进行分析,分解每个点位的细胞类型比例。如图3c所示,spaVAE的聚类结果产生了高纯度的细胞类型(JS散度
使用spaLDVAE识别空间变异基因
识别空间变异基因(SVGs)是SRT数据分析中的关键步骤。作者在实验中使用spaLDVAE识别SVGs,采用空间位置随机置换法确定显著性阈值。首先,作者在模拟数据集上评估了spaVAE,结果表明其优于SpatialDE和SPARK,并在合理水平上控制了误发现率。接着,作者将spaLDVAE应用于人类DLPFC数据集,观察到SVGs的明确空间模式。作者还应用spaLDVAE于鼠海马体Slide-seq V2数据集,观察到了清晰的空间基因表达模式。
使用spaVAE提升空间转录组学数据的分辨率
目前基于下一代测序的空间转录组学(SRT)数据无法提供单细胞分辨率的基因表达谱。由于这一技术限制,点位的大小通常大于单个细胞的大小。例如,在10X Visium平台上,一个点位通常包含几十个细胞。因此,需要计算模型来提高空间分辨率。为此,作者将spaVAE应用于两个SRT数据集:小鼠嗅球数据和HER2乳腺肿瘤数据。
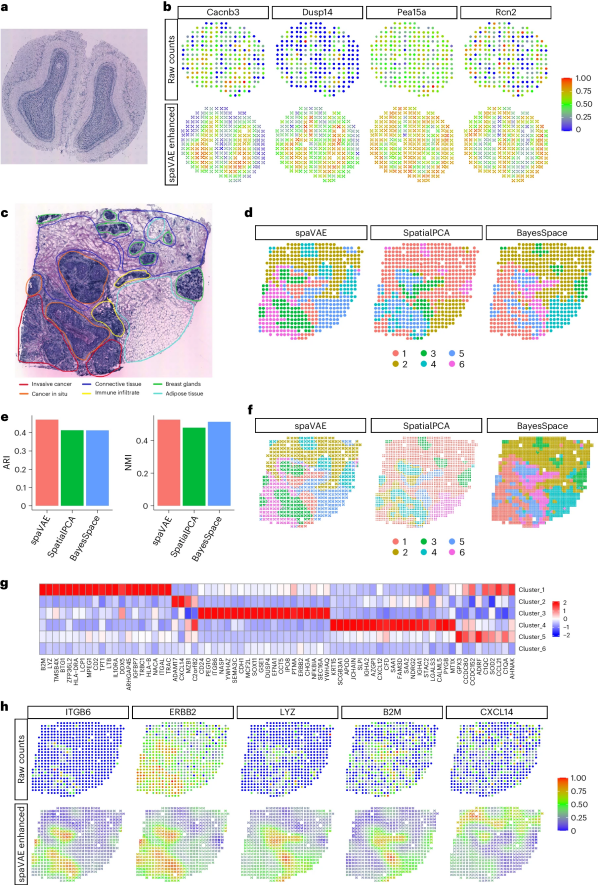
图 4
图4a展示了小鼠嗅球数据集12个样本之一的H&E染色图像。图4b展示了一些标记基因的原始计数和spaVAE增强的计数。对于每个点位,作者通过spaVAE将分辨率提高到五个子点位。如图所示,原始计数中的空间模式模糊,但经过空间增强后可以看到与H&E图像一致的清晰模式。
在HER2乳腺肿瘤数据中,Andersson等人提供了病理学家注释的组织标签(图4c)。作者在数据集上训练了spaVAE,并对潜在表示进行了k-means聚类。图4d展示了spaVAE和其他基线方法的聚类结果,图4e量化了其聚类性能,spaVAE的聚类结果优于SpatialPCA和BayesSpace。图4f展示了空间增强后的结果,增强后spaVAE产生了更复杂的空间模式。通过使用spaVAE推断的贝叶斯因子和对数折叠变化量,作者检测到了簇中的顶级上调基因(图4g)。图4h比较了这些标记基因的原始计数和spaVAE增强的计数。作者得出结论,spaVAE的空间增强功能可以显著改善这些标记基因的空间模式。
spaPeakVAE用于空间双模态ATAC-seq数据
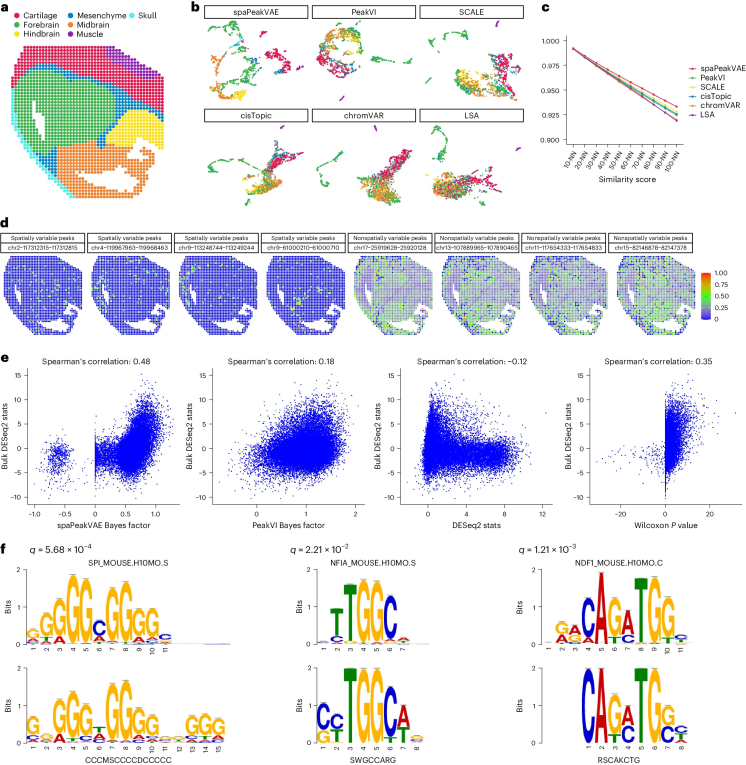
图 5
作者将spaPeakVAE应用于小鼠胚胎脑的空间双模态ATAC-seq数据集,MISAR-seq测量了小鼠胚胎脑不同区域的ATAC和信使RNA计数(图5a)。首先,作者训练了spaVAE模型并获得了mRNA数据的潜在表示。接下来,作者通过与多个用于ATAC数据的计算模型的比较,评估了spaPeakVAE的性能。结果显示spaPeakVAE的潜在表示能够很好地分离不同的标签,而在竞争方法的嵌入中,前脑、中脑和后脑总是纠缠在一起(图5b)。作者量化了不同方法对ATAC数据的嵌入与spaVAE对mRNA数据的嵌入之间的相似性,使用了一系列k-NN相似性评分(图5c)。作者发现spaPeakVAE的嵌入具有最佳的相似性评分,表明spaPeakVAE的嵌入与mRNA嵌入最为接近。图5d显示了spaPeakLDVAE为ATAC数据识别的顶级空间和非空间可变峰,作者可以在顶级空间峰中看到明显的空间模式。作者还进行了前脑对比中脑的差异可及性分析,并将差异可及性结果与对应的批量ATAC-seq数据进行比较。同样,作者观察到spaPeakVAE的差异可及性结果显著优于使用PeakVI、DESeq2和Wilcoxon符号秩检验的差异可及性分析(图5e)。为了进一步验证spaPeakVAE的差异可及性分析,作者使用基于网络的工具MEME-ChIP分析了由spaPeakVAE推断出的显著峰区域,以识别富集的基序。图5f显示了从这些显著峰中识别的基序及其与已知小鼠基序的显著匹配。基于这些分析,spaPeakVAE可以有效地表征空间ATAC-seq数据。
spaMultiVAE用于空间多组学数据
近期测序技术的进步使得在同一个细胞或点位上同时测量基因表达和表面蛋白强度成为可能。与mRNA测序不同,蛋白质分析需要条形码标记的抗体与特定蛋白质结合。然而,这种结合并不总是特异的,可能导致不利的背景强度,需要被消除。为此,作者设计了spaMultiVAE,用于建模背景和前景蛋白强度的混合。
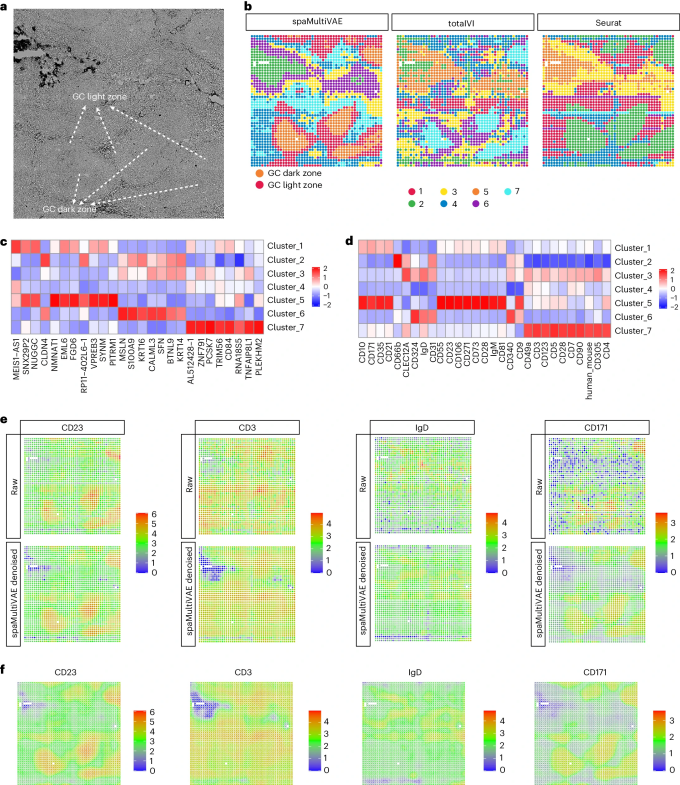
图 6
图6a、b显示了spaMultiVAE的聚类结果具有更平滑的空间模式,并且与组织图像的吻合度优于totalVI和Seurat。作者使用spaMultiVAE对基因和蛋白质进行了差异表达分析,以识别每个簇中的顶级基因和蛋白质(图6c, d)。如预期所示,簇1和簇5是生发中心的光区和暗区,在簇5中观察到了生发中心B细胞中包括CD23和IgM在内的上调标记蛋白。图6e显示了原始和spaMultiVAE去噪后的蛋白质强度。可以观察到,spaMultiVAE不仅校正了蛋白质强度中的背景噪声,还改善了空间模式。图6f展示了spaMultiVAE对蛋白质的空间分辨率增强。综上所述,spaMultiVAE能够高效地阐明空间结构并消除空间多组学数据中的不良背景噪声。
讨论
spaVAE、spaPeakVAE和spaMultiVAE是考虑空间依赖性的深度生成模型,能够从数据中自动优化依赖强度,并通过基于模型的损失函数直接表征离散计数数据。基于来自各种平台和组织的多种类型空间组学数据(如空间转录组学、空间ATAC-seq、空间多组学),这些模型在可视化、聚类、差异表达和可及性、去噪、批次整合、空间插值和分辨率增强等任务中表现出色。尽管在每个任务中可能不是最佳,但提供了利用空间信息的统一框架,具有良好的性能和潜力。作者还引入了spaLDVAE和spaPeakLDVAE,用于检测空间变异基因和空间变异峰。spaVAE和spaMultiVAE直接输入离散计数数据并消除技术偏差,而spaPeakVAE则直接处理二值输入并考虑测序偏差。除此之外,通过应用稀疏高斯过程回归技术,加速了高斯过程协方差矩阵的计算。
编译 | 于洲
审稿 | 曾全晨
参考资料
Tian T, Zhang J, Lin X, et al. Dependency-aware deep generative models for multitasking analysis of spatial omics data[J]. Nature Methods, 2024: 1-13.

