DRUGAI
近日,Cell出版社旗下Trends in Biotechnology 杂志在线发表了河南农业大学农学院陈震与姬祥课题组联合撰写题为“AI sheds new light on genome editing”的综述论文。该研究系统综述了深度学习技术在基因组编辑领域中的应用与发展。

目前,人工智能,尤其是深度学习技术,极大地革新了生物数据的获取与分析方式,推动了生物技术、医学和农业等多个领域的突破。数十年来,"序列-结构-功能"范式始终指导着蛋白质研究。如今,深度学习正通过蛋白质表征学习重塑这一范式-其不仅能破译复杂生物模式,更实现了AI驱动的蛋白质设计与改造(图1)。以CRISPR/Cas系统为主的基因组编辑技术已在基础研究、农业育种和生物医药等领域展现出巨大的应用潜力,然而如何高效发现、设计与改造新型基因组编辑工具仍是难以解决的领域瓶颈。
本研究系统综述了人工智能与基因组编辑交叉领域中的前沿进展,首先详细阐述了深度学习技术在基因组编辑工具的挖掘、改造与设计中的应用(表1)并探讨了其优缺点,并讨论了领域内面临的诸多挑战(包括技术局限性、可解释性不足以及潜在的伦理风险)。人工智能与基因组编辑领域的结合正引领生物技术迈向新的台阶,加速生物学、生物医学与生物技术的新一轮变革。
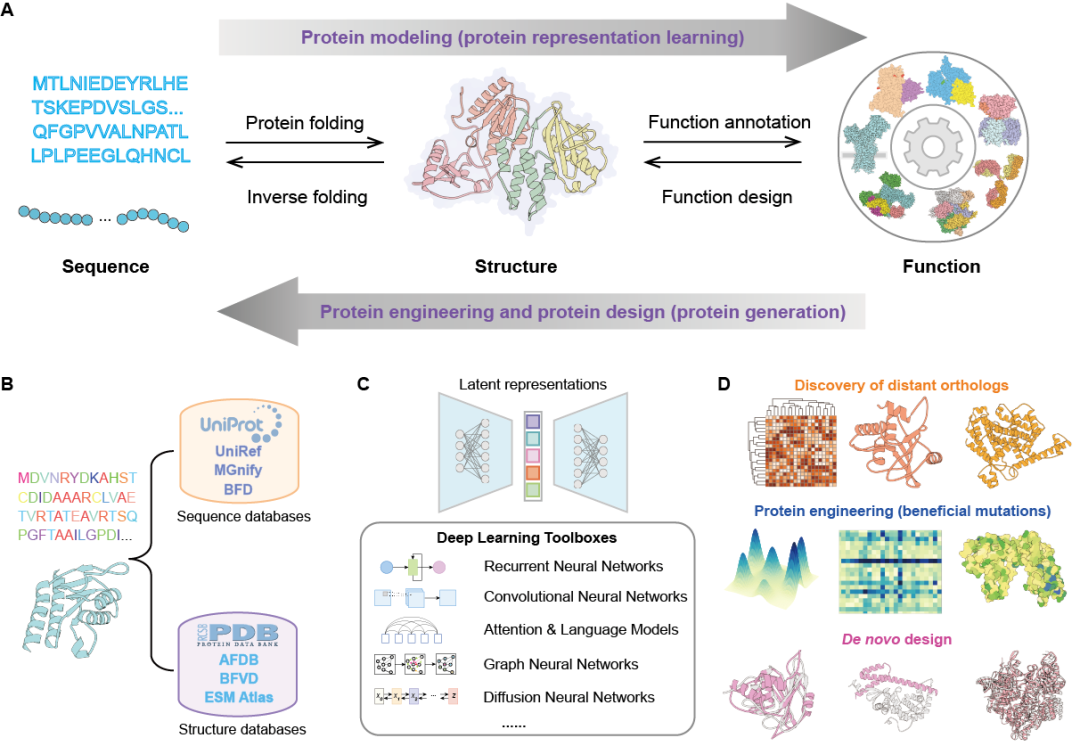
图1 深度学习在基因组编辑蛋白中的应用
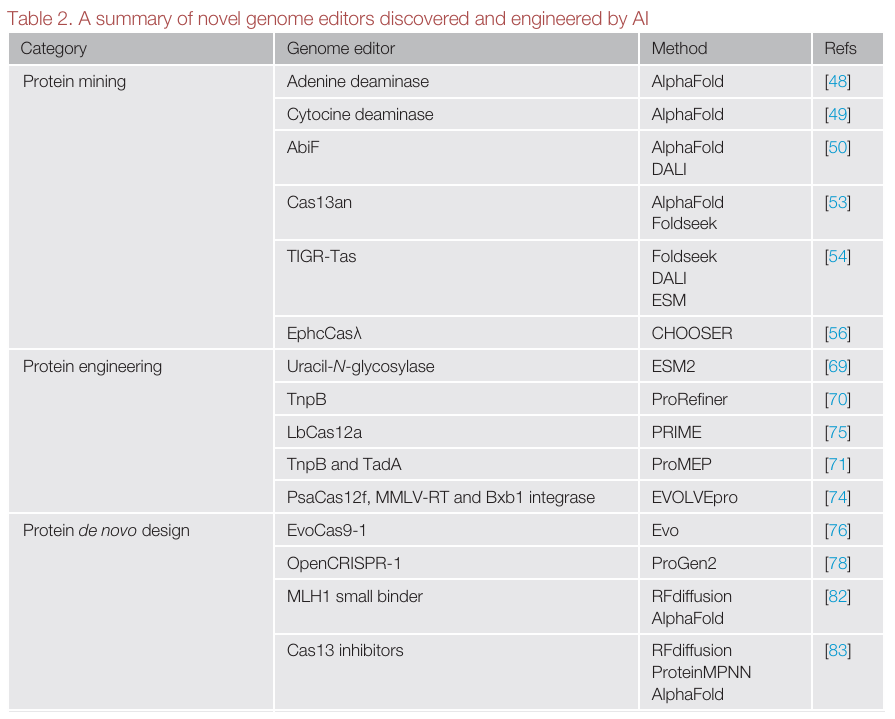
表1 人工智能发现和改造的新型基因编辑工具汇总
AI助力新型基因编辑器的发现突破了传统序列比对的限制。传统序列比对工具如BLAST和HMMER在识别远缘同源蛋白时因序列差异大而效果受限,而深度学习结合“序列–结构–功能”范式,通过蛋白质三维结构的预测和分析,大幅提升了识别能力。随着AlphaFold DB等高质量结构数据库的开放,科学家们开展了大规模结构聚类和结构同源搜索,成功挖掘出多种新型Cas蛋白,包括新型碱基编辑蛋白、RNA引导的Cas13家族蛋白、TIGR-Tas和EphcCasλ等。
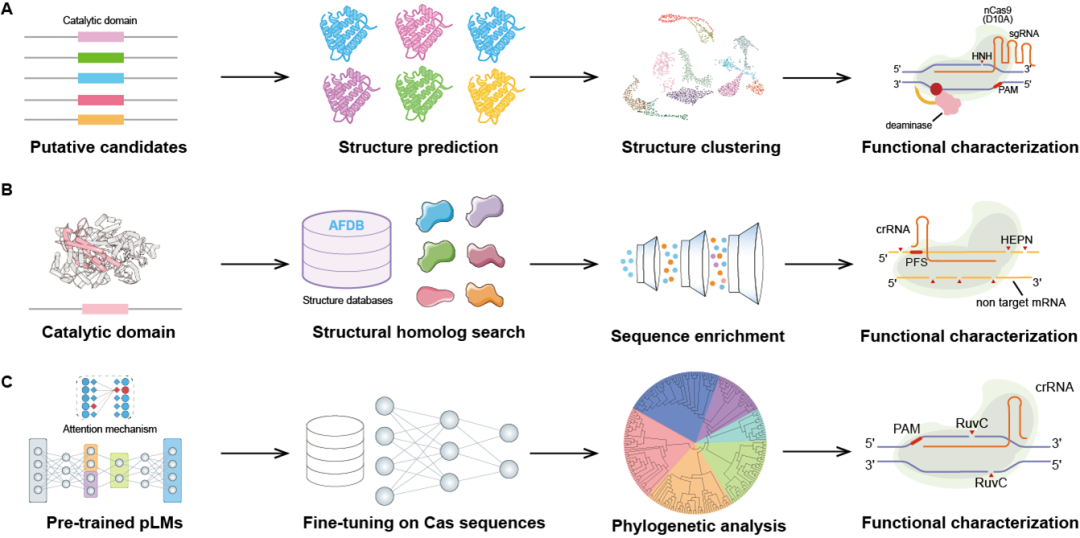
图2 人工智能辅助的基因编辑器深度挖掘
AI蛋白质工程方法实现了基因编辑蛋白的快速in silico进化。传统的高通量突变筛选和理性设计耗时费力,机器学习辅助的定向进化(MLDE)和零样本(Zero-shot)蛋白设计为这一难题提供了新的解决思路:即在实验数据不足时,直接用深度学习模型预测有益突变,大大降低了对实验筛选数据的依赖。零样本方法利用预训练模型预测有益突变,快速锁定潜在突变位点,而少样本方法结合少量实验数据与主动学习策略,可以显著提高突变的成功率。例如,结合结构信息的多模态深度模型ProMEP,进化出了效率提升多倍的TnpB和TadA变体,性能甚至超越实验室进化版ABE8e。
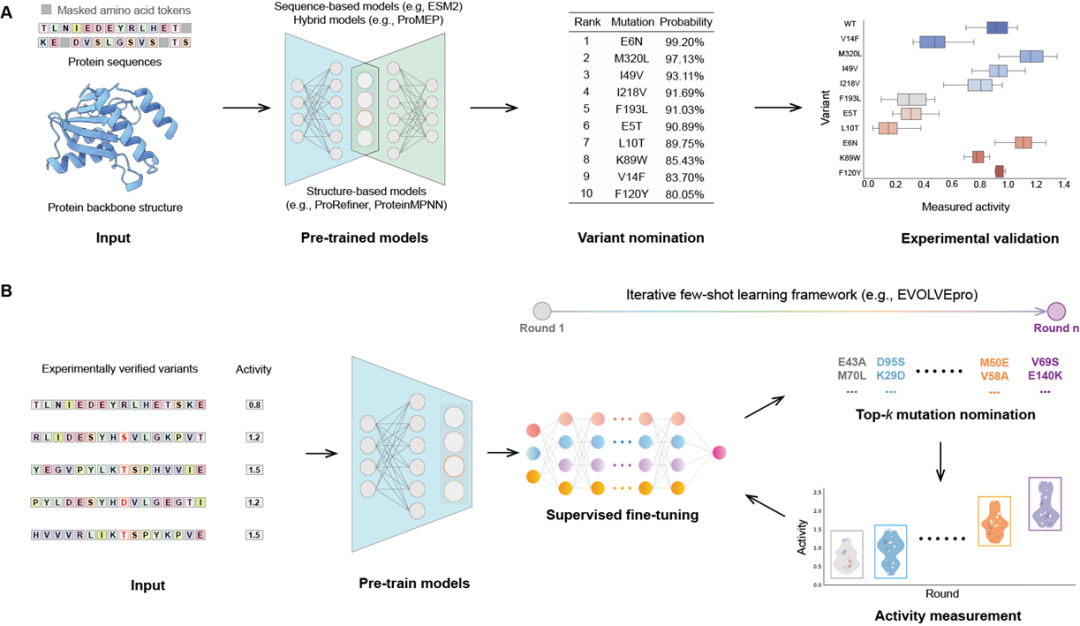
图3 基因编辑工具的高效改造
深度生成式AI模型为基因编辑工具的从头设计开辟了新路径。大语言模型通过学习大量序列数据,能够设计出自然界尚不存在的全新基因编辑器,如Open-CRISPR-1和EvoCas9-1。扩散模型更能生成符合功能需求的三维蛋白结构,研究者们结合RFdiffusion、ProteinMPNN和AlphaFold,成功开发出高效的PE7-SB,在人类细胞中将引导编辑效率提高了近30倍。虽然目前多数设计案例仍处于初步验证阶段,但生成式AI展示了设计复杂“分子机器”的巨大潜力,为基因编辑技术的未来发展奠定了坚实基础。
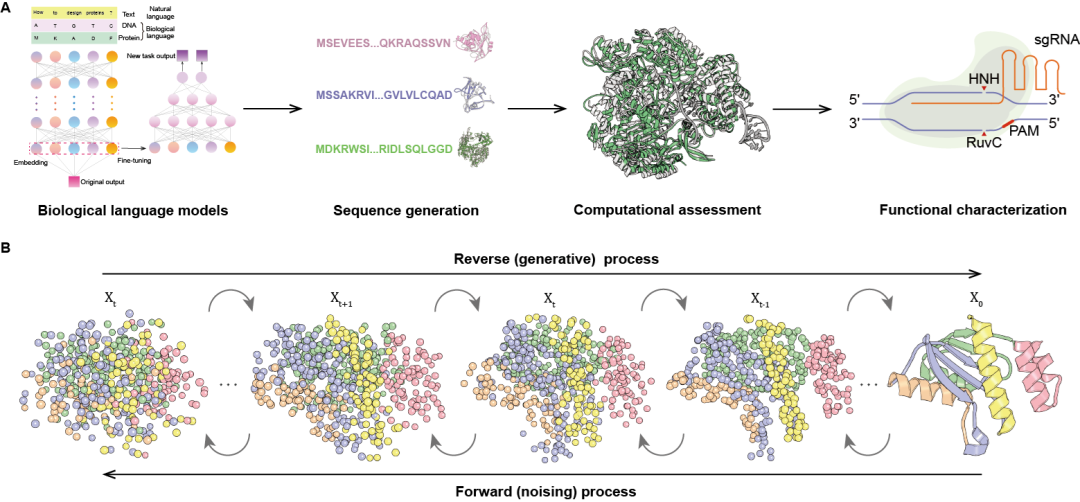
图4 基因编辑工具的从头设计
AI与生物技术的深度交叉无疑为科学研究和实际应用带来了前所未有的机遇。随着AI技术的持续发展,未来有望发现和设计出更多样化的基因编辑工具,深化对CRISPR进化机制的理解,推动基因编辑技术不断升级。
河南农业大学/小麦玉米两熟高效全国重点实验室25级博士生秦兆辉和邓兆龙为共同第一作者,陈震和姬祥教授为共同通讯作者,李成伟教授和王道文研究员为共同作者。该研究获得了国家自然科学基金、国家重点研发计划和河南省自然科学基金等项目的资助。
参考资料
Qin, Z., Deng, Z., Li, C., Wang, D., Ji, X., & Chen, Z. (2025). AI sheds new light on genome editing. Trends in Biotechnology.
内容中包含的图片若涉及版权问题,请及时与我们联系删除

