大家好,今天给大家分享一篇近期发表在Macromolecules上的研究进展,题为:Sequence-Based Computational Design of High-Affinity Amphiphilic Copolymers for Protein Targeting: A Machine Learningand Coarse Grained Simulation Approach。该工作的通讯作者是来自南京理工大学的贾旭教授和徐枭副教授。
蛋白亲和试剂(PARs)是一类用于特异性识别和结合蛋白质的分子,其早期研究源于单克隆抗体的开发,但由于天然抗体面临生产成本高、稳定性差等问题,促使研究者转向开发合成聚合物作为替代品。尽管这些合成聚合物在结构互补性方面的研究取得了一定进展,但对化学组成互补性的研究仍较薄弱,序列控制困难限制了其结合性能的进一步提升。随着序列可控聚合物合成技术的发展,以及计算模拟和机器学习方法的引入,研究者开始探索通过高效建模和数据驱动策略来设计高亲和力、低成本的合成聚合物类 PARs,以实现对蛋白质更精确的靶向和识别。
本文开发了一种新颖的高通量模拟流程,结合基于机器学习的数据增强方法,从大量候选序列中识别出与靶蛋白具有良好相互作用的关键聚合物序列。生成了3500多种聚合物的结合亲和力数据,构成了用于训练和测试 ML 模型的充足数据库,ML 模型随后被用来进一步扩展“序列–性能”关系。作者预测了超过30,000种聚合物的性质,从中识别出具有最高结合亲和力的聚合物,从而筛选出潜在的 PAR 候选分子。最后,通过更高精度的计算方法对 ML 模型的预测结果进行了验证。
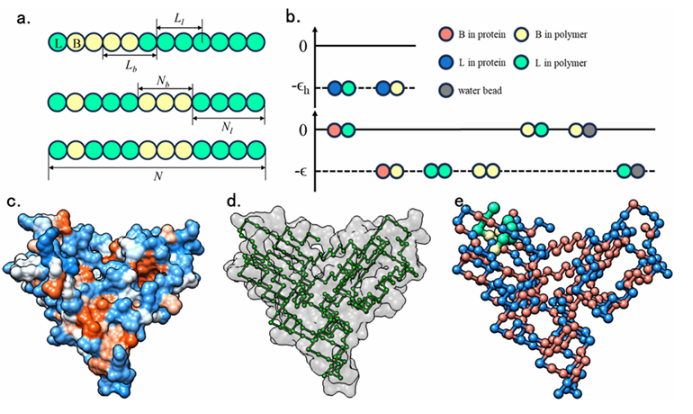
图1. 晶格模拟示意图
首先,作者构建了一个简化的粗粒度晶格模型来提高计算效率,采用上皮细胞黏附分子(EpCAM)作为模型蛋白,两性聚合物和蛋白单体单元更分为亲水(L)和疏水(B)两类。通过接触能量定义其与蛋白之间的相互作用,并以晶格方式近似表示蛋白质的疏水性表面。(图1)
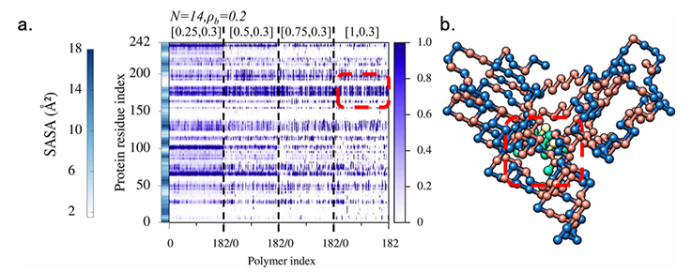
图2. 蛋白质上的共聚物结合位点
考虑到构象的复杂性,作者聚焦于聚合度为12和14的短链共聚物。根据模拟轨迹计算了共聚物与每个蛋白质“珠”的接触情况。热图的强度表示每个蛋白“珠”与共聚物接触的概率,可作为亲和力的指标。作者分析了不同相互作用参数条件下共聚物与蛋白质的结合位置,并发现多数共聚物趋向于与蛋白表面一个共同区域结合,即一个疏水结合口袋。该区域的识别具有高度一致性,且与蛋白的溶剂可接触面积(SASA)密切相关,说明共聚物确实能够在粗粒度尺度上识别蛋白的关键结构特征。(图2)
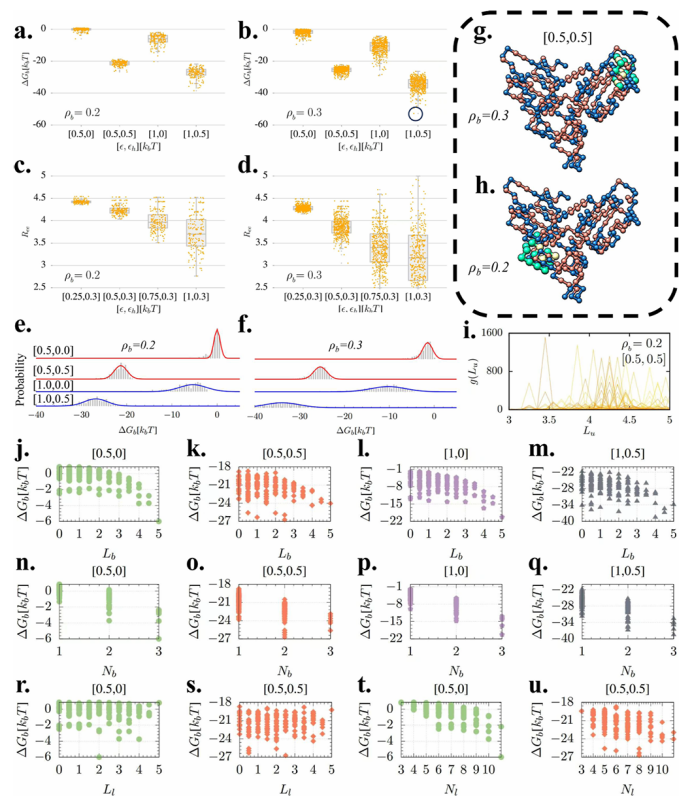
图3. 晶格模拟结果
随后,作者探讨了共聚物序列结构对结合自由能(ΔGb)的影响。通过统计模拟结果,作者发现疏水片段的长度和在链上的位置对ΔGb有显著影响。特别是当疏水片段集中分布于链的一端时,或者是长且连续的疏水序列,通常会获得更低的结合自由能,这种序列设计有助于增强蛋白识别能力。(图3)
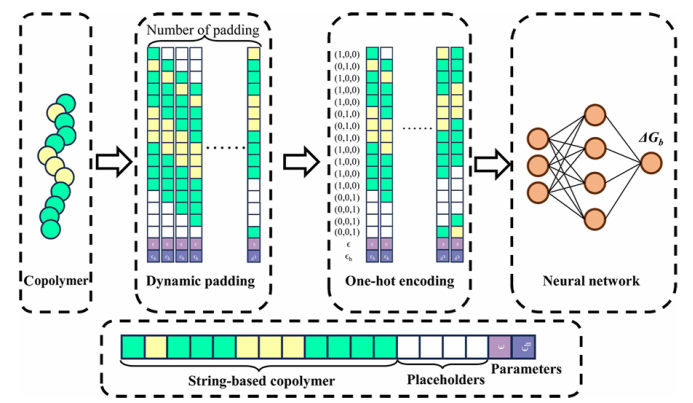
图4. 共聚物特征化与神经网络架构
为了进一步提升筛选效率,作者设计了一个神经网络架构,通过独热编码与动态填充等策略将共聚物序列转化为可用于机器学习模型的向量表示。该神经网络模型能够输入任意给定序列,输出其预测的ΔGb。 (图4)
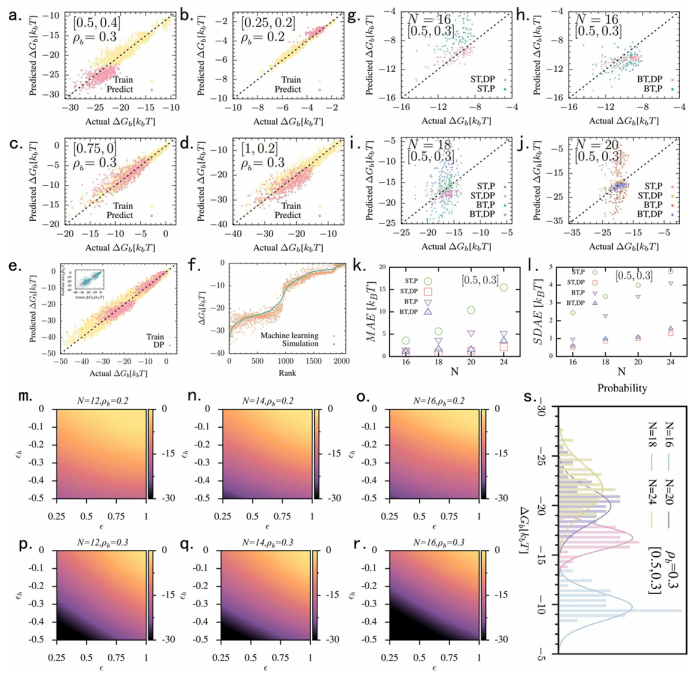
图5. 神经网络模型预测性能表现
接下来,作者详细评估了该神经网络模型的性能。作者设置了不同训练集策略,包括小训练集ST和大训练集BT,以及不同编码策略,包括动态填充DP与普通填充P。ST训练数据限于与预测共聚物相同相互作用参数的共聚物;BT则包含所有模拟共聚物,并在共聚物描述符中加入一个双位向量表示相互作用参数。作者发现动态填充法效果更好,而BT 集并未显著改善模型性能,可能是混合不同参数下的短链共聚物反而可能稀释了特定特征的表达,导致在长链预测中仍存在较大误差。此外,作者通过将模型应用于超过2000个新序列的亲和力预测并排序,尽管存在一定离散性,预测值与模拟数据总体一致。(图 5)
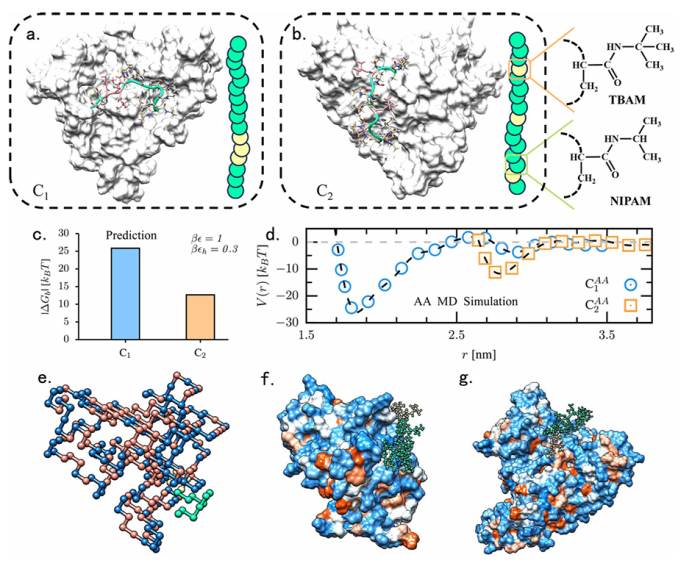
图6. 全原子分子动力学模拟验证神经网络预测
最后,作者对神经网络模型预测的最优(C1)和最差(C2)的结合候选物进行全原子分子动力学模拟。将 L 和 B 单体分别替换为叔丁基丙烯酰胺(TBAm)和N-异丙基丙烯酰胺( NIPAm) 单体,这两种单体常用于热响应性共聚物。结果显示,模型预测结果与此模拟所得的结合构象与ΔGb趋势一致,验证了神经网络输出的合理性。同时,作者还将共聚物与另一种蛋白(HSA)结合,发现其结合模式也基本延续原有规律,进一步证明该策略在不同目标蛋白上具有一定的泛化能力。(图 6)
综上,作者提出了一种结合晶格模拟与机器学习的高效计算范式,用于筛选针对目标蛋白的序列特异性结合聚合物。这一方法加深了对聚合物与蛋白结合模式的理解,并有望为下一代聚合物抗体的设计提供理论支持。
作者:ZXY 审校:ZHR
DOI: 10.1021/acs.macromol.5c01112
Link: https://doi.org/10.1021/acs.macromol.5c01112
内容中包含的图片若涉及版权问题,请及时与我们联系删除

