DRUGAI
本文介绍一篇来自深圳理工大学的冯志炜教授和薛颖教授、浙江大学侯廷军教授、浙江工业大学陈艳教授及匹兹堡大学的李威教授联合发表的研究。该研究提出了 AntiBMPNN,一种基于结构引导的图神经网络方法,能够在抗体工程中进行精确的序列设计并提升结合亲和力。AntiBMPNN 基于细化后的 message-passing neural network(MPNN)架构,结合抗体专属的三维结构数据,并使用频率驱动的打分函数与 AlphaFold 3 相协作,实现序列设计与结构恢复。通过该方法,模型在 perplexity 指标上达到了约 1.5,序列恢复率超过 80%,显著优于 ProteinMPNN方法。此外,研究还提出了一种频率基础的排序函数,用于评估候选序列的结构保真度、位置稳定性及生化特性,并通过 AlphaFold 3 验证结构重建品质。这种创新设置使得 AntiBMPNN 在单点突变设计中实验验证成功率高达 75%。在具体任务中,AntiBMPNN 相较于 AbMPNN、AntiFold 和 ProteinMPNN 在 CDR1–3 区域的设计表现优异,表明 AntiBMPNN 设计的序列性能显著超过原始序列和现有方法,突出了其在治疗性抗体设计中的实用潜力。

研究背景
在现代医学领域,抗体已成为至关重要的治疗工具,在癌症、自身免疫疾病和传染病等广泛疾病领域表现出显著疗效。传统抗体序列设计主要依赖实验方法,过程繁琐且耗时费力。近年来,计算抗体设计方法成为优化抗体功能与特异性的有效途径。该方法主要聚焦于对抗体结构中的关键氨基酸进行定向改造,而非进行大规模的序列改变。一种常见策略是在现有抗体结构中引入特定氨基酸,以此增强抗体与抗原的亲和力及选择性。另一种策略则是通过探索不同互补决定区(CDR)环的构象与序列,提升抗体识别抗原的能力。此外,以Rosetta为代表的序列设计算法,能够预测抗体与抗原之间的结构及能量相互作用,进而优化关键氨基酸,显著提高抗体与靶标表位的结合能力。
随着深度学习技术的蓬勃发展,ProteinMPNN等新型工具在蛋白质结构预测和优化领域取得重大突破。然而,ProteinMPNN在抗体超变区序列预测的准确率仅约为40%,这表明抗体设计领域仍面临诸多挑战。为克服这些难题,专门针对抗体结构的AbMPNN模型通过精细优化的训练,在超变区序列恢复率和结构聚类方面表现突出。AntiFold工具则利用反向折叠方法和抗原信息,大幅提升了抗体序列恢复与亲和力预测的准确性。尽管如此,这些方法目前缺乏充分的实验验证。而IgDesign作为首个经实验验证的抗体逆向折叠模型,成功设计了针对多个治疗靶点的抗体序列,并通过实验验证表现优于传统方法。然而,其代码未公开,这在一定程度上限制了更广泛的评估。最新提出的RFantibody流水线整合了多个先进工具,实现了抗体的从头设计,然而,设计过程的复杂性以及高昂的验证成本,仍是亟待解决的突出问题。
方法概述
本研究提出了名为AntiBMPNN的深度学习框架,专门用于优化抗体序列设计。它通过利用特制的抗体3D数据集、经过精细调整的消息传递神经网络(MPNN)、基于频率的评分函数以及AlphaFold 3,从而实现了高精度的抗体序列设计(图1)。与ProteinMPNN相比,AntiBMPNN表现出显著优势,体现为更低的困惑度(1.5)和更高的序列恢复率(≥80%)。实验结果显示,AntiBMPNN在单点抗体设计中成功率高达75%。在大多数情形下,AntiBMPNN的表现优于现有方法,包括AbMPNN、AntiFold以及ProteinMPNN,在优化互补决定区(CDRs)的CDR1、CDR2和CDR3序列时均表现出更好的结合亲和力。在NanoJ3体系中,AntiBMPNN对CDR1区域进行优化后,获得的EC50为9.2 nM,明显优于ProteinMPNN的135.2 nM和AntiFold的59.3 nM,与AbMPNN(EC50为6.6 nM)表现相当。在D6抗体的CDR2区域,AntiBMPNN取得了0.3 nM的EC50,表现明显优于AbMPNN(2.3 nM)、AntiFold(0.7 nM)和ProteinMPNN(0.7 nM)。对于J3的CDR3区域,AntiBMPNN实现了1.7 nM的EC50值,明显优于AbMPNN(51.2 nM)、AntiFold和ProteinMPNN(后两者均未检测到明显的结合能力)。以上结果充分展现了AntiBMPNN在设计高亲和力抗体序列上的突出能力,表明该方法在抗体序列优化中具有重要的实用价值。
结果与讨论
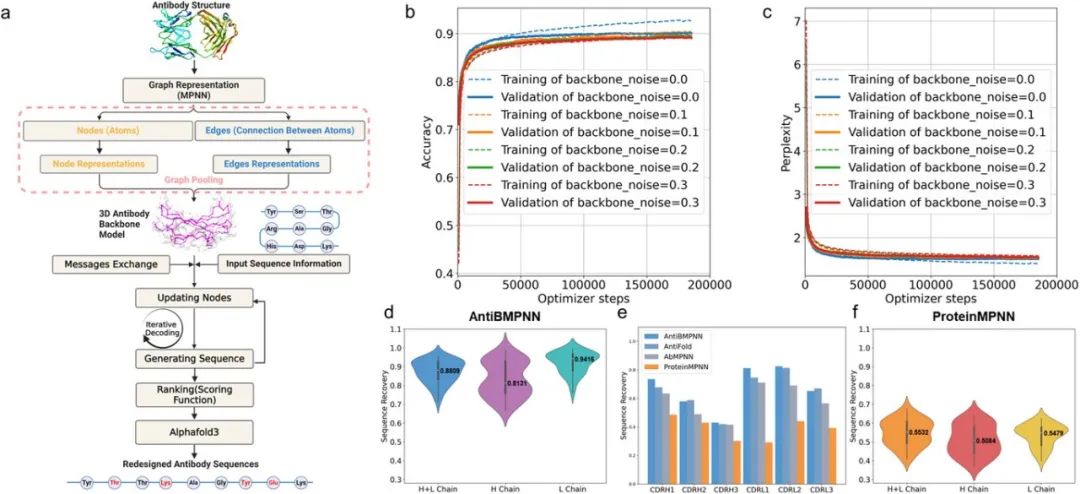
图1. AntiBMPNN算法框架和性能表现。
AntiBMPNN的训练性能
在6578 个抗体结构的训练数据上比较不同骨架噪声水平时,AntiBMPNN 在无噪声(0 级)条件下取得最高准确率和最低困惑度。当噪声进一步增大到 0.4-1 时,模型准确率持续下降,显示出对数据噪声的敏感性,强调了预处理和质量控制对抗体结构预测的重要性。在另一项包含 120 个 3D 结构(H+L 链、H 链、L 链)的综合测试中,AntiBMPNN 的中位序列恢复率分别为 88.09%、81.31% 和 94.16%,显著优于 ProteinMPNN(分别为 55.32%、50.84%、54.79%)。此外,AntiBMPNN 的序列恢复率与残基埋藏度呈正相关:深埋核心可达 93%-97%,表面区域为 85%-91%。针对六条 CDR 环(CDRH1、CDRH2、CDRH3、CDRL1、CDRL2、CDRL3)的比较显示,AntiBMPNN 在 CDRH1、CDRH3、CDRL1 和 CDRL2 四条环上取得最高恢复率(0.7358、0.5800、0.8129、0.8259);AntiFold 则在 CDRH2 和 CDRL3 略占优势。总体来看,所有模型在轻链环上的恢复率普遍高于重链环,而 CDRH3 仍是最具挑战的区域(最高仅 0.4314),反映了其序列设计的复杂性。
参数对AntiBMPNN性能的影响
在对训练噪声水平与采样温度的影响进行评估时,作者发现 AntiBMPNN 与 ProteinMPNN 呈现相似的趋势:随着噪声或温度升高,两者的序列恢复率均持续下降。AntiBMPNN 在三种测试集上的最低平均恢复率约为 60%,而 ProteinMPNN 仅约 30%,说明高噪声或高温度会削弱模型准确复现序列的能力,但也可能有利于生成更多样的新序列。图 2 则展示了测试序列与训练序列相似性对恢复率的影响。在 40 条 H 链和 40 条 L 链晶体/冷冻电镜结构上,作者计算了序列相似度并用线性回归绘制趋势线,得出较高的皮尔逊相关系数:H 链约 0.80、L 链约 0.65。结果表明,相似度越高,序列恢复越好,说明 AntiBMPNN 对已见过的序列片段表现更可靠,对全新或高度变异序列的泛化能力则相对受限。进一步实验表明,将骨架噪声或采样温度中的任一参数固定在 0.5(中等且稳定)并调节另一参数,可显著提升 H 链与 L 链的恢复表现。这表明适度而稳定的噪声或温度(≈0.5)有助于优化 AntiBMPNN 的序列恢复率。值得注意的是,虽然 L 链整体恢复率高于 H 链,H 链的恢复率与序列相似度的相关性更强,暗示两类链在相似度对恢复效果的敏感度上存在差异。
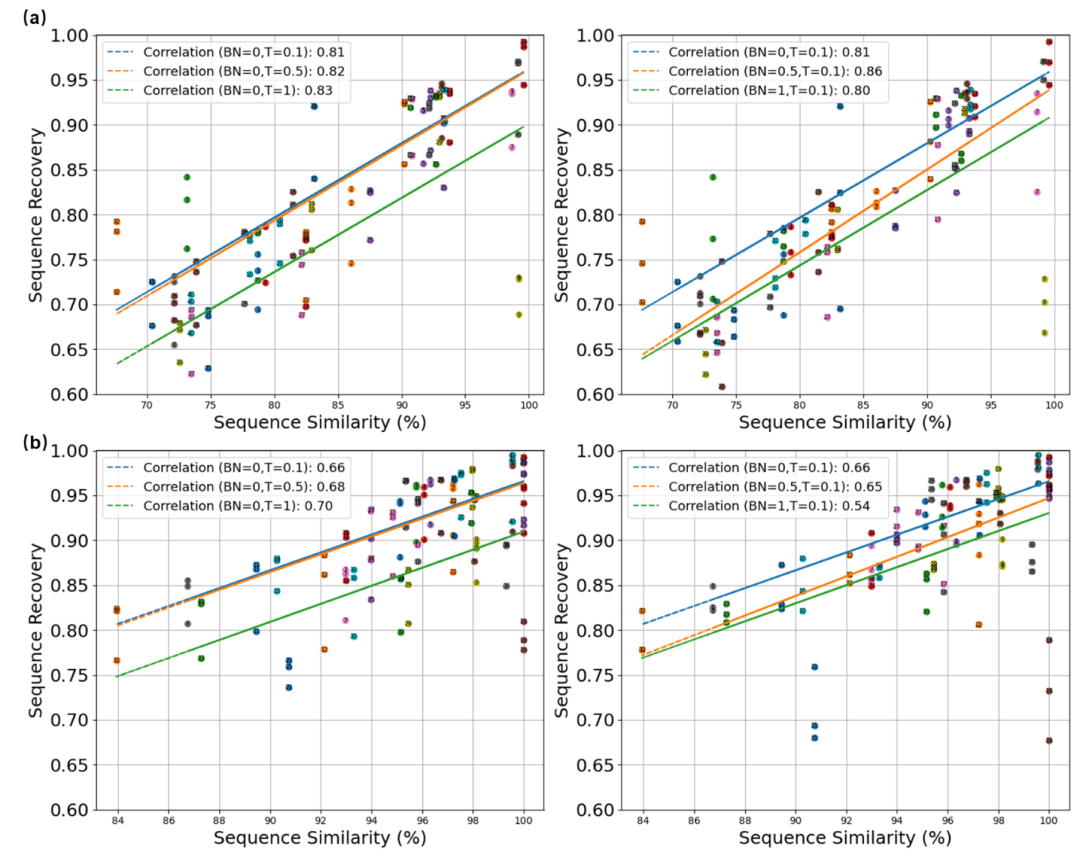
图2. 序列相似性对 AntiBMPNN 和 ProteinMPNN 序列恢复的影响。
基于频率的评分函数和 AlphaFold 3 用于序列选择
AntiBMPNN 在不固定或固定指定残基的情况下批量生成序列,但因相同序列恢复率下仍会出现大量重复序列且评分差异大,用户难以挑选。为此,作者设计了基于出现频次的评分函数(流程见图3a):先按全长序列聚类,再用各序列在全集中的出现次数排序。以抗体 huJ3 为例,AntiBMPNN 仅在 CDR3 的六个位点(K97、I101、Y103、N104、S105、N106)进行设计,生成 32768 条序列,最终归为 147 个簇。出现频次最高的序列 “ARSKSTYLSRDSSGYDY”(位点 94-110)出现 6495 次,与原序列相比在 I101、Y103、N104、N106 四处发生替换,整体序列恢复率为 0.67。频次统计与各突变位点的化学性质分布(图 3b,c)直观展示了模型的偏好,便于筛选候选序列。随后作者用 AlphaFold 3 进一步评估高排名序列的结构可靠性与抗原结合潜力:先在单体模式下为每条抗体序列生成 5 个模型,取最高 pLDDT 分值的模型代表其自我折叠可能性;再与相应抗原(如 gp120)联合建模生成 5 个复合物模型,取 ranking_score 最高者代表其结合构象(图3d)。每种建模方法 pLDDT 或 ranking_score 最优的 1–2 条序列被选入后续实验验证。
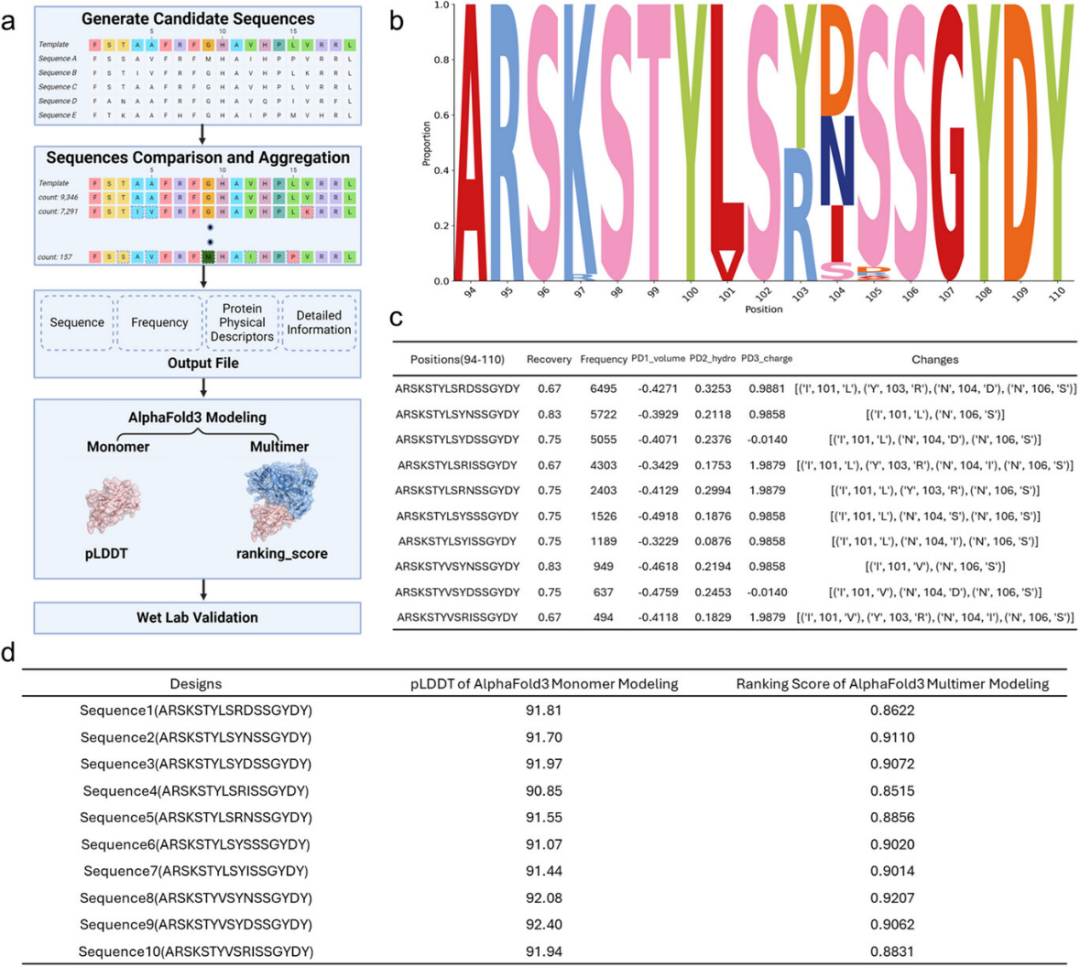
图3. AntiBMPNN和AlphaFold3中的评分函数的集成,对每个设计序列的候选进行排序
使用 AntiBMPNN 进行单点序列设计并进行实验验证
作者将人源化的骆驼纳米抗体 huJ3为例,其中 CDR3(位点94-110)的 “ARSKSTYISYNSNGYDY” 片段尤为关键;既往研究已指出其中 “STY” 三肽(位点 98–100)对配体结合极为敏感。为开展序列设计,作者首先以 PDB 7ri1 中的 J3 结构为模板,构建了 huJ3 及其与抗原 gp120 的复合物三维模型。随后聚焦 “SYNSN”(位点 102–106)进行单点突变设计并实验验证。AntiBMPNN 预测的最高频替换为 S102S(保持不变,99.0%)、Y103R(90.2%)、N104S(99.7%)、S105P(98.4%)和 N106S(>99.9%);同时也探索了 S102N、N104A、N104M 与 Y103H 等低频变体。八个单点设计中有六个保持了与父本 huJ3 相当的亲和力(EC50=2.3-6.7 nM),包括 S105P、N106S、N104A、N104M、S102N、N104S;而 Y103H 使亲和力降低约 13 倍,Y103R 则几乎失去结合能力。这些计算与实验结果进一步验证了 AntiBMPNN 的可靠性,并为将其扩展至更广泛的抗体 CDR 设计提供了依据。
使用 AntiBMPNN 进行 huJ3 CDR1 全长序列设计及实验验证
研究者在骨架噪声 BN = 0.5、温度 T = 0.1 的条件下,针对 huJ3 抗体的 CDR1 区(26-33 位,基序 “GSIFNQYA”)进行全长序列设计。结果显示 26、27、29、32、33 位点(G、S、F、Y、A)高度保守,即使将 BN 提高到 0.7 仍保持不变,而 28、30、31 位(I、N、Q)则呈现较大可变性。基于评分函数和 AlphaFold 3,筛选出五条候选序列 1-V1 至 1-V5 进行实验验证。序列比对显示这些变体在 CDR1 的若干残基与对照(nanoJ3、huJ3)不同;ELISA 结果表明,1-V1 至 1-V4 的 EC50 介于 9.2-99.6 nM,1-V5 则未检测到结合活性。结构分析指出,Q31 常被小体积的甘氨酸取代导致接触不足,而 1-V1 通过 I28R 补偿,R28 与 D30 与 gp120 表面形成关键相互作用,因此亲和力最好(EC50 ≈ 9.2 nM)。相较之下,CDR1 对抗原结合的重要性通常低于 CDR3;下一步作者将利用 AntiBMPNN 方法设计 huJ3 的 CDR3 并进行实验验证。
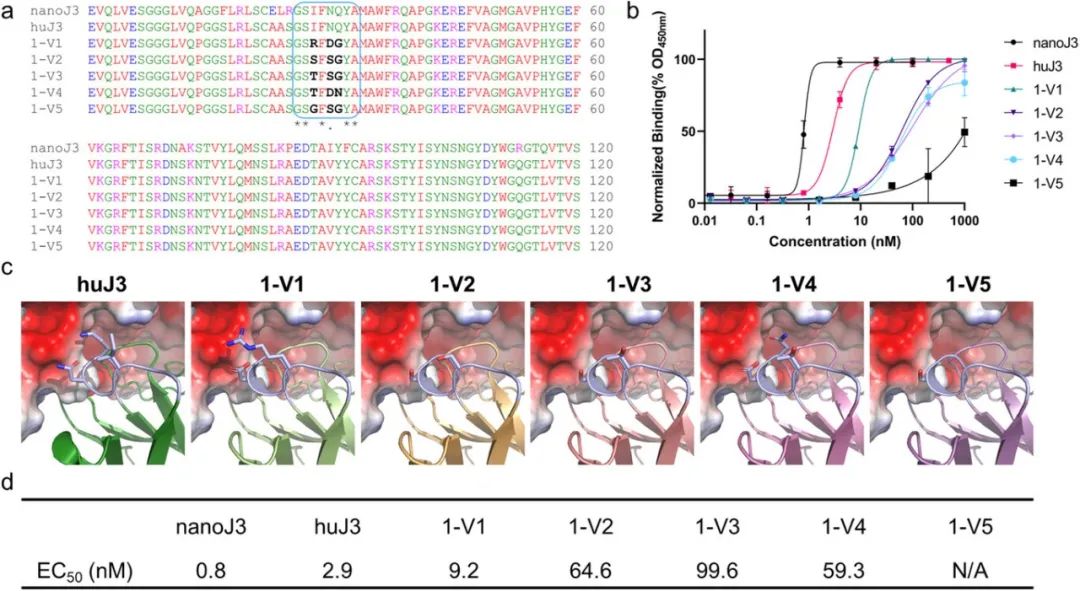
图4. AntiBMPNN设计的huJ3 CDR1的实验验证结果。
使用 AntiBMPNN 进行 huJ3 CDR3(残基 94 至 105)序列设计并进行实验验证
研究团队继续对 huJ3 抗体的 CDR3 区域进行定向设计,靶向 94-105 位残基(完整 CDR3 序列为 “ARSKSTYISYNSNGYDY”,94-110 位)。鉴于原始研究表明 “STY” 保守基序中的 T98R 与 Y99G 单点突变会完全失去结合活性,本次设计固定了 98-100 位的 “STY” 以保持功能。采用与 CDR1 相同的建模体系(huJ3-gp120 复合物,BN = 0.5,T = 0.1),结果发现 R95、K97、S102、N104、S105、N106 等位点可被多种氨基酸替换。依据评分函数与 AlphaFold 3,筛选出 11 条高分序列进行实验验证。
实验显示,重设计获得的 3-V1 至 3-V9 的 EC50 值跨度大(1.7-418.9 nM)。其中 3-V1 的 EC50 为 1.7 nM,优于母本 huJ3(2.9 nM),其 H97、N102、S106 三个新引入残基与 gp120 形成关键相互作用。3-V3 的 H97、N102、S104 设计亦带来 6.6 nM 的 EC50,略低于 huJ3。相反,3-V10 与 3-V11 由于在关键位点(如 R95K、T99I、Y100R)做出不当改动,完全失去结合活性。整体结果进一步验证了 AntiBMPNN 在 CDR3 精准重设计中的有效性与挑战并存的特征。
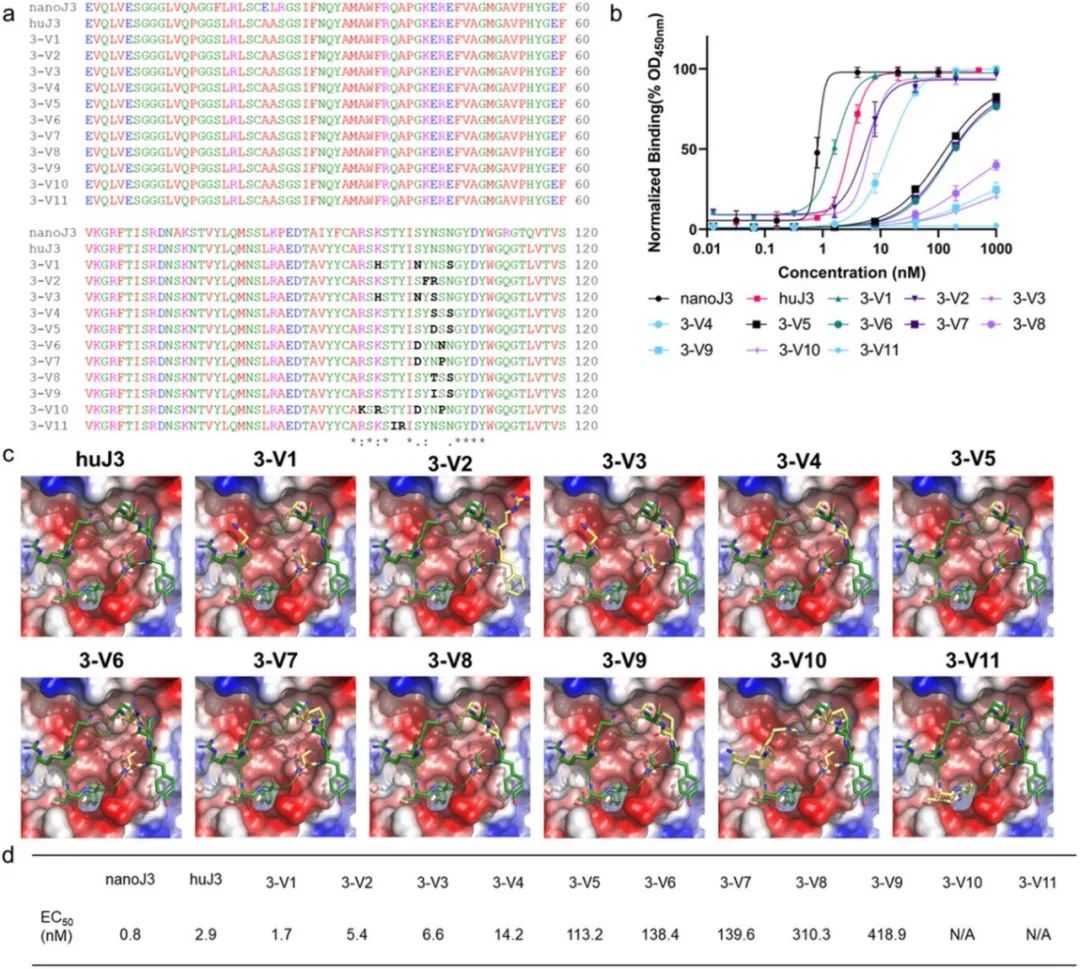
图5. AntiBMPNN 设计的 huJ3 CDR3 变体的实验验证结果.
使用 AntiBMPNN 进行抗 CD16A VH 抗体 (D6) 的 CDR2 序列设计及实验验证
为评估 AntiBMPNN 的稳健性和实用性,研究者将其用于靶向 CD16A 的单域抗体 D6(VH)的 CDR2 设计。首先构建了 D6 的 3D 结构,在 BN = 0.5、T = 0.1 条件下,针对 CDR2 基序 “SIYYSGSTN”(50-58 位)进行定向突变。结果显示 50、51、53、54、56 位残基较易被替换,而 52、55、57、58 位相对保守。考虑到 D6 原有的 R66L 变异不影响亲和力,设计时保留并与 CDR2 方案合并。依评分函数选出 2-V1 至 2-V4 四个候选体实验验证:序列比对显示关键差异;ELISA 结果与对照 D6 比较后,结构对接指出 Y53 对结合至关重要,其酚羟基与抗原表面高度互补,突变(2-V1、2-V4)显著降低亲和力;S50 则耐受性较高,适度延伸侧链可提高(2-V2)或不损伤(2-V3)亲和力。各变体 EC50 值如图 6d 所示,整体印证了 AntiBMPNN 在 CDR2 精准优化中的效能与位点敏感性。
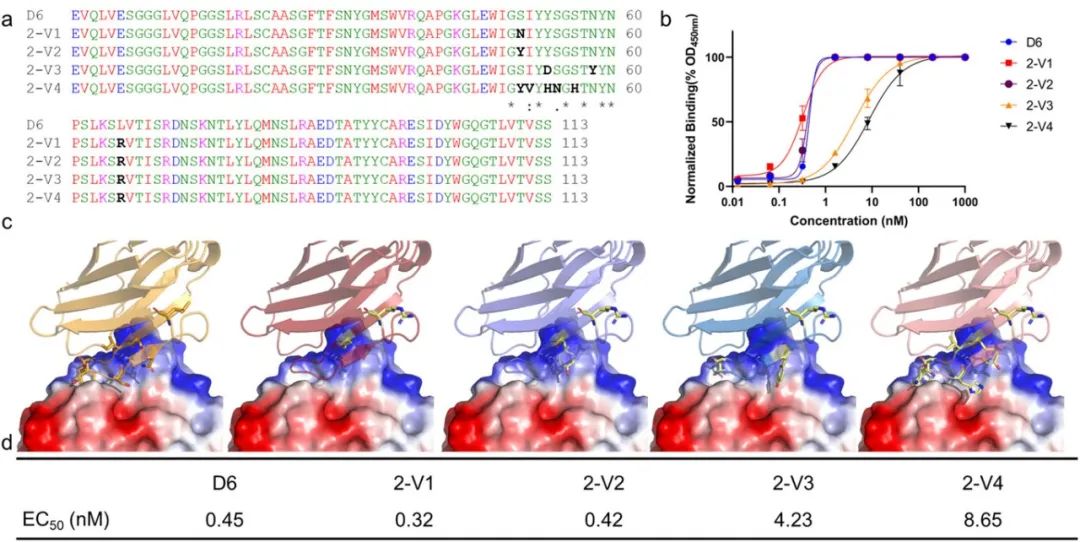
图6. AntiBMPNN 设计的 D6 CDR2 变体的实验验证结果。
AntiBMPNN与现有基于实验数据的抗体序列设计方法的比较分析
为了评估不同生成模型的性能,本研究首先以AntiBMPNN、ProteinMPNN和AbMPNN三种模型对CDR3区(97至106位氨基酸残基)进行了重新设计,保持关键“STY”序列不变。这些模型产生了含2至5个突变的变体,但所有设计序列均未保持可测量的结合活性,这强调了维持关键功能残基和严格控制突变范围的重要性。
作者进一步对AntiBMPNN与AbMPNN、AntiFold和ProteinMPNN等方法进行了全面对比。针对huJ3体系,优化CDR1区的关键残基28、30、31位,AntiBMPNN设计的“RDG”序列取得了出色的结合亲和力(EC50为9.2 nm),优于其他模型。相较之下,AbMPNN设计的两个序列分别为非结合的“GSS”和较优的“PSK”(EC50为6.6 nm);AntiFold最佳序列“TDN”的EC50为59.3 nm;ProteinMPNN设计的序列则表现更差(EC50分别为135.2 nm和无结合活性)。因此,AntiBMPNN在CDR1区优化中的表现显著优于其他模型。
在D6抗体中,针对CDR2区的50和66位残基进行优化,AntiBMPNN设计的最佳序列“NR”达到了极低的EC50(0.3 nm),远优于其他模型。AbMPNN的“LG”序列无法在大肠杆菌中表达,“TG”序列EC50为2.3 nm;AntiFold最佳序列“SR”的EC50为0.7 nm,而ProteinMPNN表现欠佳(EC50超过1000 nm)。AntiBMPNN展现了在CDR2区优化上的高精准性和稳定优势。此外,huJ3的CDR3区优化(残基97、102、106位)中,AntiBMPNN设计的“HNS”序列实现了1.7 nm的优秀EC50,显著超过其他方法。AbMPNN设计的“GYG”和“SSS”序列效果较差(无结合和EC50为51.2 nm),AntiFold和ProteinMPNN也未取得有效结合。
最后,本研究使用AlphaFold 3为所有设计序列及其抗原复合物生成了“ranking_score”,但发现这些结构可信度评分与实验测得的结合活性并不总是相关。例如,在huJ3再设计中,结构评分几乎相同的两个序列,其EC50值却差异超过一个数量级(6.6 nm和135.2 nm)。此外,D6抗体由于缺乏实验解析结构,导致AlphaFold 3的预测可信度较低。这表明仅依靠结构评分预测功能结果存在局限,需结合其他指标或实验验证。
总结
作者介绍了 AntiBMPNN,一种结构驱动的图神经网络框架,用于精准抗体设计与工程。该模型在特定的三维抗体结构数据集上进行微调,展示出卓越的序列恢复能力,并在多项基准测试中显著优于已有方法。作者进一步将其应用于人源化纳米抗体(如 huJ3)和其他抗体 CDR 区域的设计,为实验验证筛选出高亲和力候选序列,证明了模型的实用价值。此外,通过结合 AlphaFold 3 对候选序列进行结构与配体结合模拟,作者还提出了高效的筛选策略,为后续实验研究提供强有力的支持。总体而言,本研究构建了一个基于结构信息的深度学习平台,为抗体工程提供了精准设计的新路径。
参考资料
Ze-Yu Sun, Jiayi Yuan, Divya Jaiswal, Jingxuan Ge, Tianjian Liang, Jiahui Wei, Jinghong Cao, Yulong Li, Xiaojie Chu, Yan Chen*, Ying Xue*, Wei Li*, Tingjun Hou*, Zhiwei Feng*. AntiBMPNN: Structure-Guided Graph Neural Networks for Precision Antibody Engineering. Advanced Science, Published online June 27, 2025. doi:10.1002/advs.202504278
内容中包含的图片若涉及版权问题,请及时与我们联系删除

