DRUGAI
尽管RNA靶向药物开发近年来取得了显著进展,但受限于可用的RNA–小分子相互作用数据有限及RNA结构信息稀缺的双重瓶颈,基于数据驱动的深度学习方法仍面临重大挑战。为此,研究人员提出了一种新颖的基于序列的深度学习框架RNAsmol,该模型集成了扰动与数据增强策略、图结构分子特征建模、注意力机制驱动的特征融合模块等多个技术组件,旨在实现对RNA–小分子结合事件的准确预测。RNAsmol在处理数据偏斜方面引入了扰动机制以平衡阴性样本与未知样本空间之间的不均衡,进而揭示RNA–小分子之间潜在的结合规律。模型在十折交叉验证、未见样本测试及诱饵识别任务中均优于现有方法,并通过可视化展示分子结合模式及模型学习到的权重分布,进一步提升了解释能力。值得强调的是,RNAsmol无需依赖RNA三维结构即可提供高质量预测,为当前RNA靶点药物筛选任务提供了通用且高效的工具,具有广泛的应用潜力。

现代药物开发过程不仅耗时耗资,且高度依赖于对疾病靶点的精准识别与候选小分子的有效筛选。尽管当前大多数批准药物仍以蛋白质为靶点,但许多蛋白因缺乏可结合的结构性口袋,被归类为“不可成药”目标,极大限制了药物开发的空间。与此同时,随着非编码RNA功能的逐步揭示,研究人员意识到RNA,尤其是诸如lncRNA、miRNA、核糖开关等结构,承载着大量尚未挖掘的治疗靶点潜力。根据基因组数据统计,约70%的转录产物属于不同类型的RNA,而其中有相当一部分与肿瘤、神经疾病、病毒感染等人类重大疾病密切相关。
尽管RNA靶向的小分子如Evrysdi和ribocil的临床成功表明了这一方向的可行性,但目前的发现过程大多依赖偶然筛选,缺乏系统性。随着自动化筛选平台、小分子阵列和RNA结合分子库(如ROBIN)的建立,RNA–小分子结合的研究逐步具备了数据驱动建模的可能性。然而,即便如此,实验成本高、样本稀缺、RNA结构缺乏的现实问题仍严重阻碍了模型性能的提升。
当前部分研究尝试基于RNA三维结构构建结合位点识别模型,但由于公共数据库中RNA结构数量极少(在RCSB PDB中仅占约3.5%),这些方法的可扩展性受到质疑。此外,相较于蛋白–配体预测任务的繁荣,RNA–小分子预测仍处于早期阶段。因此,研究人员提出了一种完全基于序列的预测框架RNAsmol,旨在利用扰动与增强策略从稀疏的样本网络中学习稳定的RNA–小分子结合模式。
结果
RNAsmol整体框架设计
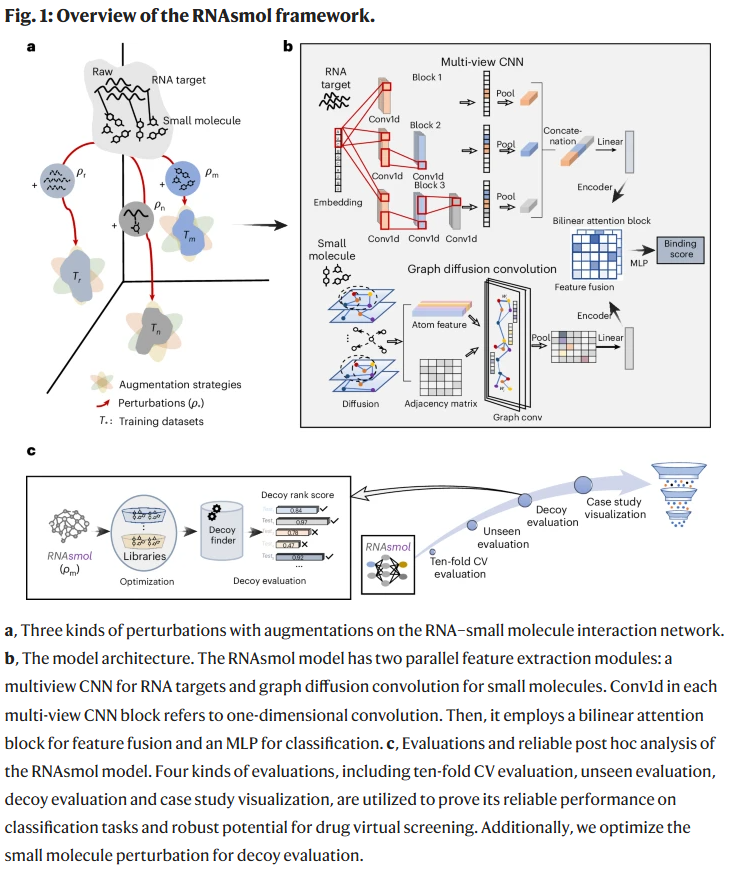
RNAsmol由四大模块组成:RNA目标编码器(多视角CNN)、小分子编码器(图扩散卷积网络)、特征融合模块(双线性注意力机制)以及分类模块(多层感知机)。为了缓解训练样本不足的问题,研究人员在原始RNA–小分子互作网络上构建了三类扰动策略:基于RNA序列的碱基双核苷酸打乱、基于结构指纹相似度的小分子替代扰动、以及RNA–小分子互作图中负样本边的采样。同时,每类扰动配备相应的增强策略,例如同源RNA序列引入、SMILES随机化与结构相似配体替换等,生成了三类增强训练数据。
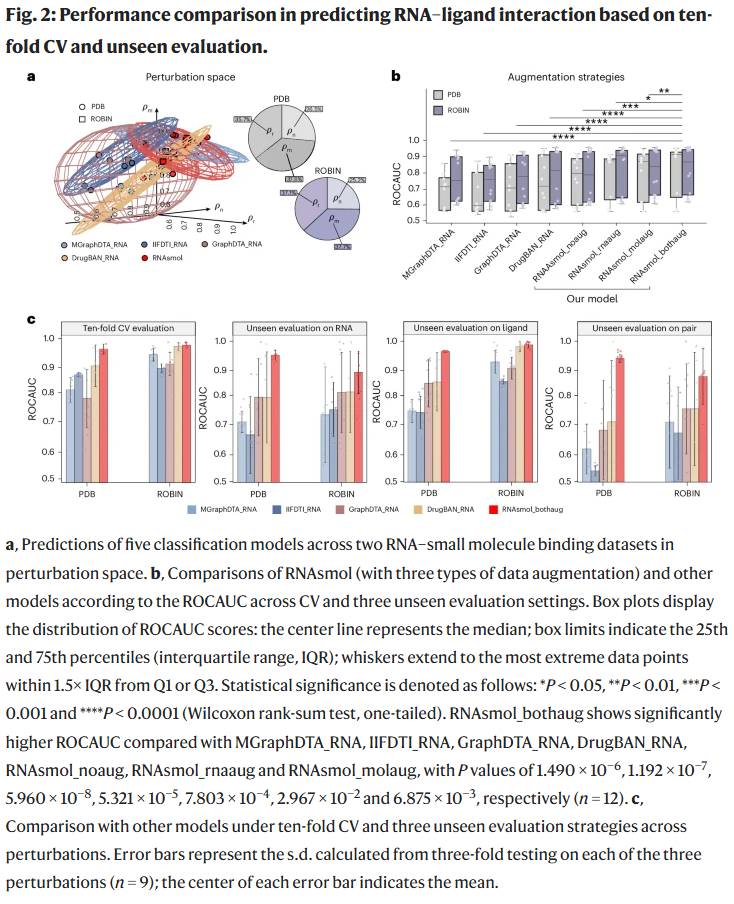
扰动空间下的预测能力评估
在系统评估中,RNAsmol相较MGraphDTA_RNA、IIFDTI_RNA、GraphDTA_RNA和DrugBAN_RNA等代表性模型,表现出更强的泛化能力与稳定性。在三种扰动设置下,RNAsmol均维持在高水平的ROC-AUC与PRAUC表现,且在未见RNA、小分子或RNA–小分子对的评估中,均优于其他方法。特别是双重增强版本RNAsmol_bothaug,在所有测试中实现了最高性能提升,平均提高了14.43%。
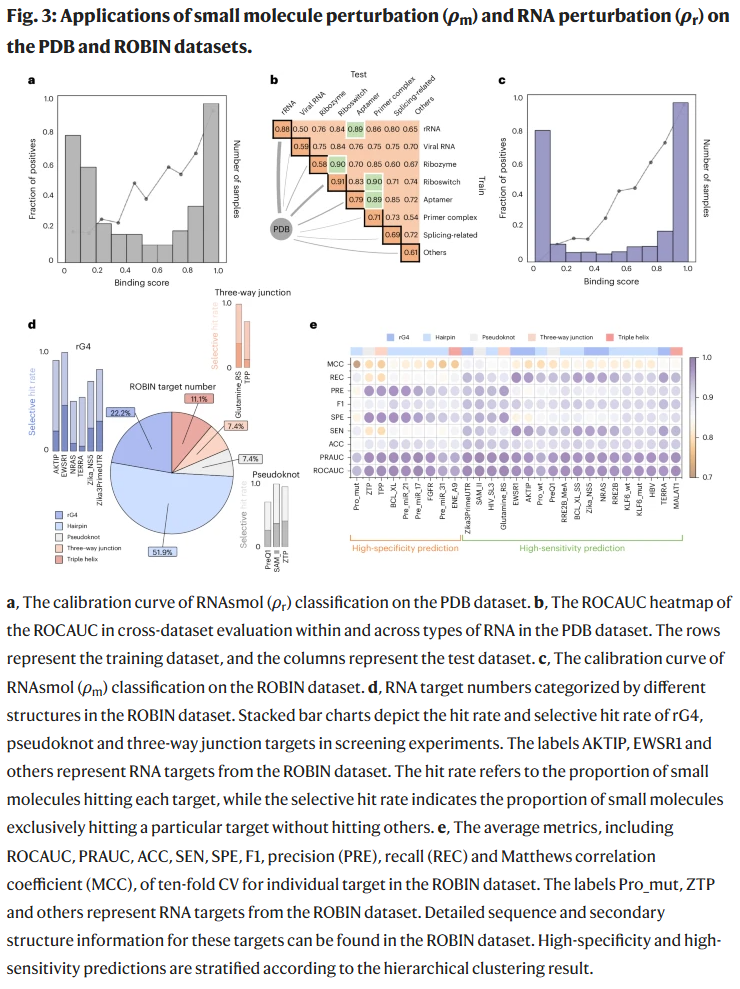
跨数据集与结构类别的泛化测试
跨PDB与ROBIN数据集测试揭示:小分子扰动因其结构特征相似性强,跨域影响较小;而RNA扰动更敏感,反映出RNAsmol在RNA类型层面学到的判别能力更强。研究人员还通过结构层次聚类分析发现,不同RNA二级结构(如G-四链体、三分支结、伪结等)展现出不同的命中率与选择性,提示RNAsmol具备识别结构特异性结合模式的潜能。
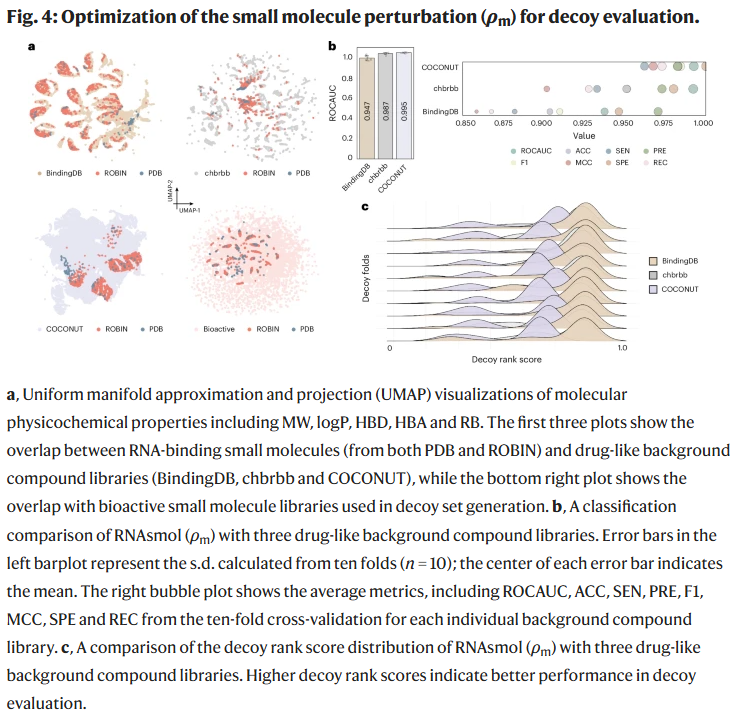
模型优化与诱饵评估
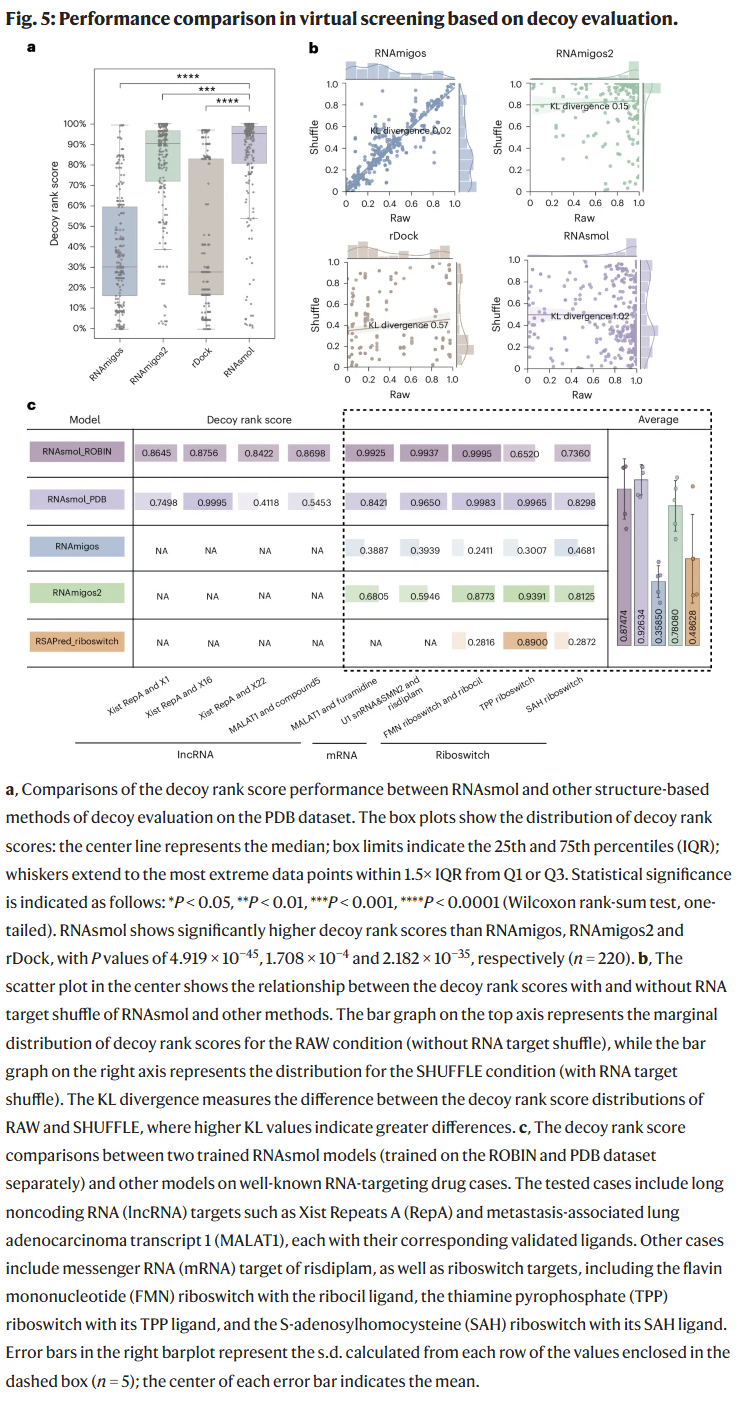
在诱饵评估任务中,RNAsmol使用不同背景化合物库(如BindingDB、COCONUT、ChemBridge)优化分子扰动设置后,取得了显著提升。结果表明,选择BindingDB作为背景库有助于提高模型在真实药物空间中的筛选性能,且RNA配体通常展现出结构选择性,有助于模型学习精准特征。在多种真实RNA药物案例测试中,如risdiplam、ribocil、Xist、MALAT1等,RNAsmol均表现优于传统结构对接模型和现有RNA–小分子预测模型。
预测可解释性可视化分析
研究人员进一步利用Grad-CAM方法可视化模型预测权重,发现在ZTP与PreQ1核糖开关结合复合物中,靠近结合位点的碱基和原子拥有更高的权重值,显著高于远离位点的区域。这一结果不仅验证了模型学习能力,还展示了其在缺乏结构信息情况下对分子结合热点的识别能力。

讨论
RNAsmol代表了一种针对RNA–小分子互作问题的创新建模范式。其关键在于绕开了RNA结构匮乏的瓶颈,提出了一套依赖序列信息、整合扰动与增强策略的建模流程,为RNA靶点药物筛选提供了全新工具。与传统基于RNA结构口袋的对接方法相比,RNAsmol以数据驱动方式学习序列层面的结合模式,不仅适用于结构未知的RNA,还能在结构异质性背景下保持较高鲁棒性。
此外,扰动策略在训练阶段引入伪阴性样本,有效缓解了深度模型记忆训练样本而非学习真实规则的问题。数据增强方法也极大扩展了训练空间,提升了模型对新型RNA类型或新型小分子的泛化能力。研究人员指出,未来可进一步引入实验结合位点注释,细化Grad-CAM解释模块,从而提升预测热点区域的准确性与生物学解释力。
更重要的是,这一框架具有跨任务迁移潜力。虽然本研究聚焦RNA–小分子互作,但类似思路完全可推广至蛋白–配体、多肽–核酸等其他大分子体系,特别是在构建正负样本极度不平衡的场景中,将扰动与增强策略相结合的思想或成为突破模型性能瓶颈的关键路径。
整理 | WJM
参考资料
Ma, H., Gao, L., Jin, Y. et al. RNA–ligand interaction scoring via data perturbation and augmentation modeling. Nat Comput Sci (2025).
https://doi.org/10.1038/s43588-025-00820-x
内容中包含的图片若涉及版权问题,请及时与我们联系删除

