DRUGAI
复杂疾病具有错综复杂的细胞动力学。单细胞转录组学提供了关键洞见,但在详细分析疾病进展和针对性 in silico 药物干预方面,计算工具仍存在空白。研究人员引入了 UNAGI,一种深度生成神经网络,专为分析时间序列单细胞转录组数据而设计。该工具能够捕捉疾病进展背后的复杂细胞动力学,增强药物扰动建模与筛选。当将 UNAGI 应用于特发性肺纤维化患者的数据集时,它学习到的疾病信息化细胞嵌入加深了对病程的理解,并鉴定出潜在的治疗药物候选者。蛋白质组学验证了 UNAGI 在细胞动力学分析方面的准确性;在带纤维化混合物处理的人体精准切片实验中,也证实了其对硝苯地平具有抗纤维化作用的预测。UNAGI 的多功能性延伸到其他疾病(包括 COVID-19),展示了其在解码复杂细胞动力学、助力多种病理学下寻找治疗方案的广泛适用性。

复杂疾病随时间演进,是遗传与环境因素相互作用的结果。这些异质因素在个体和群体中的交互极其复杂,给疾病进展的理解带来了挑战。治疗多因素疾病需要同时干预多个相互作用的过程,但大多数疗法都基于动物或细胞模型,这些模型无法重现人类疾病的复杂性与动态变化。因此,需要新方法来捕捉疾病动力学与细胞复杂性,以促进高效治疗策略的发现和实施。
基于临床数据和电子病历的方法(如布尔网络、贝叶斯网络、支持向量机和决策树)可以绘制疾病连续状态图,但无法深入分子、细胞和基因机制,这主要因缺乏高分辨率的基因组分析。单细胞 RNA 测序(scRNA-seq)正站在解决这一难题的前沿,能够在单细胞分辨率下剖析复杂系统,发现罕见细胞类型,并揭示异常细胞群体。但现有方法往往将 scRNA-seq 数据视为离散快照,忽略了时间序列数据的连续性与进展性。
尽管已有方法(如 scVI、scGen 等)可进行 in silico 扰动,但它们并非为时间序列数据或复杂疾病专门设计,通常缺乏对疾病特异性信号的优化,也无法在无监督情况下充分模拟药物干预。面对日益丰富的公共药物数据库(如 Connectivity Map),亟需一种可无监督理解疾病进展并进行大规模药物虚拟筛选的方法,同时配备交互式可视化,以高效探索潜在药物并推动后续实验验证。
结果
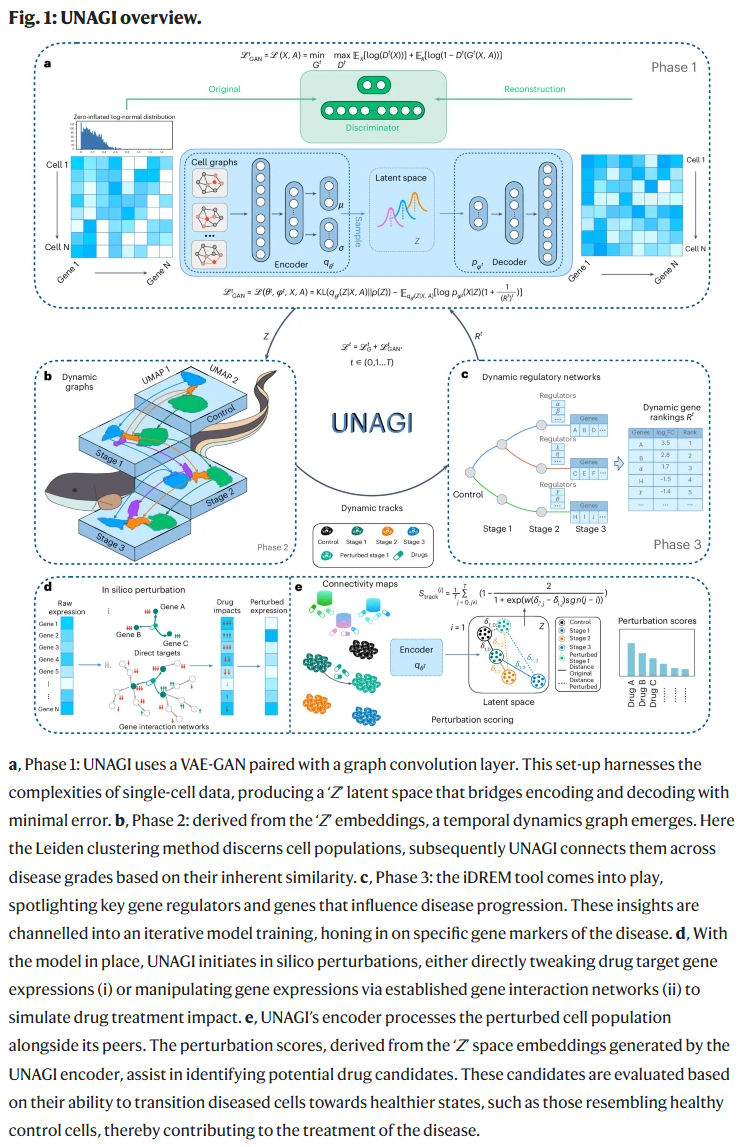
概念框架与模型架构
嵌入学习(Phase 1):将单细胞数据视为零膨胀对数正态分布,引入图卷积层(GCN)以整合细胞间邻接关系,缓解稀疏与噪声;随后 VAE-GAN 在潜空间中学习稳定且可生成的细胞嵌入,GAN 判别器保证生成细胞真实性。
时序动力学图构建(Phase 2):对各病程阶段细胞嵌入进行 Leiden 聚类并 UMAP 可视化,通过计算相邻阶段簇间的多维正态分布 KL 散度与前 100 个差异表达基因(DEG)距离之和,选择统计显著连接构建有向轨迹图,揭示细胞状态迁移路径与分支。
基因调控网络重建:沿着每条细胞轨迹,运用 iDREM 推断关键基因调控子及其下游靶基因,形成病程特异的动态 GRN,为后续嵌入学习提供因果洞见。
迭代训练策略(Phase 3):初始迭代中所有基因权重均等;重建 GRN 后,提升关键基因与调控子的权重,同时进行权重衰减以抑制噪声基因;在下一轮 VAE-GAN 学习阶段,将这些权重融入重构损失,持续强调疾病相关基因,直至收敛。
无监督 in silico 扰动(Phase 4):利用训练好的生成模型,在潜空间施加药物或通路干预,计算扰动后细胞嵌入与健康对照簇的距离变化,输出扰动分数并筛选排名靠前的候选药物或通路。
IPF 应用与验证
使用 19 名供体(10 健康、9 IPF 患者,共 231,477 细胞、2,484 基因)snRNA-seq 数据集,UNAGI 生成的低维嵌入在 ARI、孤立标签 F1、ASW 等指标上优于 scVI、RVAgene 等方法。
成纤维细胞轨迹:识别出 FibAlv-4、FibAdv-17 两条主要轨迹,分别对应肺泡与增殖性、血管周与导管周成纤维细胞。FibAlv-4 路径上,EP300、CTCF、RAD21、SMC3 等调控子居于核心,靶基因包括 LTBP1、LTBP2 及 COL3A1、SERPINE1;富集通路涵盖胶原/ECM 重塑、PI3K-Akt-mTOR、SLIT/ROBO 信号,以及 NCAM1 相互作用等新颖通路。FibAdv-17 路径揭示了基质重塑相关通路(胶原形成、组织化、三聚化与降解)与 MET-PTK2 信号,以及 IPF 关联基因如 KCNMA1、NPAS2、ITGA8、DIO2 等。
药物候选预测:无监督扰动模块优先预测出硝苯地平、HDAC 抑制剂等,后续通过蛋白质组学分析与人体精切肺切片实验验证其潜在抗纤维化作用。
泛化至 COVID-19
在来自 130 位患者(年龄 50–69 岁)的 PBMC scRNA-seq 数据集中(共 246,948 细胞),UNAGI 同样成功重构了不同严重度群体的细胞动力学,验证了其对其他复杂疾病的适用性。
结论
UNAGI 通过紧密结合嵌入学习与因果推断,实现了多尺度的细胞动力学与基因调控网络建模,并在此基础上开展大规模 in silico 药物虚拟筛选。与现有方法相比,其优势在于:
疾病聚焦嵌入:迭代融入关键基因权重,提升模型对病程特异变化的敏感度与嵌入可解释性;
动力学–因果集成:KL 散度+DEG 距离构建的动力学图与 iDREM 推断的 GRN 互为补充,精确揭示细胞状态迁移及其调控网络;
无监督药物筛选:无需带标注的扰动数据,即可模拟千余种药物/通路干预,并提供可交互评分,促进候选靶点与药物的高效发现;
广泛应用前景:在 IPF 与 COVID-19 数据上均表现优异,可推广至其他复杂疾病或发育系统(如胚胎发生、器官形成、神经发生)以挖掘调控机制并指导干预策略。
总之,UNAGI 为解码复杂疾病中细胞动力学与基因调控提供了强大计算工具,并为药物发现与精准治疗开辟新路径。
整理 | WJM
参考资料
Zheng, Y., Schupp, J.C., Adams, T. et al. A deep generative model for deciphering cellular dynamics and in silico drug discovery in complex diseases. Nat. Biomed. Eng (2025).
https://doi.org/10.1038/s41551-025-01423-7
内容中包含的图片若涉及版权问题,请及时与我们联系删除

