蛋白质结合剂(如抗体、抑制肽)在疾病诊断、成像分析及靶向药物递送等关键场景中发挥着不可替代的作用。传统上,高特异性蛋白质结合剂的开发高度依赖噬菌体展示、定向进化等实验技术,但这类方法普遍面临资源消耗巨大、研发周期冗长的挑战,且受限于蛋白质序列组合复杂性的固有瓶颈。
随着人工智能的发展,蛋白质语言模型(PLMs)成为了理解蛋白质序列与功能关系的重要工具。面向蛋白质结合剂设计,PLMs 基于语言模型的生成能力,可直接从目标蛋白序列出发,设计具有高结合亲和力的配体蛋白或抗体片段。但同时其也面临挑战,例如缺乏兼具长上下文建模能力与真实生成力的 PLMs,尤其在设计具有复杂结合界面和长蛋白质结合剂方面存在显著技术缺口。
基于此,来自阿联酋阿布扎比 Inception AI 研究所与美国硅谷 Cerebras Systems 公司的联合研究团队,提出了首个仅依赖蛋白质序列信息、无需三维结构输入的 PLMs 家族——Prot42 。该模型利用自回归和仅解码架构(decoder-only)的生成能力,能在无结构信息时生成高亲和力蛋白质结合剂和序列特异性 DNA 结合蛋白。在 PEER 基准测试、蛋白质结合剂生成和 DNA 序列特异性结合剂生成实验中,Prot42 表现优异。
相关研究以「Prot42: a Novel Family of Protein Language Models for Target-aware Protein Binder Generation」为题,已在 arXiv 发表预印本。
研究亮点* Prot42 采用渐进式上下文扩展训练策略,从初始 1,024 氨基酸逐步扩展至 8,192 氨基酸。* 在 PEER 基准测试中,Prot42 在蛋白质功能预测、亚细胞定位、相互作用建模等 14 项任务中表现优异。* 与依赖 3D 结构的 AlphaProteo 不同,Prot42 仅需目标蛋白序列即可生成结合剂。

论文地址:
更多 AI 前沿论文:
https://go.hyper.ai/UuE1o
开源项目「awesome-ai4s」汇集了百余篇 AI4S 论文解读,并提供海量数据集与工具:
https://github.com/hyperai/awesome-ai4s
该研究中使用了多个关键数据集来训练和评估其模型性能。这些数据集不仅涵盖了广泛的蛋白质序列信息,还涉及蛋白质与 DNA 的相互作用数据,为 Prot42 提供了丰富的训练素材。
为了设计能够与靶向 DNA 序列结合的蛋白质,研究人员使用了 PDIdb 2010 数据集。作为 Norambuena 和 Melo 的外显数据集,该数据集包含 922 个独特的 DNA – 蛋白质对,用于训练和评估 Prot42 在生成特定 DNA 序列结合蛋白方面的能力。为了评估四种 DNA – 蛋白质模型,研究人员从各种 PDB 结构中提取了 DNA 片段,包括 1TUP 、 1BC8 、 1YO5 、 1L3L 、 2O4A 、 1OCT 、 1A1F 和 1JJ6 。
Prot42 模型的预训练数据集主要来源于 UniRef50 数据库,该数据库包含了 6,320 万个氨基酸序列,涵盖了广泛的生物种类和蛋白质功能。这些序列通过聚类处理,将相似度超过 50% 的序列归为一组,从而减少了数据冗余,提高了训练效率。
在训练 Prot42 之前,研究团队对 UniRef50 数据集进行了预处理,使用 20 种标准氨基酸的词汇对其进行标记,使用 Xtoken 代表氨基酸残留(X 用于标记不常见或模糊的氨基酸残基)。
在数据预处理阶段,研究团队对序列进行了最大上下文长度为 1,024 个 tokens 的处理,并排除了超过此长度的序列,最终得到了一个包含 5,710 万个序列的过滤数据集,初始填充密度为 27% 。为了提高数据利用率和计算效率,研究团队采用了可变序列长度(VSL)填充策略,最大限度地提高了固定上下文长度内的 tokens 占用率,最终将数据集减少到 1,620 万个填充序列,填充效率达到 96% 。
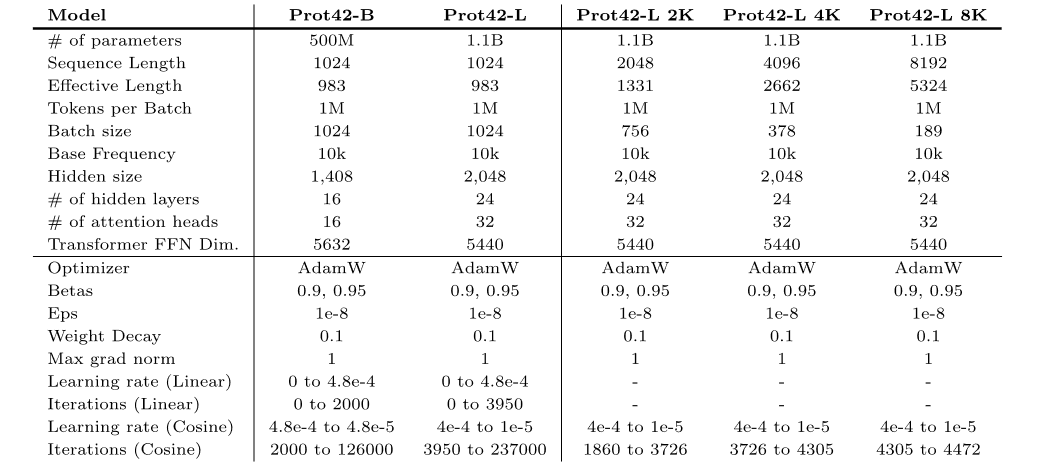
STRING 数据库是一个综合性的蛋白质-蛋白质相互作用数据库,整合了实验数据、计算预测和文本挖掘结果,提供了蛋白质相互作用的置信度评分。为了训练 Prot42 生成蛋白质结合剂,研究团队从 STRING 数据库中筛选了置信度评分 ≥ 90% 的蛋白质相互作用对,确保了训练数据的高可靠性。此外,序列长度被限制在 250 个氨基酸以内,以聚焦于可管理的单域结合蛋白。经过筛选,最终的数据集包含 74,066 个蛋白质 – 蛋白质相互作用对。训练集 D(train)(pb)包含 59,252 个样本和一个验证集 D(val)(pb)包含 14,814 个样本。
本文提到的 Prot42 是一种基于自回归解码器架构的 PLMs,逐个生成氨基酸序列,利用前一个生成的氨基酸预测下一个氨基酸,这种架构使得模型能够捕捉序列中的长距离依赖关系,能够直接从大量未标记的蛋白质序列数据库中学习丰富的表征,有效弥合了已知蛋白质序列数量庞大与蛋白质序列占比相对较小(同时,模型包含多个 Transformer 层,每层包含多头自注意力机制和前馈神经网络,用于捕捉序列中的复杂模式。
其设计灵感来源于自然语言处理领域的突破性进展,特别是 LLaMA 模型。 Prot42 通过在大规模未标记蛋白质序列上进行预训练,捕捉了蛋白质的进化、结构和功能信息,从而实现了高亲和力的蛋白质结合剂生成。
在此基础上,研究人员预训练了 2 个模型变体,即 Prot42-B 和 Prot42-L 。
* Prot42-B:基础版本,模型参数量达 5 亿,支持的最大序列长度为 1,024 个氨基酸。
* Prot42-L:大型版本,模型参数量达 11 亿,同样支持最大序列长度为 1,024 个氨基酸。通过连续预训练策略,Prot42-L 的上下文长度从 1,024 个氨基酸逐步扩展至 8,192 个氨基酸,这一过程中使用了逐渐增加的上下文长度和恒定的 batch 大小(100 万个非填充 tokens),确保了模型在处理长序列时的稳定性和效率,显著提升了模型处理长序列和复杂蛋白质结构的能力。此外,Prot42-L 还包含 24 个隐藏层,每层有 32 个注意力头,隐藏层维度为 2,048 。
为了在下游任务验证之前评估 Prot42 模型的性能,研究人员使用了复杂性(PPL)评估自回归语言模型的标准度量,即 Prot42 模型在不同上下文长度下的性能。所有模型在 1,024 个 tokens 时,困惑度都相对较高,但在 2,048 个 tokens 时有显著改善,降至约 6.5 。结果显示,基础模型以及针对较短上下文进行微调的模型,在各自的最大上下文长度范围内,呈现出相似的性能模式。 8k 上下文模型的表现尤为引人注目——尽管在中等长度序列(2,048 – 4,096 个 tokens)中,其困惑度略高一些,但它能够处理长达 8,192 个 tokens 的序列,并在最大长度时达到了最低困惑度 5.1 。在超过 4,096 个 tokens 后,困惑度曲线呈现下降趋势。如下图所示。
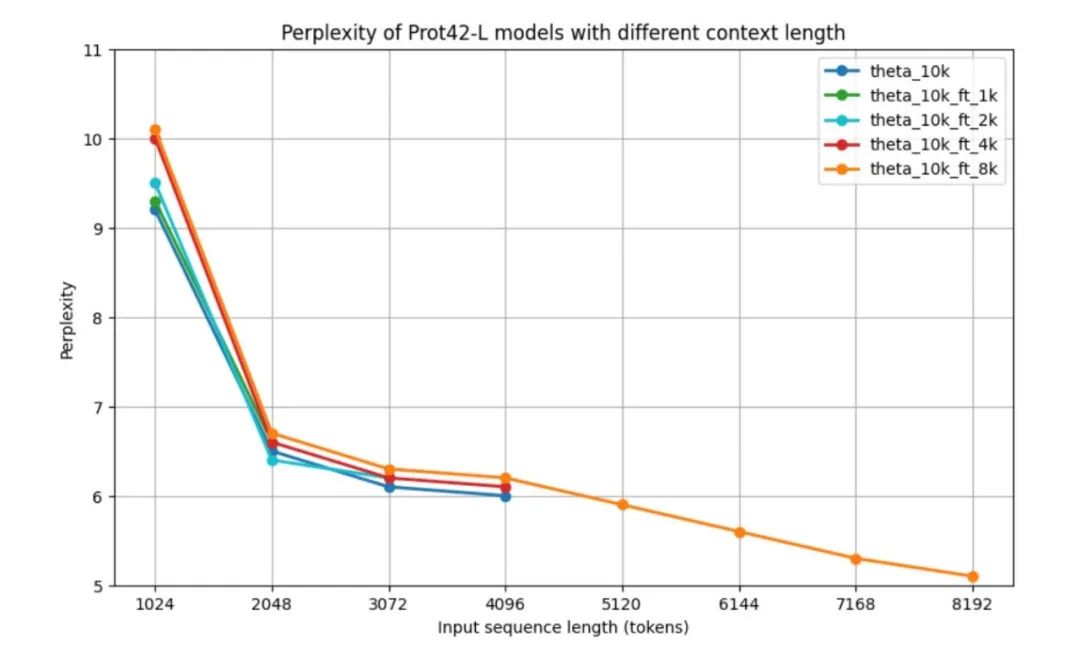
随着上下文长度的增加,模型的 PPL 逐渐降低,表明模型在处理长序列时的能力得到了显著提升。特别是 8K 上下文模型达到最低的 PPL,表明其能够有效利用扩展的上下文窗口来捕捉蛋白质序列中的长程依赖关系。扩展的上下文窗口是蛋白质序列建模领域的一项重大进展,使得能够更准确地表示复杂蛋白质以及蛋白质 – 蛋白质相互作用,这对于生成有效的蛋白质结合剂至关重要。
通过一系列严格的实验评估,Prot42 在多个关键任务上展现了卓越的性能,证明了其在蛋白质结合剂生成和特定 DNA 序列结合蛋白设计方面的有效性。
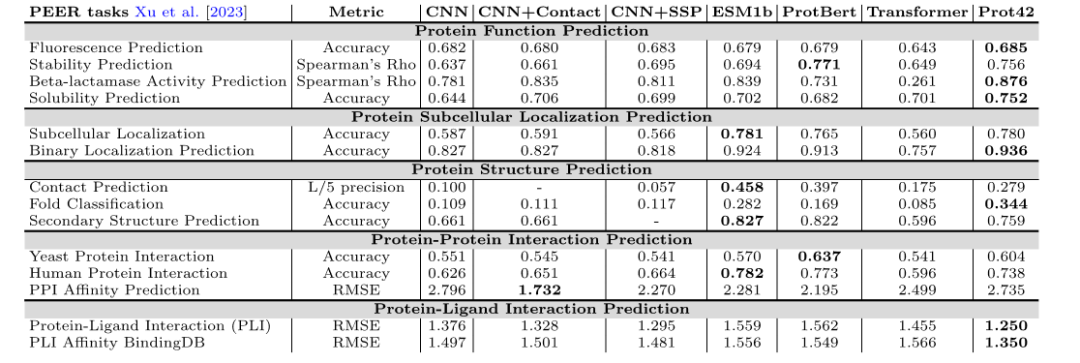
在 PEER 基准测试中,Prot42 模型在多个蛋白质功能预测任务上表现出色,包括荧光预测、稳定性预测、β-内酰胺酶活性预测和溶解度预测等。与现有模型相比,Prot42 在稳定性预测、溶解度预测和 β-内酰胺酶活性预测等方面取得了显著优势,表明其在高分辨率蛋白质工程任务中的巨大潜力。
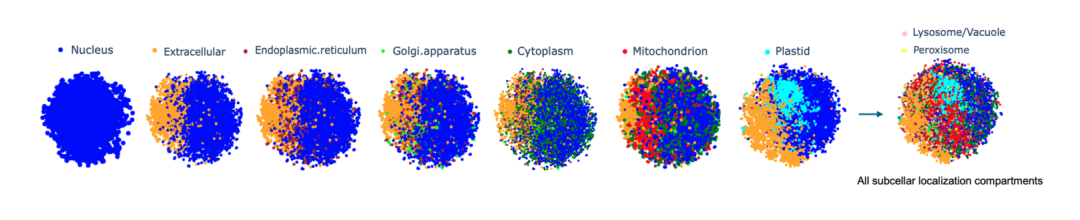
研究人员把每一个蛋白质序列表示为一个大小为 32×2048 的高维向量,在整个蛋白质序列中嵌入 Prot42-L 模型并进行计算。为了直观地评估质量在嵌入和区室的分化,研究人员应用了 t 分布的随机邻域嵌入 (t-SNE) 来降低维度,使得蛋白质基团的可视化变得清晰。经验证,Prot42 在蛋白质亚细胞定位预测任务上表现出色,其准确性与现有先进模型相当。通过可视化分析,研究团队进一步验证了 Prot42 模型在捕捉蛋白质亚细胞定位特征方面的有效性。
在蛋白质结构预测任务中,Prot42 模型在接触预测、折叠分类和二级结构预测等方面取得了优异成绩。这些结果表明,Prot42 模型能够捕捉蛋白质结构中的细微差别,为复杂的生物相互作用建模和制药应用提供了有力支持。
在蛋白质-蛋白质相互作用和蛋白质-配体相互作用预测任务中,Prot42 模型展示了高精度和可靠性,在蛋白质-配体相互作用预测中,研究人员利用 Chem42 生成化学嵌入向量,并且与 ChemBert 进行了对比分析,将其作为另一种化学表征模型,即便如此,其性能指标仍优于现有方法,且接近使用 Chem42 所取得的结果。特别是在使用 Chem42 生成化学嵌入的情况下,其预测结果接近专业化学模型。这表明 Prot42 在结合化学信息方面具有很好的扩展性,为药物设计提供了有力支持。
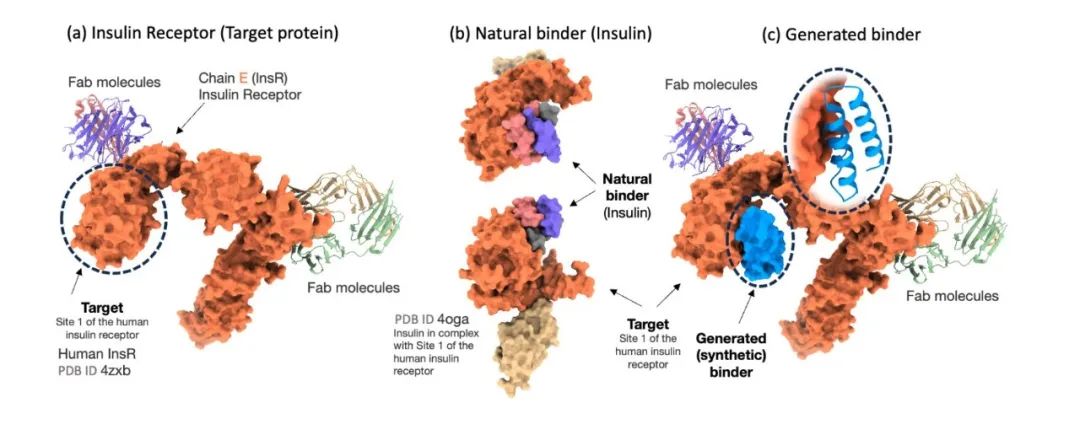
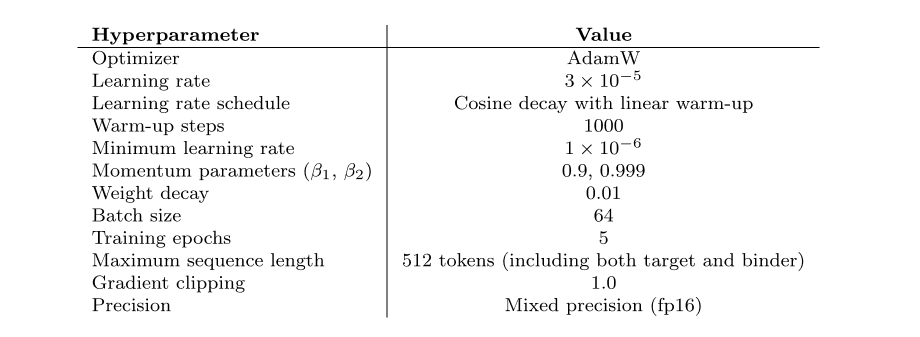
为严谨评估 Prot42 模型在蛋白质结合剂生成方面的效果,研究人员将该模型与专门为蛋白质结合剂预测设计的先进模型 AlphaProteo 进行了对比。实验结果显示,Prot42 模型在多个治疗相关目标上生成了具有强预测亲和力的结合剂,特别是在 IL-7Rα、 PD-L1 、 TrkA 和 VEGF-A 等目标上,Prot42 模型的表现显著优于 AlphaProteo 模型。这些结果表明,Prot42 模型在蛋白质结合剂生成方面具有显著优势。如下图所示。
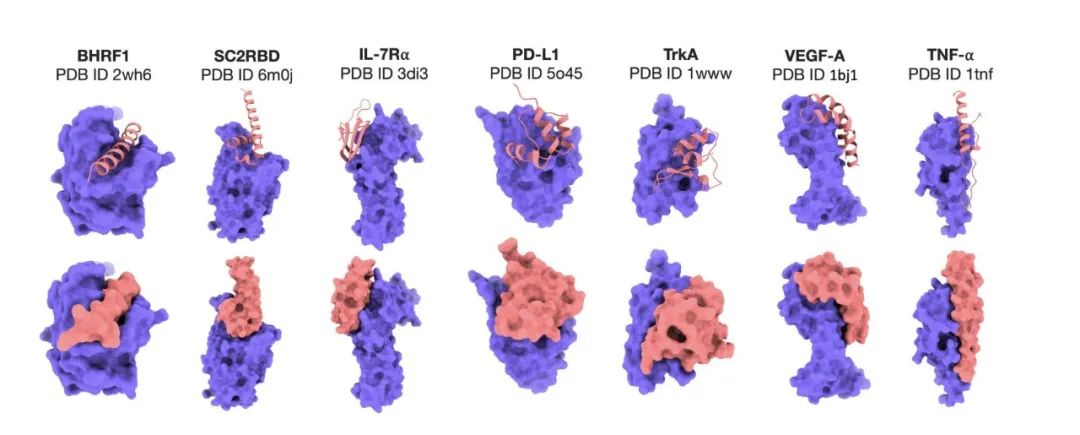
在 DNA 序列特异性结合剂生成实验中,Prot42 同样取得了显著成果。实验结果显示,通过结合基因嵌入和蛋白质嵌入的多模态策略,Prot42 模型能够生成与目标 DNA 序列特异性结合的蛋白质,并且展现出高度的亲和力,且通过 DeepPBS 模型评估的结合特异性较高。这些结果表明,Prot42 模型在 DNA 序列特异性结合剂生成方面也具有巨大潜力,为基因调控和基因组编辑应用提供了新的工具。
随着生物技术与人工智能的深度融合,蛋白质设计这一前沿领域正经历革命性变革。作为生命活动的核心执行者,蛋白质的结构与功能解析一直是科学研究的难点,而 AI 技术的介入,正加速破解这一复杂谜题,为新药研发、酶工程改造等场景开辟全新路径。
近些年,AI 技术再度突破,以生成式 AI 为核心的新技术正将蛋白质设计推向「创世纪」阶段。
美国密苏里大学许东教授团队提出了结构感知蛋白质语言感知模型(S-PLM),通过引入多视图对比学习,将蛋白质序列和 3D 结构信息对齐到统一的潜在空间中,利用 Swin Transformer 处理 AlphaFold 预测的结构信息,将其与基于 ESM2 的序列嵌入融合,从而创建了一个结构感知的 PLM,并在 Advanced Science 上发表文章「S-PLM: Structure-Aware Protein Language Model via Contrastive Learning Between Sequence and Structure」。 S-PLM 通过将蛋白质序列与其三维结构在统一的潜在空间中对齐,巧妙地将结构信息融入序列表示中,同时探索了高效微调策略,使模型能够在不同的蛋白质预测任务中取得卓越表现,标志着在蛋白质结构和功能预测领域的一个重要进步。
论文地址:
https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202404212
除此之外,清华大学研究团队等提出了一个统一的蛋白质语言模型 xTrimoPGLM,这是一个统一的预训练框架和基础模型,可扩展到 1,000 亿个参数,旨在用于各种蛋白质相关的任务,包括理解和生成 (或设计) 。通过利用通用语言模型(GLM)作为其双向注意力和自回归目标的骨干,该模型不同于先前的仅编码器或仅因果解码 PLMs 。该研究探索了超大规模 PLM 的统一理解和生成预训练,进一步揭示了蛋白质序列设计的新可能性,促进了蛋白质相关应用更广泛领域的进一步发展。以「xTrimoPGLM: unified 100-billion-parameter pretrained transformer for deciphering the language of proteins」为题,发表在 Nature 子刊。
论文地址:
https://www.nature.com/articles/s41592-025-02636-z
Prot42 的突破不仅是技术层面的进步,更映射着「数据驱动+AI 设计」模式在生命科学领域逐渐趋于成熟。未来,研究团队计划通过实验验证 Prot42 生成的结合剂,用实际功能测试补充计算评估。这一步将巩固模型在实际应用中的效用,并完善其预测准确性,弥合 AI 驱动序列生成与实验生物技术之间的差距。
参考资料:
1.https://arxiv.org/abs/2504.04453
2.https://mp.weixin.qq.com/s/SDUsXpAc8mONsQPkUx4cvA
3.https://mp.weixin.qq.com/s/x7_Wnws35Qzf3J0kBapBGQ
4.https://mp.weixin.qq.com/s/SDUsXpAc8mONsQPkUx4cvA
内容中包含的图片若涉及版权问题,请及时与我们联系删除

