过去十年间,得益于癌症基因组图谱(TCGA)和癌细胞系百科全书(CCLE)等计划的推动,癌症组学取得显著发展,特别是在肿瘤DNA和RNA层面,并生成了大量数据;但目前人们对肿瘤中蛋白质翻译及翻译后修饰(PTM)相关知识仍有待完善。反相蛋白质微阵列(RPPA)技术可对癌症样本进行大规模功能蛋白质组学数据分析,提供了对癌症机制的重要见解,有助于发现新型生物标志物和治疗靶点。
近日,美国德克萨斯大学MD安德森癌症中心梁晗团队利用RPPA技术,分析了8,000份TCGA患者肿瘤样本和900份CCLE细胞系样本,构建了一个全面、高质量的的泛癌功能蛋白质组学数据库RPPA500,包括近500种高质量抗体,涵盖所有主要癌症标志通路。为提高该资源可访问性和分析能力,同时推出了一个由先进大语言模型(LLMs)驱动的直观生物信息学平台DrBioRight 2.0,用户可通过自然语言探索以蛋白质为中心的癌症组学数据,执行高级分析、可视化结果并进行互动讨论交流。

研究团队首先构建了RPPA500数据集,共包含约9,000个样本。其中,TCGA队列有7,828个患者肿瘤样本,涉及32种癌症类型;CCLE队列涵盖878个癌细胞系。经筛选,RPPA500蛋白质集包含447种蛋白质标记,包括 357 种总蛋白和90种PTM蛋白,且高度富集治疗靶点和生物标志物。RPPA500蛋白质panel全面覆盖了50个标志性基因集;相较其他蛋白质panel,总蛋白数量增加115%,PTM蛋白数量增加67%,极大提升人们从蛋白质层面理解癌症生物学的能力。
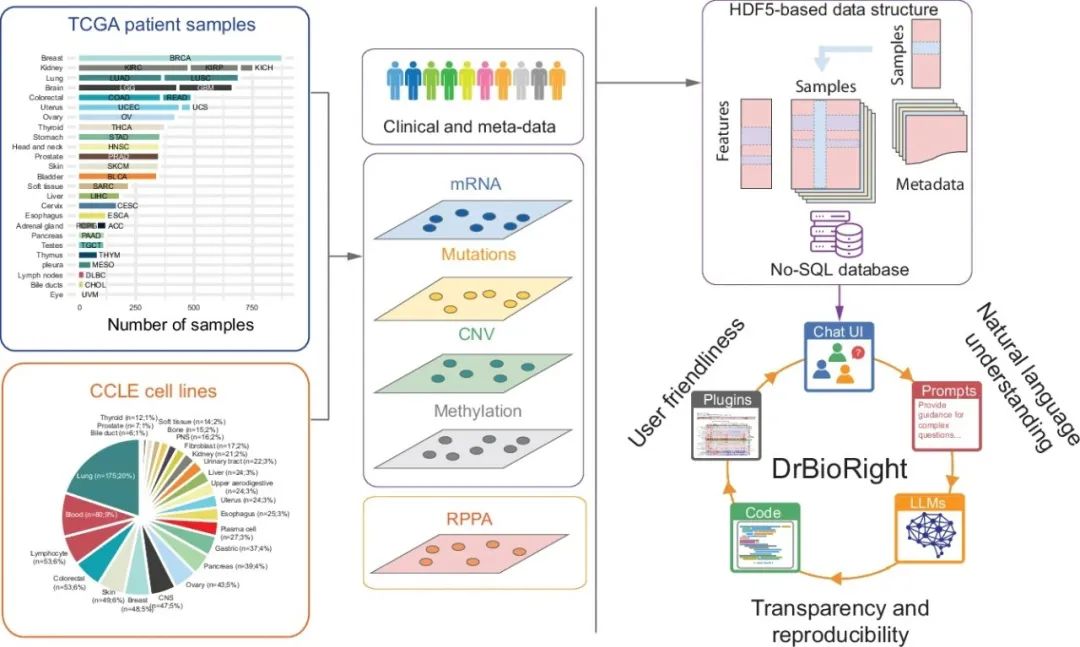
图1. DrBioRight 2.0中的数据集成工作流程和关键创新概述
同时,研究团队还开发了一个基于LLM的新型聊天机器人DrBioRight 2.0,其具备自然语言处理功能,使用户能够直观、智能地探索、分析和可视化上述RPPA数据。具体而言,研究团队首先生成了一个统一多组学数据集,对患者临床、分子层面、蛋白质层面及细胞系表型数据进行标准化、归一化处理,将超10亿数据值以HDF5 格式在云服务器非关系型数据库中整理、重构;然后重新审查了蛋白质标志物,通过交叉对比在不同层面对其进行详细注释,以利于用户分析。
与传统平台相比,DrBioRight 2.0拥有自然语言理解能力、透明度、可重复性以及用户友好性等功能,这些特殊功能由以下几项关键的前沿技术提供支持:①聊天界面:基于会话的实时聊天界面;②提示词:高度可定制的面向LLM的特定领域知识提示;③ LLM:由LLM赋能的生成式AI:④代码生成:无缝的代码生成与校正循环;⑤插件:深度嵌套的交互式插件增强了数据的有效可视化和分析。
使用DrBioRight 2.0时,用户输入相关指令,便可生成交互式热图、自动化进行相关性分析和生存分析等,且分析结果支持下载和本地复现。
图2. DrBioRight 2.0平台概述
DrBioRight 2.0的系统架构由No-SQL数据库、LLM驱动的后端分析模块和交互式聊天界面等三个核心部分组成。
DrBioRight 2.0创新点在于当用户开始一项分析时,只需选择一种疾病(如肺腺癌[LUAD]),系统便会自动将相关的多组学数据链接到用户的项目空间,以进行查询和分析;后端LLM可预测用户意图,区分是一般性查询或需要代码生成或生物信息学分析的深度查询;随后基于思维链输出逻辑流程辅助用户理解,同时即时生成基于文本答案或编程脚本;代码在提交前也会经过系统审查和验证,自动纠正常见错误,最后通过交互聊天界面显示结果。特别地,研究团队还集成了一个评分功能,通过用户反馈评分和专家手动评估,利用人类反馈强化学习(RLHF)实现LLM的持续迭代优化。
为提升DrBioRight 2.0性能,研究团队采用前沿技术增强LLM。运用多智能体(multi-agent)工作流程,借助图架构建立分层智能体团队,各团队由一个或多个智能体或工具组成,负责特定分析任务。此外,研究团队还通过专家评审整理、标准化数千个用户查询,创建训练和测试数据集分三阶段来微调LLM:①基于提示-响应进行初始监督微调;②开发评估系统量化专家评分,用评估数据训练奖励模型;③使用Hugging Face PPO策略优化强化学习。
研究团队通过独立测试集对多个平台进行了性能评估。结果显示,传统TCPA平台仅能处理26%的问题,GPT-4的成功率为58%,而经过微调的DrBioRight 2.0在相同问题上成功率高达90%,这凸显了融入领域特定知识、微调过程和多智能体工作流的重要性。值得注意的是,DrBioRight 2.0还对于大规模多组学数据的整合和存储进行了深度优化,确保数据的高效访问和处理,大大缩短了获取分析结果的时间,并能够适应不断变化的科研场景。
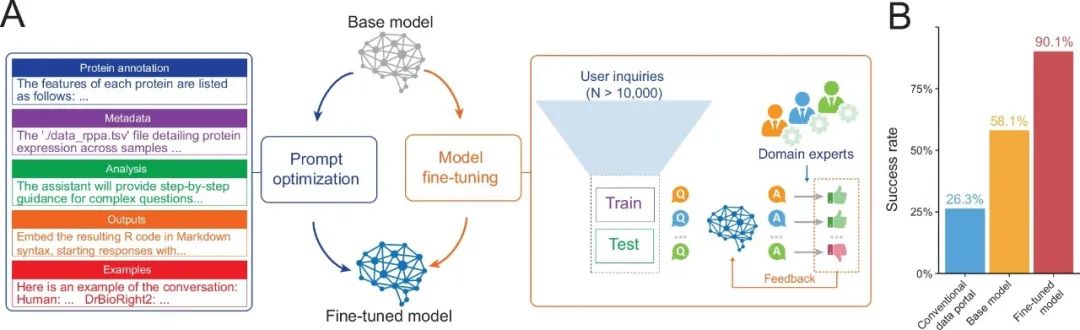
图3. 微调过程和模型评估概述
综上,DrBioRight 2.0是癌症蛋白质组学数据研究的重大进展,实现了三大里程碑:拓宽了蛋白质研究空间,为生物医学研究提供了独特资源;LLM赋能的智能交互平台可实现直观、多功能且高定制对话分析,降低使用门槛;数据与大语言模型深度融合,提升了资源效用,加速了用户与开发者之间的反馈循环。DrBioRight 2.0有望引领数据分析和共享平台发生突破性变革,可推动形成服务生物医学研究者的综合生态系统。
https://drbioright.org
参考文献:
Liu, W., Li, J., Tang, Y. et al. DrBioRight 2.0: an LLM-powered bioinformatics chatbot for large-scale cancer functional proteomics analysis. Nat Commun 16, 2256 (2025). https://doi.org/10.1038/s41467-025-57430-4
·END·
内容中包含的图片若涉及版权问题,请及时与我们联系删除

