
Simulating 500 million years of evolution with a language model
https://www.science.org/doi/10.1126/science.ads0018
摘要
30 多亿年的进化产生了编码在天然蛋白质空间中的生物图像。在这里,我们展示了在进化数据上进行大规模训练的语言模型可以生成远离已知蛋白质的功能性蛋白质。我们提出了 ESM3,这是一种前沿多模态生成语言模型,可以推理蛋白质的序列、结构和功能。ESM3 可以遵循结合其模态的复杂提示,并且对齐高度敏感以提高其保真度。我们已经促使 ESM3 生成荧光蛋白。在我们合成的几代中,我们发现一种明亮的荧光蛋白与已知荧光蛋白相距很远(58% 的序列同一性),我们估计这相当于模拟了 5 亿年的进化。
如今存在的蛋白质是在数十亿年的自然进化过程中发展成为现在的形式,经过了巨大的进化筛选。在地质时期进行的平行实验中,大自然创造了随机突变并进行选择,根据蛋白质的无数序列、结构和功能对其进行筛选。
因此,我们今天观察到的蛋白质模式反映了生物学中深层隐藏变量的作用,这些变量塑造了蛋白质随时间推移的进化。地球自然多样性的基因测序调查正在对蛋白质的序列(1-3)和结构(4、5)进行分类,其中包含数十亿个序列和数亿个结构,这些结构阐明了生命过程中的变化模式。人们正在形成一种共识,即这些序列背后是一种蛋白质生物学的基本语言,可以使用语言模型来理解(6-11)。
目前,已经开发和评估了许多蛋白质序列的语言模型(5-10、12-17 )。研究发现,语言模型中出现的表征反映了蛋白质的生物结构和功能(6-8、18 ),并且无需任何监督即可学习这些属性(19、20 ),并且随着规模的扩大而不断改进(5、21) 。在人工智能领域,已经发现了缩放定律,可以预测能力会随着规模的扩大而增长,描述了计算、参数和数据的前沿(22-24)。
这里,我们介绍了 ESM3,这是一种前沿的多模态生成模型,可以推理蛋白质的序列、结构和功能。ESM3 被训练为针对每种模态的离散标记的生成掩码语言模型。结构推理是通过将三维 (3D) 原子结构编码为离散标记来实现的,而不是使用最近预测 (25) 和蛋白质生成模型 (26-28) 中使用的复杂架构和 3D 空间中的扩散。离散标记的全对全建模是可扩展的,允许使用其模态的任意组合来提示 ESM3,从而实现可控制地生成尊重提示组合的蛋白质。我们观察到 ESM3 对提示反应灵敏,并能为复杂的提示组合找到创造性的解决方案,包括我们在自然界中找不到匹配结构的解决方案。所有规模的模型都可以对齐以更好地遵循提示,而较大的模型对齐的响应速度要快得多,显示出在对齐后解决最困难提示的更大能力。我们报告了利用 ESM3 生成的绿色荧光蛋白 (GFP) 变体 ( 29 , 30 ),其与现有蛋白质的差异程度相当于模拟了超过 5 亿年的进化。
ESM3
ESM3 通过语言建模实现了蛋白质三个基本属性(序列、结构和功能)的可扩展生成模型。之前对蛋白质的生成建模工作主要集中在单个模态上,利用复杂的架构和将蛋白质表示为 3D 对象的结构的训练目标。到目前为止,唯一已扩展的语言模型是蛋白质序列。在 ESM3 中,序列、结构和功能通过离散标记的字母表示。模态作为单独的序列轨道输入和输出,这些序列轨道融合到模型中的单个潜在空间中。这种简单性使 ESM3 能够利用可扩展的转换器架构来训练多达 980 亿个参数和超过一万亿次浮点运算的计算,展示了对序列、结构和功能的复杂推理能力的出现。
ESM3 采用生成掩蔽语言模型目标在其所有轨道上进行训练,如下式所示:
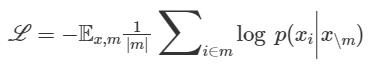
将随机掩码m应用于描述蛋白质的标记x,并对模型进行监督以预测被掩码的标记的身份。在训练期间,使用噪声计划对掩码进行采样,该计划会改变被掩码的位置的比例,以便 ESM3 可以看到掩码序列、结构和功能的许多不同组合,并能预测任何模态组合的完成情况。这与经典的掩码语言建模(31)不同,因为监督应用于所有可能的掩码率,而不是单一固定的掩码率。在给定任意先前标记组合的情况下,这种监督会将下一个标记的所有可能预测的概率分布分解为因式分解,从而确保可以从任何起点以任何顺序生成标记(32 – 34)。
要从 ESM3 生成,需要迭代采样 token。从完全或部分屏蔽的上下文开始,可以一次采样一个 token,也可以并行采样,并且以任何顺序采样,直到所有位置都完全不屏蔽(图 1A在图片查看器中打开)。除了支持生成之外,ESM3 的训练目标对于表征学习也非常有效。高掩蔽率可提高生成能力,而较低的掩蔽率可提高表征学习。我们选择使用噪声计划来训练 ESM3,以平衡生成能力和表征学习(补充材料,第 A.2.2 节)。
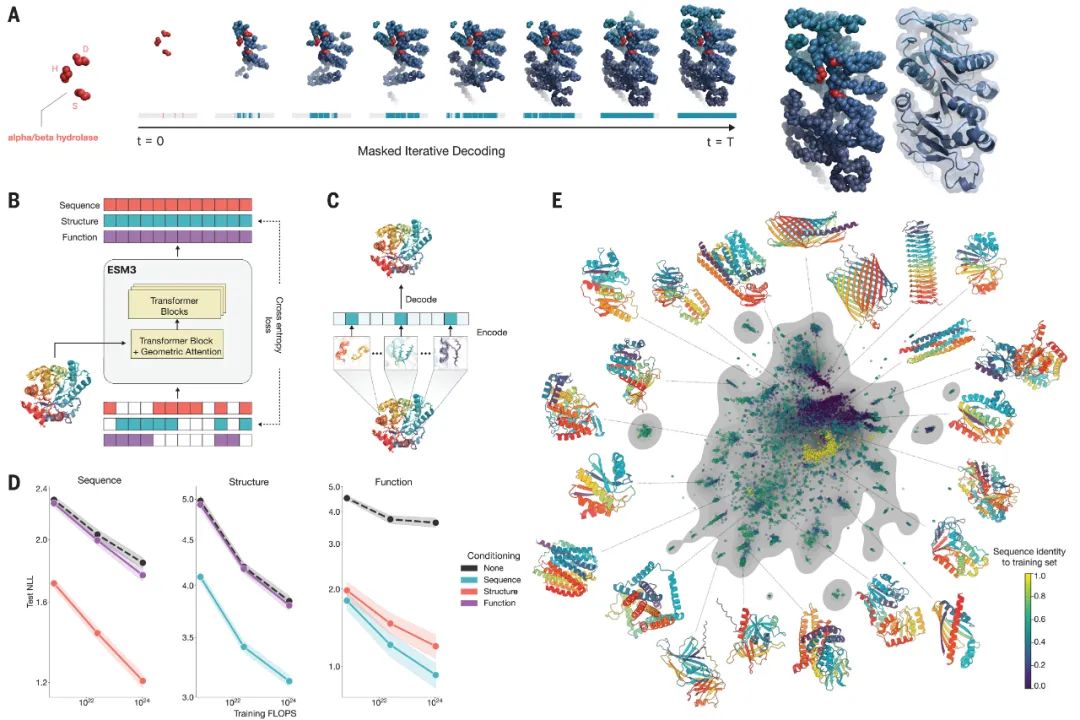
图 1 . ESM3 是一个推理蛋白质序列、结构和功能的生成语言模型。( A ) 使用 ESM3 进行迭代采样。生成 alpha/beta 水解酶。序列、结构和功能均可用于提示模型。在每个时间步 t,对一小部分掩码位置进行采样,直到所有位置均未掩码。( B ) ESM3 架构。序列、结构和功能表示为输入和输出处的离散标记轨道。该模型是一系列转换器块,其中所有轨道都融合在单个潜在空间内。第一个块中的几何注意力允许对原子坐标进行条件反射。ESM3 受到监督以预测掩码标记。( C ) 结构标记化。每个氨基酸周围的局部原子结构被编码为标记。( D ) 模型在三个规模上进行训练:1.4B、7B 和 98B 参数。测试集上的负对数似然(在掩码率上取平均值)作为训练 FLOP 的函数,表明对每个输入轨道的条件响应随着 FLOP 的增加而改善(95% 置信区间)。(E)来自 ESM3 98B 的无条件生成(按序列同一性着色为与训练集中最近的序列),由 ESM3 嵌入,并通过均匀流形近似和投影 (UMAP) 投影,以及来自 UniProt 的随机采样序列(灰色)。生成多样、高质量,并覆盖自然序列的分布。
ESM3 是一个双向转换器。序列、结构和功能标记在输入端嵌入和融合,然后通过一组转换器块进行处理(图 1B在图片查看器中打开)。在模型的输出端,浅层多层感知器头将最终层表示投影到每个轨道的标记概率中。ESM3 使用标记化,而不是专门的架构组件,来表示学习到的多模态特征空间中蛋白质的复杂性。这种方法可以实现高效且高度可扩展的训练。
蛋白质结构通过离散自动编码器( 35 )进行标记,该编码器经过训练可以将三维结构压缩为离散的标记(图 1C)在图片查看器中打开)。我们提出了一种不变的几何注意力机制来有效地处理 3D 结构。该机制在由每个氨基酸的键几何形状定义的局部参考框架中运行,并允许局部框架通过转换为全局框架进行全局交互(补充材料,第 A.1.6 节)。每个氨基酸周围的局部结构邻域被编码成一系列离散标记,每个氨基酸一个。
在预测或生成蛋白质结构时,ESM3 输出的结构标记会通过解码器,解码器会重建完整的原子结构。自动编码器经过训练,可以对原子坐标进行编码和重建,几何损失会监督键矢量和法线的成对距离和相对方向(补充材料,第 A.1.7.3.1 节)。这种标记化几乎完美地重建了蛋白质结构[使用 CAMEO 时,均方根差 (RMSD)
由于每个结构标记的局部邻域包含有关结构相邻部分的信息,我们还为模型提供了一种机制,通过第一个 Transformer 块中的几何注意直接以主链原子坐标为条件。为了支持更高级别的结构抽象,我们包含了二级结构 (SS8) 标记和溶剂可及表面积 (SASA) 标记的轨道。描述生物活性的关键词,如结合、酶功能以及域或折叠分类,可以对蛋白质结构和功能进行更高级别的语义描述。这些关键词源自 InterPro ( 36 ) 中的自由文本描述和基因本体 (GO) 术语中每个残基的单词,这些关键词被标记化(补充材料,第 A.1.8 节)、嵌入并在网络输入处求和。残基级注释为单个残基的功能提供多热标记,例如催化位点和翻译后修饰(补充材料,第 A.1.8.3 节)。
最大的 ESM3 模型基于从序列和结构数据库 ( 2 , 36 – 39 )收集的 27.8 亿个天然蛋白质进行训练。由于相对于序列,只有一小部分结构是通过实验确定的,因此我们利用了预测结构 ( 4 , 5 )。使用隐马尔可夫模型库 ( 40 ),用功能关键词注释序列。我们还使用逆折叠模型 (补充材料,第 A.2.1.3 节) 为所有结构(包括预测结构)生成了合成序列。总体而言,这将训练数据增加到 31.5 亿个蛋白质序列、2.36 亿个蛋白质结构和 5.39 亿个具有功能注释的蛋白质,总计 7710 亿个唯一标记。训练数据集的完整详细信息在补充材料第 A.2.1 节中描述。
我们训练了三种规模的 ESM3 模型:14 亿、70 亿和 980 亿个参数(分别为 14 亿、70 亿和 980 亿)。在评估表征学习性能对架构超参数的响应的初始系列实验中,我们发现对增加深度的响应大于对宽度的响应。这种行为决定了最终架构选择相对较深的网络,其中 980 亿参数模型包含 216 个转换器块(补充材料,第 A.1.5 节)。
将 ESM3 的参数从 1.4B 扩展到 98B 后,测试集上所有轨迹的损失都得到了显著改善,其中序列损失的改善最为显著(图 1D)在图片查看器中打开和图 S11)。无条件和条件负对数似然之间的差距随着规模的扩大而扩大。对功能关键词的条件化主要限制高掩蔽率下的序列,因此尽管在高掩蔽率下观察到对关键词条件化的响应,但在平均负对数似然中不太明显(图 S12)。测试损失的这些增加带来了更好的表征学习(表 S8 和图 S8)。在单序列结构预测中,ESM3 98B 超越了 ESMFold [CAMEO 测试集的平均局部距离差异测试 (LDDT) 为 0.880 对比 0.861;表 S9)。无需提示即可从模型生成序列(无条件生成)会产生高质量蛋白质,其平均预测 LDDT(pLDDT)为 0.84,预测模板建模得分(pTM)为 0.52,这些蛋白质在序列(平均成对序列同一性 0.155)和结构(平均成对 TM 得分 0.48)上都具有多样性,涵盖了已知蛋白质的分布(图 1E在图片查看器中打开和图S14)。
我们的结果表明,通过语言建模(由标记化、高效架构和掩码标记预测实现)进行扩展,可以持续改进表征和生成应用程序。这种方法允许模型构建一个共享的多模态表征空间,该空间是从数据中学习而来的,而不是明确地硬编码到其架构中。随着计算和数据的增加,该模型可以学习越来越丰富和更通用的特征空间。在以下部分中,我们将展示这种方法如何实现蛋白质可控生成的高保真度。
采用 ESM3 的可编程设计
我们探索了 ESM3 遵循具有不同组成的复杂提示的能力。ESM3 可以使用来自其每个输入轨道的指令进行提示:序列、结构坐标、SS8、SASA 和功能关键词。这允许在多个抽象级别上指定提示,从原子级结构到描述功能和折叠拓扑的高级关键词。
我们评估了 ESM3 在每个音轨中独立遵循提示的能力(图 2A在图片查看器中打开)。使用时间上保留的天然蛋白质测试集(补充材料,第 A.3.8 节)为每条轨道构建一组提示。使用 ESMFold 评估所得代与提示的一致性和结构预测置信度 (pTM)。我们为每个轨道定义了四个一致性指标:(i)约束位点 RMSD (cRMSD),提示坐标(即主链原子的位置)与代中相应坐标之间的 RMSD;(ii)SS3 准确度,提示和代之间的三类二级结构匹配的残基分数;(iii)SASA Spearman ρ,SASA 提示与代相应区域之间的相关性;(iv)关键词恢复,即 InterProScan 恢复的提示关键词分数 ( 40 )。在所有轨道中,7B 参数 ESM3 找到遵循提示且具有 ESMFold 自信预测的结构的解决方案(pTM > 0.8)。观察到一些模式转换,包括在关键词提示下,其中一小部分代人有信心预测无法恢复关键词的结构。
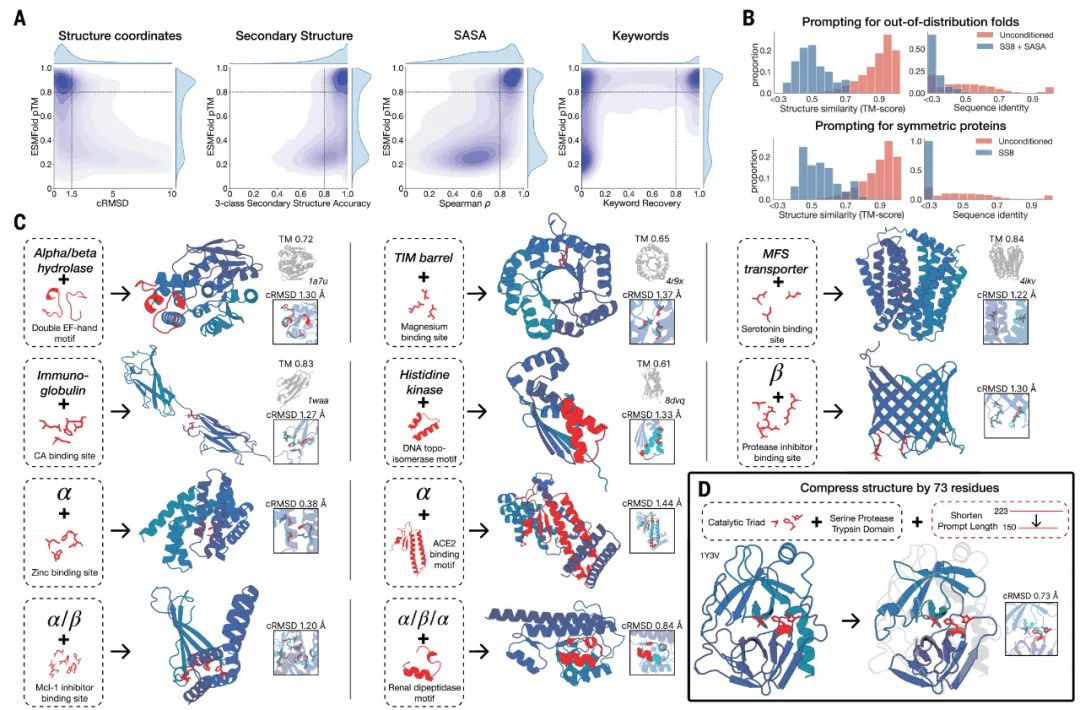
图 2.使用 ESM3 进行生成编程。( A ) ESM3 可以遵循来自其每个输入轨道的提示。显示了每个轨道的提示忠实度密度。生成与提示保持一致(主链 cRMSD、SS3 准确度、SASA Spearman ρ 和关键词恢复)并且具有高结构预测置信度 (pTM)。( B ) 可以提示 ESM3 生成与训练集和天然蛋白质在结构(左)和序列(右)上不同的蛋白质。在响应源自分布外的天然结构(顶部)和计算设计的对称蛋白质(底部)的提示时,提示的生成(蓝色)与无条件生成(红色)相比转向更新颖的空间。( C ) ESM3 为各种复杂提示组合生成创造性的解决方案。我们展示了原子级基序与通过关键词或二级结构提示指定的高级指令的组合。对提示的保真度通过与参考结构的相似性(对于关键词提示)和全原子 RMSD(对于基序提示)来显示。解决方案与基序提示来源的支架不同(中位 TM 得分 0.36 ± 0.14),对于许多基序(例如,血清素、钙、蛋白酶抑制剂和 Mcl-1 抑制剂结合位点),我们找不到与包含相同基序的其他蛋白质的显著相似性。(D)特别有创意行为的例子。ESM3 将丝氨酸蛋白酶压缩 33%,同时保持活性位点结构。
无条件生成反映了天然蛋白质的分布。由于我们观察到 ESM3 可以忠实地遵循提示,我们推断提示可以引导模型生成不同于训练集和天然蛋白质的蛋白质。首先,我们测试了模型遵循分布外提示的能力。我们从保留结构(TM 0.8,pLDDT > 0.8),与训练集中的蛋白质具有较低的序列和结构相似性(序列同一性
ESM3 能够遵循复杂的提示,并能够从不同的轨道和不同的抽象层次编写提示。为了评估这种能力,我们用需要解决单个原子空间协调的基序来提示 ESM3,包括参与序列中相距较远的残基之间的三级接触的原子,例如催化中心和配体结合位点。我们将原子级基序提示与高级提示相结合,高级提示可以是二级结构提示或指定折叠结构的关键词提示。对于原子级基序和高级提示的每种独特组合,我们都会生成序列,直到成功(对于原子级提示,当所有原子 RMSD 0.6 到代表性结构时;对于二级结构提示,当 SS3 准确度 >80% 时;对于所有提示,当整个生成的蛋白质的 pTM >0.8 和 pLDDT >0.8 时)。
我们发现 ESM3 能够解决各种各样的此类任务(图 2C在图片查看器中打开)。它无需检索基序的原始支架即可完成此操作(中位 TM 得分为 0.40 ± 0.10;补充材料,第 A.3.10 节)。在某些情况下,支架是从具有相似基序的现有蛋白质转移而来的(例如,ESM3 设计的锌结合基序的 α 螺旋支架与 Ni 2+结合蛋白具有很高的相似性,PDB:5DQW、5DQY;图 2C在图片查看器中打开,第 3 行,第 1 列)。对于许多基序(例如,血清素、钙、蛋白酶抑制剂和 Mcl-1 抑制剂的结合位点),Foldseek(41)找不到其他含有相同基序的蛋白质。在这些情况下,我们观察到有时基序被移植到完全不同的折叠中(例如,β-桶中的蛋白酶抑制剂结合位点基序与膜结合铜转运体最相似,PDB:7PGE;图 2C在图片查看器中打开,第 3 行,第 3 列)。在其他时候,支架与 PDB、ESMAtlas 和 AlphaFold 数据库中所有已知蛋白质的结构相似性较低(最大 TM 得分
通过提示工程实验,我们观察到了对提示的特别有创意的反应。在这里,我们重点介绍了蛋白质压缩的一个例子(图 2D在图片查看器中打开)。从天然胰蛋白酶 (PDB 1Y3V) 开始,我们提示了催化三联体的序列和坐标以及描述胰蛋白酶的功能关键词,但将总代长减少了三分之一(从 223 个残基减少到 150 个残基)。ESM3 设计保持了活性位点的协调性(全原子 RMSD 0.73 Å)和整体折叠,具有很高的可设计性(pTM 0.84,scTM 平均值 0.97,SD 0.006),尽管序列长度大大减少,并且折叠仅由功能关键词提示指定(补充材料,第 A.3.11 节)。
这些示例说明了 ESM3 能够为其输入轨道中指定的任何提示(单独或组合)找到创造性的解决方案。此功能使蛋白质设计成为一种合理的方法,提供从高级拓扑到原子坐标等各种抽象级别的控制,使用生成模型弥合提示与生物复杂性之间的差距。
生物对齐
尽管我们已经观察到基础模型的性能随着规模的扩大而显著提升,但更大的模型可能具有我们未观察到的更大的潜在能力。尽管基础 ESM3 模型尚未针对这些目标进行明确优化,但可以提示它们执行困难的任务,例如三级主题支架和提示组合。由于我们评估生成输出的属性(例如对提示的遵守程度或支架的置信度)在预训练期间只能由模型间接看到,因此通过微调将模型直接与生成任务对齐可能会导致与更大模型之间更大的能力差异。
我们研究了如何对齐基础模型(43、44 )以生成满足具有挑战性的提示的蛋白质。对于每个模型,我们构建了一个主链原子坐标提示数据集,该数据集由连续的残基跨度和三级基序组成(也指定了接触氨基酸的身份)。我们为每个提示生成了多个蛋白质序列,并使用 ESM3 折叠每个序列,对与提示的一致性(主链 cRMSD)和结构预测置信度 (pTM) 进行评分。将高质量样本与同一提示的低质量样本配对,以构建偏好数据集(补充材料,第 A.4 节)。然后使用偏好优化损失对 ESM3 进行微调(45、46 ),这会导致模型对高质量样本的可能性高于对低质量样本的可能性。
在对齐每个基础模型后,我们评估了它们的绝对性能和代数分布的变化。我们专注于一系列具有挑战性的提示,这些提示需要协调三级接触中残基的主链原子。我们使用 ESMFold 来评估生成高质量支架(pTM >0.8)的能力,这些支架遵循高分辨率(主链 cRMSD
与基础模型相比,对齐模型解决了两倍的三级协调任务(图 3A在图片查看器中打开虽然基础模型在任务解决百分比上存在差异(1.4B 为 9.5%,7B 为 19.0%,98B 为 26.8%;图 3A在图片查看器中打开),通过比对发现能力差异更大(1.4B、7B 和 98B 模型分别从 9.5% 增加到 18.8%、19.0% 增加到 37.4% 和 26.8% 增加到 65.5%)。偏好调整的模型不仅可以解决更大比例的任务,而且可以为每个任务找到更多的解决方案,以具有主链 cRMSD 0.8 的不同结构簇 (TM >0.8) 的数量来评估(图 3B)在图片查看器中打开)观察到每个配体结合基序的 ESMFold pTM 和骨架 cRMSD 分布发生了变化(图 3C在图片查看器中打开和图 S18)。在 98B 尺度上,微调模型在 46 种测试配体中的 37 种上产生了比基础模型更明显的成功聚类,而其余 9 种配体既不能通过基础模型也不能通过对齐模型解决,这表明对齐几乎普遍提高了对生成蛋白质结构预测的提示和置信度的忠实度。这些结果代表了最先进的基序支架性能(表 S16)。与仅最大化正例可能性的监督微调基线相比,偏好调整在所有尺度上都带来了更大的改进(补充材料,第 A.4.6 节)。
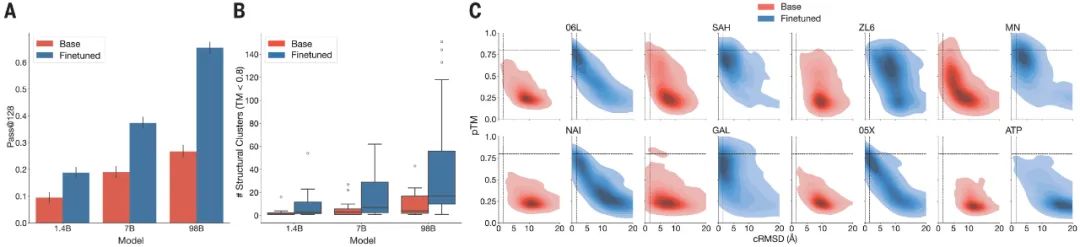
图 3.通过对齐,解决复杂任务的能力随着规模的扩大而提高。ESM3 与由提示代构建的偏好对数据集对齐,以遵循三级协调提示,其中具有所需属性高分数(高 pTM、低 cRMSD)的正样本与具有较差分数的负样本配对。偏好调整损失鼓励模型将更高的可能性放在正样本上。训练后,通过提示三级接触残基的主链原子坐标来评估模型。(A)微调对使用 128 代解决的任务比例的影响(Pass@128;误差线表示 2 个 SD)。随着规模的扩大,模型之间的差距很大。对齐的响应显示了在最大模型中解决复杂任务的潜在能力。(B)为每个三级基序生成的不同解决方案的数量(聚集在 TM > 0.8)。微调后,对于成功的配体,通常会有许多独特的解决方案。 ( C ) 显示了基础模型(左)和对齐模型(右)在 98B 规模下针对大量随机选择的配体所生成的提示代密度。对齐后,提示保真度(主链 cRMSD)和生成质量 (pTM) 趋于大幅提高。
我们对对齐的实验表明,不同模型规模之间的能力存在相当大的差异。与对齐前的基础模型相比,最大的对齐模型得到了显着改进,与对齐后较小的模型相比也是如此。通过对齐,模型学会从少量示例中进行概括;代数分布发生变化,以提高支架的质量和与提示的一致性,从而增加解决的任务比例和不同解决方案的数量。
对齐要求模型通过示例进行学习。能够识别微调示例所说明的底层属性,并将这些示例推广到新任务,意味着微调访问的属性具有内部表示。这个表示空间是通过预训练过程学习的,在此过程中,模型在进化过程中对蛋白质进行训练,这表明它反映并包含了蛋白质生物学的多样性和复杂性。这样的表示空间可能包含支持许多生物学特性推广的特征。较大模型对对齐的响应性更强,这表明它们的内部表示空间更好地近似了这些底层属性,这表明通过预训练中学习的特征进行迁移的能力很强,并且随着规模的扩大而增强。
产生远距离荧光蛋白
我们试图了解基础预训练的 ESM3 模型是否具有足够的生物保真度来生成功能性蛋白质。我们着手创建一个与现有序列相似性较低的功能性 GFP。我们选择荧光功能是因为它很难实现、易于测量,并且是自然界中最美丽的机制之一。
GFP 家族中的蛋白质负责水母的荧光和珊瑚的鲜艳色彩(47),并且它们的独特之处在于无需辅因子或底物即可形成荧光发色团(30)。这一特性使 GFP 序列可以插入其他生物体的基因组中,以可见的方式标记分子、细胞结构或过程,从而提供广泛应用于整个生物科学领域的基础工具包。
GFP 家族是数十年来蛋白质工程研究的主题,但迄今为止,已知的 GFP 序列多样性绝大部分来自于对自然界的探索,因为蛋白质工程研究大多只探索了天然荧光序列中的少数突变。合理的设计和诱变已经产生了具有改良特性的 GFP 序列,例如更高的亮度或稳定性或不同颜色的变体,这些序列融合了少量突变(通常是总共 238 个氨基酸编码序列中的 5 到 15 个)。在少数情况下,利用高通量实验和机器学习,科学家已经能够引入多达 40 到 50 个突变(即 80% 的序列同一性)同时保留荧光(48 – 50)。
要生成一种与天然变体序列距离较远的工程 GFP,需要实现其荧光背后的复杂生物化学和物理学。在所有 GFP 中,自催化过程会从蛋白质核心中的三个关键氨基酸形成发色团。GFP 的结构是弯曲的中央 α 螺旋,周围环绕着 11 链 β 桶,其中有向内的配位残基,这使该反应成为可能 ( 51 )。一旦形成,发色团不仅必须吸收光,还必须发射光才能发出荧光。光发射对发色团的局部电子环境高度敏感。GFP 的适应度景观反映了活性位点和实现其功能所需的周围三级相互作用的精确配置,因为一些随机突变就足以将荧光降至零( 48,52 )。
为了生成 GFP 序列,我们直接提示碱基预训练的 7B 参数 ESM3 生成一个 229 个残基的蛋白质,该蛋白质以 Thr 62、Thr 65、Tyr 66、Gly 67、Arg 96、Glu 222的位置为条件,这些位置是生成发色团的关键残基(图 4A)在图片查看器中打开)。我们还对 1QY3 实验结构中残基 58 至 71 的结构进行了条件化,这些残基的结构对于发色团形成的能量有利性具有结构上的重要意义 ( 53 )。具体来说,在输入时提供序列标记、结构标记和骨架的原子坐标,并且从对应于 229 个残基的几乎完全屏蔽的标记阵列开始生成,用于条件化的标记位置除外。
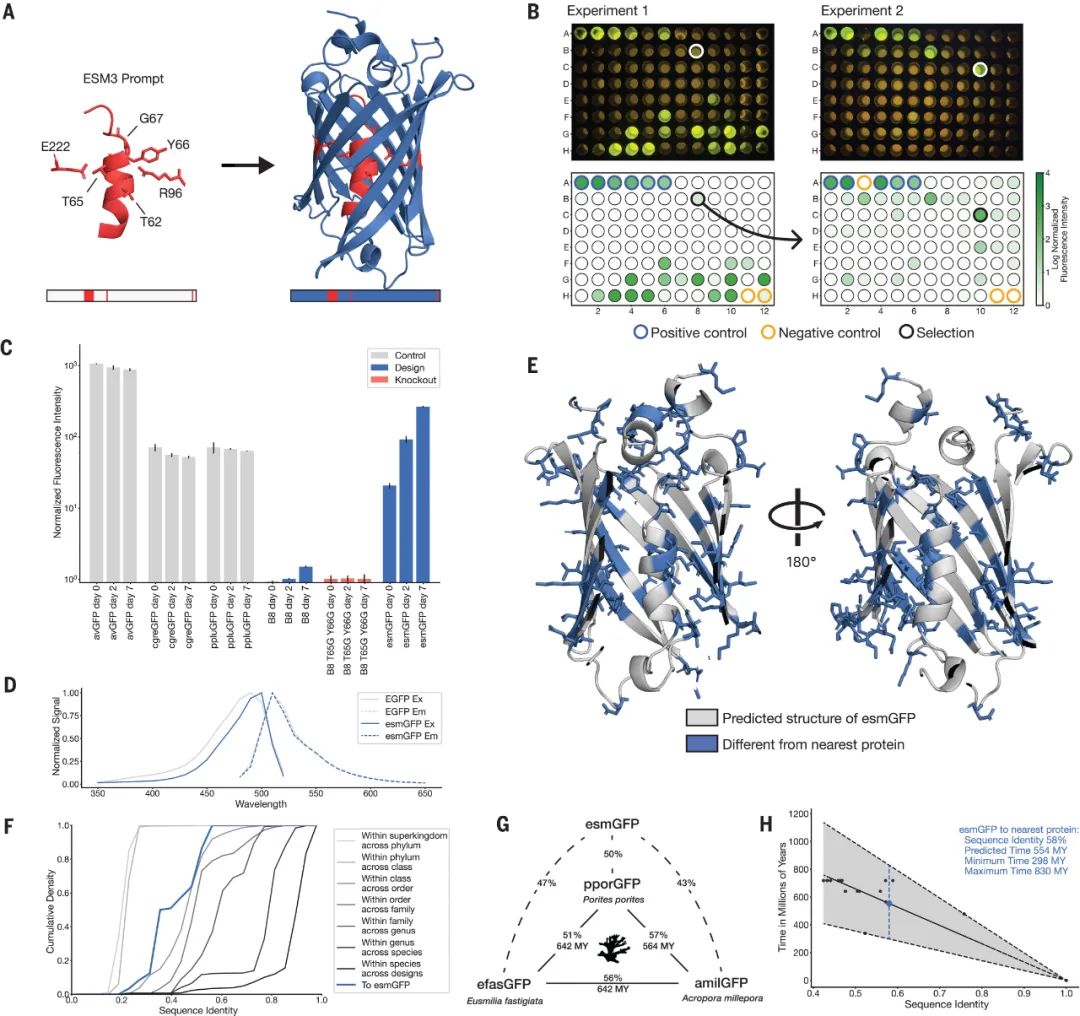
图4.利用思路链生成远距离荧光蛋白。( A ) 我们向 ESM3 提示了形成和催化发色团反应所需的残基序列和结构,以及天然 GFP 中央 α 螺旋部分的结构(左)。通过一系列思路,ESM3 生成设计候选(右)。( B ) ESM3 在两个实验中发现了一个与其他已知 GFP 相距甚远的明亮 GFP。我们测量了大肠杆菌裂解液中的荧光。上行,板的照片。下行,板读取器荧光定量。已知 GFP 的阳性对照用蓝色圆圈标记,没有 GFP 序列或没有大肠杆菌的阴性对照用橙色圆圈标记。在第一个实验(左)中,我们表达了具有一系列序列同一性的设计。在标记良好的 B8 中出现了一个与已知荧光蛋白具有低序列同一性(57%)的显著设计(底部用黑色圆圈突出显示,顶部用白色圆圈突出显示)。我们继续从 B8 中的蛋白质开始的思路,进行第二个实验(右)。标记良好的 C10(与已知荧光蛋白的序列同一性为 58%;同样,在底部用黑色圆圈突出显示,在顶部用白色圆圈突出显示)中出现了明亮的图案,我们将其命名为 esmGFP。(C)esmGFP 表现出与常见 GFP 相似的荧光强度。显示了实验 2 中一组蛋白质的归一化荧光。(D)esmGFP 的激发和发射光谱与 EGFP 的光谱叠加。(E)esmGFP 预测结构的中央 α 螺旋和 β 桶内部的两个剪切视图。esmGFP 相对于其最近邻居 tagRFP 具有的 96 个突变以蓝色显示。(F)跨分类群的荧光蛋白之间序列同一性的累积密度。esmGFP 与所有其他 FP 的相似度通常出现在跨目但在同一类内比较序列时。 ( G ) 以百万年 (MY) 为单位的进化距离和三种示例珊瑚虫 GFP 和 esmGFP 的序列同一性。( H ) 根据 GFP 序列同一性估计以百万年 (MY) 为单位的进化距离。我们估计 esmGFP 与最接近的已知蛋白质相隔 5 亿多年的自然进化。
我们使用以下思路链程序生成设计。该模型首先生成结构标记,有效地创建蛋白质主链。具有足够好的活性位点原子协调但整体结构与 1QY3 主链不同的主链通过过滤器进入链的下一步。我们将生成的结构添加到原始提示中,以生成以新提示为条件的序列。然后,我们执行迭代联合优化,交替优化序列和结构。我们拒绝了失去活性位点原子协调的思路链(补充材料,第 A.5.1 节)。我们从生成协议迭代联合优化阶段的中间点和最终点抽取了数万个候选 GFP 设计的计算池。我们根据与已知荧光蛋白的序列相似性对设计进行分类,并使用各种指标对设计进行筛选和排名(补充材料,第 A.5.1.5 节)。
我们在 96 孔板上进行了第一次实验,共 88 个设计,评估了每个序列相似性桶中的前几代。合成每个生成的蛋白质,在大肠杆菌中表达,并在 485 nm 的激发波长下测量荧光活性(图 4B)在图片查看器中打开,左图)。我们测量了与阳性对照相似的亮度,这些亮度来自许多与自然产生的 GFP 具有更高序列同一性的设计。我们还在 B8 孔中发现了一个设计(以黑色圆圈突出显示),它与 1QY3 序列的序列同一性仅为 36%,与最接近的现有荧光蛋白 tagRFP 的序列同一性为 57%。这种设计的亮度比天然 GFP 低 50 倍,其发色团在一周内成熟,而不是在一天之内,但它在序列空间的一部分中呈现出功能信号,据我们所知,这种信号在自然界或通过蛋白质工程尚未发现。
我们继续思路,从 B8 孔中的设计序列开始,使用与上述相同的迭代联合优化和排序程序生成亮度更高的蛋白质。我们创建了第二个 96 孔板的设计,并使用相同的板读取器分析,我们发现该组中的一些设计的亮度在自然界中发现的 GFP 范围内。最佳设计位于第二块板的 C10 孔中(图 4B)在图片查看器中打开,右),我们将其指定为 esmGFP。
我们发现 esmGFP 在天然 GFP 的分布中表现出亮度。我们评估了发色团成熟的 0、2 和 7 天时的荧光强度,并绘制了 esmGFP、B8 的重复、B8 的发色团敲除以及三种天然 GFP:avGFP、cgreGFP 和 ppluGFP 的这些测量值(图 4C)在图片查看器中打开)。esmGFP 的成熟时间比我们测量的已知 GFP 更长,但在 2 天后就达到了相当的亮度。为了验证荧光是由预期的 Thr 65和 Tyr 66介导的,我们表明,这些残基突变为甘氨酸的 B8 和 esmGFP 变体失去了荧光活性(图 S22)。
对 esmGFP 的激发和发射光谱的分析表明,它的峰值激发发生在 496 nm,相对于 EGFP 的 489 nm 峰值偏移了 7 nm,但两种蛋白质的发射峰值均为 512 nm(图 4D)在图片查看器中打开)。光谱形状表明,esmGFP 的激发光谱的半峰全宽 (FWHM) 较窄(esmGFP 为 39 mm,EGFP 为 56 nm),而它们的发射光谱的 FWHM 非常相似(分别为 35 和 39 nm)。总体而言,esmGFP 表现出与已知 GFP 一致的光谱特性。
接下来,我们试图了解 esmGFP 与已知蛋白质的比较情况。对非冗余蛋白质序列数据库进行BLAST ( 54 ) 搜索和对 ESM3 训练集进行 MMseqs ( 55 ) 搜索均报告了相同的最高匹配值 tagRFP,它也是 B8 的最近邻居,具有 58% 的序列同一性,代表整个序列中有 96 个突变。tagRFP 是一种设计变体,自然界中与 esmGFP 最接近的野生型序列是 eqFP578,这是一种红色荧光蛋白,与 esmGFP 有 107 个序列位置(53% 同一性)的差异。esmGFP 和 tagRFP 之间的序列差异发生在整个结构中(图 4E在图片查看器中打开),其中 22 个突变发生在蛋白质内部,由于发色团接近且相互作用密度高,蛋白质内部对突变高度敏感 ( 56 )。
对 648 种天然和设计的 GFP 样荧光蛋白序列比对的检查表明,esmGFP 与所有其他 FP 具有相似性,这种相似性通常在跨分类目但在同一分类类别内比较序列时发现(图 4F在图片查看器中打开)。例如,esmGFP 与其他 FP 之间的差异与属于石珊瑚目 (Scleractinia) 和海葵目 (Actiniaria) 的 FP 之间的差异程度相似,这两个目都属于海洋无脊椎动物中较大的珊瑚纲 (Anthozoa)(图 4G)在图片查看器中打开与 esmGFP 最接近的 FP 来自珊瑚虫纲(珊瑚和海葵;平均序列同一性 51.4%),但 esmGFP 也与水螅纲(水母)中的 FP 具有一定的序列同一性,在水螅纲中发现了 avGFP(平均序列同一性 33.4%;图 S23)。
我们可以从进化生物学中了解到,具有相似序列同一性的蛋白质在自然进化中出现需要多长时间。在图 4G中在图片查看器中打开,我们将 esmGFP 与三种珊瑚虫 GFP 一起展示。我们使用了珊瑚虫的时间校准系统发育分析(57),估计了数百万年前(MYA)到最后共同祖先的时间,以估计这些物种中每对之间的进化时间。使用一个更大的数据集,其中包含六种珊瑚虫 GFP 和物种,我们拥有准确的 MYA 到最后共同祖先和 GFP 序列身份,我们构建了一个简单的估计器,将 FP 之间的序列身份与物种之间的进化时间 MY 相关联(图 4H在图片查看器中打开) 来校准自然进化。基于此分析,我们估计 esmGFP 相当于从自然界中发现的最接近的蛋白质进化了 5 亿年。
讨论
我们发现,语言模型可以达到与自然进化探索的空间相去甚远的蛋白质设计空间,并且可以生成需要进化数亿年才能发现的功能性蛋白质。蛋白质语言模型并不明确地在进化的物理约束范围内工作,而是可以隐式地构建进化可能遵循的众多潜在路径的模型。
蛋白质可以看作存在于一个有组织的空间中,其中每个蛋白质都与每个相距一个突变事件的其他蛋白质相邻(58)。进化的结构表现为这个空间内的网络,通过进化可以在它们之间采取的路径将所有蛋白质连接起来。进化可以遵循的路径是每个蛋白质转变为下一个蛋白质而不会导致其所属系统整体功能丧失的路径。
语言模型正是在这个空间中看到蛋白质。它认为蛋白质的数据填充了这个空间,在某些区域密集,在其他区域稀疏,揭示了进化可以到达的部分。由于下一个标记是由进化产生的,因此,为了解决预测下一个标记的训练任务,语言模型必须预测进化如何穿过可能的蛋白质空间。
模拟是现实的计算表示。从这个意义上说,可以预测进化可能结果的语言模型可以说是进化的模拟器。ESM3 是一个新兴的模拟器,它通过解决进化产生的数据的标记预测任务来学习。理论上,神经网络可以发现它们被训练来预测的数据的底层结构(59、60 ) 。这样,解决标记预测任务将需要模型学习决定进化可以采取哪些步骤的深层结构,即蛋白质的基本生物学。
在 ESM3 生成荧光蛋白的过程中,最有趣的是 B8 的第一个思路。在 B8 最近的邻居中,有 96 个突变,
可能的蛋白质,其中只有极小的一部分可以发挥作用,因为即使经过几次随机突变,荧光也会急剧下降。B8 附近 C10 和其他明亮设计的存在证实了在对 B8 的第一个思路中,ESM3 发现了蛋白质空间的一部分,尽管大自然尚未探索过,但却充满了荧光蛋白。



内容中包含的图片若涉及版权问题,请及时与我们联系删除

