DRUGAI
人工神经网络为非生物信息处理提供了强大的范式。为了探究类似的原理是否能够在活细胞内实现计算,研究人员结合全新设计的蛋白质异源二聚体和工程化的病毒蛋白酶,构建了一个执行“胜者为王(winner-take-all)”神经网络分类的合成蛋白质电路。该“Perceptein”电路通过可逆结合相互作用实现加权输入求和,并通过不可逆的蛋白水解切割实现自激活和相互抑制。这些相互作用共同产生了丰富的蛋白质种类,由多达八种共表达的起始蛋白质衍生而来。完整系统在哺乳动物细胞中实现了具有可调决策边界的多输出信号分类,并可用于有条件地调控细胞死亡。这些结果展示了如何通过工程化蛋白质网络,实现活细胞内的可编程信号分类。

细胞是天然的分类机器。它们利用相互作用的基因和蛋白质电路,根据多种输入信号的水平或动态做出截然不同的决策。例如,p53肿瘤抑制通路能够对细胞所遇到的压力类型和程度进行分类,并诱导细胞衰老或凋亡。在发育过程中,神经管中的细胞通过分类骨形态发生蛋白(BMP)和Hedgehog信号的振幅,确定特定的前体细胞命运。在免疫系统中,多个细胞因子的输入分类可以调控T细胞的命运。实现合成信号分类系统的可编程化,能够帮助工程化基因和细胞疗法,让细胞区分正常与疾病状态。合成生物学的一个重要目标是设计分类电路,以根据不同类型的输入信号控制细胞的输出。
胜者为王(winner-take-all)的神经网络是一种强大的分类架构。在这些系统中,当且仅当某个输出节点的加权输入总和超过输出层中所有其他节点时,该节点才会被激活。这种架构提供了一种紧凑的信号分类机制,只需单层神经网络即可实现;确保输出呈现全或无的特性;并允许调控决策边界——即在输入空间中划分产生不同输出的分界线。
理论研究提出了一些关于工程化生化神经计算系统的方案。实验上,构建合成分类系统的努力已经取得了一些成果,例如在试管中实现了基于DNA或酶的分类器,以及在细胞中实现了基于microRNA(miRNA)或基因的分类器。相比之下,蛋白质水平的分类器具有多个优势:它可以在细胞内瞬时表达,直接与内源性输入和输出相互作用,避免转录延迟;此外,它不依赖内源性转录调控,能够在多种细胞类型中发挥作用。从更广泛的角度来看,构建基于蛋白质的神经网络将证明,通过相互作用的蛋白质,可以实现复杂的计算系统。
合成蛋白质生物学的两大进展为设计蛋白质水平的神经网络提供了可能性。首先,一系列模块化结合域(包括全新设计的异源二聚体,DHDs)使得蛋白质相互作用的可编程控制成为可能。其次,这些相互作用可以用于重组分裂的病毒蛋白酶,从而实现对其活性的调控。利用这些技术进展,研究人员设计了基于DHDs和分裂蛋白酶的胜者为王电路,并证明这些电路能够在活的哺乳动物细胞中对输入蛋白质的相对丰度进行分类。
结果
系统设计
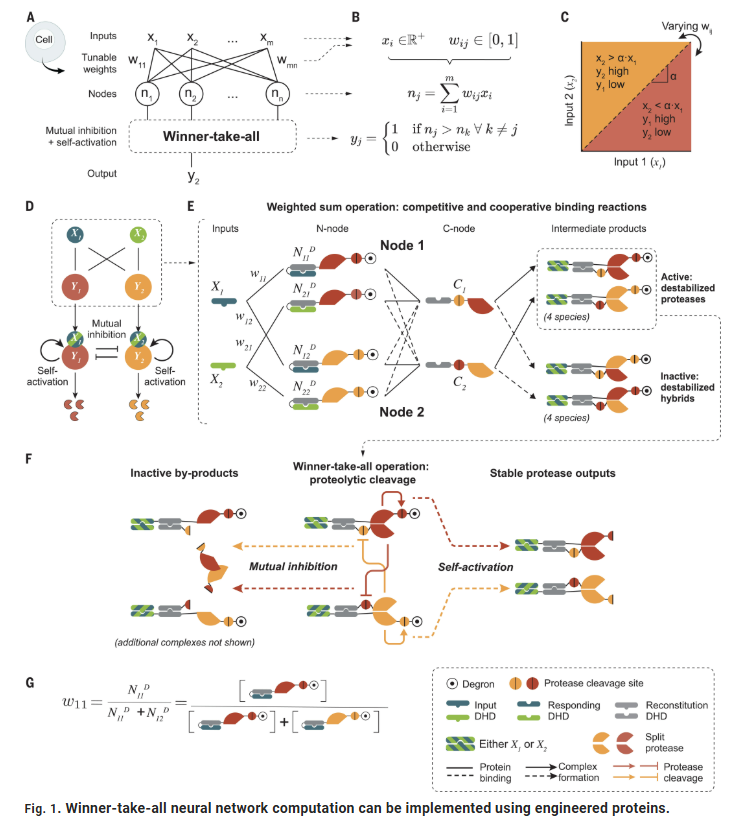
三种关键电路特性使“胜者为王”动态得以实现:加权输入求和使神经网络中的每个节点能够以可调强度响应输入分子;节点间的相互抑制消除丰度较低的物种;节点的自激活允许存活的物种放大并维持自身活性。
研究人员设计了一套嵌合蛋白来提供这些特性。作为输入,构建了一系列“输入DHD”单体。这些输入与称为N节点的蛋白结合并激活它们,N节点包含N端蛋白酶半体。激活后,N节点与包含C端蛋白酶半体的C节点结合,形成功能性或非功能性蛋白酶,后者通过相互抑制机制发挥作用。所有节点蛋白共享相同的重组域,以确保类似的蛋白酶重组动力学。
该设计实现了输入的加权求和。每种输入物种可同时与多个N节点亚基结合,输入蛋白根据N节点的相对丰度分配,输入加权通过N节点蛋白的丰度比例进行调控,最终实现总加权求和。
电路设计还通过蛋白降解和蛋白酶切割实现了自激活与相互抑制。我们在N节点蛋白上融合了可降解域(DHFR degron),在没有输入的情况下,这些蛋白会迅速降解。一旦重组形成功能性蛋白酶,它们通过切割自身N节点亚基中的降解域,从而稳定自身浓度。此外,每种功能性蛋白酶还能通过切割C节点的重组域,抑制其他类型的功能性蛋白酶。
这些设计特性共同实现了加权求和、相互抑制和自激活,从而支持胜者为王的分类行为。我们将这些系统称为Perceptein网络,类似于人工神经网络架构中的感知器。
建模与仿真
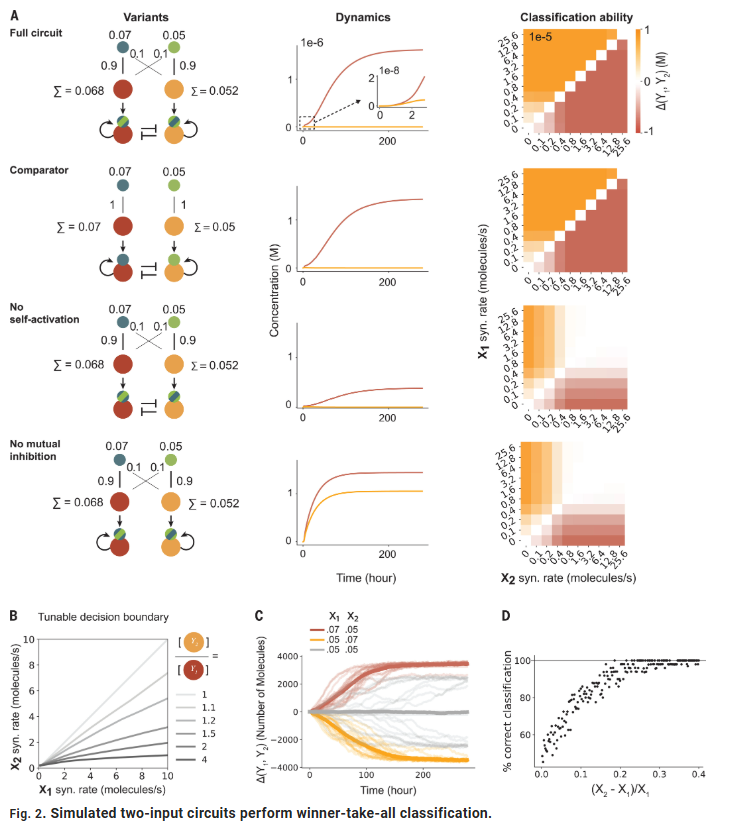
研究人员通过简化的蛋白质相互作用模型对系统进行仿真。结果显示,该系统能够成功地实现胜者为王的分类行为:当一个输入值超过另一个时,相应的蛋白酶活性迅速累积,并在较短时间内形成明显差异。仿真还表明,分类行为适用于较宽的输入浓度范围,即使输入值差异仅为10%时,系统依然表现出准确的分类能力。
进一步的仿真测试揭示了电路中各特征的必要性:去除自激活特性会显著降低输出动态范围,甚至完全失去分类能力;去除相互抑制特性会加速响应,但输出不再呈现“全或无”的特征。此外,通过调控节点蛋白浓度,可以调整分类决策边界,使其对输入信号的差异表现出不同的敏感性。
实验验证
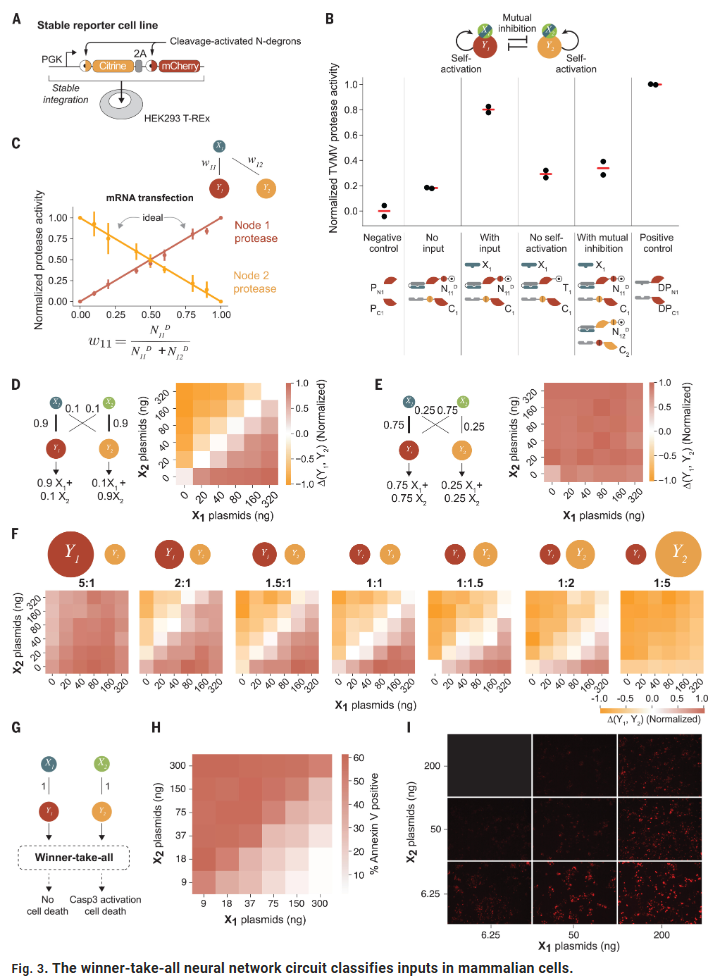
为验证Perceptein电路在哺乳动物细胞中的可行性,研究人员构建了完整的双输入双输出电路所需的蛋白质组件。我们选择了两种经过验证的分裂病毒蛋白酶(TEVP和TVMVP),并将其分裂半体与DHD结合域、蛋白酶切割位点和降解域融合,以控制功能性蛋白酶的重组。
研究人员在人胚胎肾293细胞(HEK 293)中建立了稳定的报告细胞系,包含两个带有降解标签的荧光蛋白,用于检测不同蛋白酶的活性。通过流式细胞术,研究人员验证了每种蛋白酶仅对其特异性目标起作用。这些构建体和报告细胞系使我们能够快速迭代测试电路设计,并确认Perceptein电路在哺乳动物细胞中实现了胜者为王的分类行为。
研究人员逐一验证了“胜者为王”神经网络的各个模块。首先测试输入信号是否能触发对应蛋白酶的重组活性。结果表明,当输入X1与其匹配的N节点和C节点蛋白共转染时,相应的荧光报告蛋白被抑制,符合蛋白酶重组的预期表现。在没有输入的情况下,报告蛋白水平与负对照(缺乏DHD结合域的分裂蛋白酶)相似,证实了输入触发的特异性蛋白酶活性。
接着,验证了输入加权步骤。通过调控不同比例的N11和N12 mRNA,同时保持输入X1恒定,我们发现重组蛋白酶的活性与N节点蛋白的相对丰度成线性关系,这符合模型预测。
自激活与相互抑制模块也通过实验进行了验证。对带有可切割降解标签的N节点蛋白进行转染,流式细胞术结果表明,具备降解位点的N节点能够通过蛋白酶切割实现自我稳定,而不具备该位点的负对照组活性接近背景水平。此外,过量的Y2节点组分显著抑制Y1节点的蛋白酶活性,验证了相互抑制的有效性。
接着,研究人员测试了完整电路。通过向报告细胞共转染输入蛋白和节点蛋白,观察到,当输入X1超过X2时,Y1节点活性显著高于Y2;反之亦然,且输入相等时输出最小。这一结果与模拟预测一致,表现出类似于人工神经网络输出层的Softmax函数行为。此外,通过调节节点蛋白的丰度,可以实现决策边界的可调节性。
当输入数量等于节点数量时,进一步简化电路,去除交叉相互作用,构建了一个更简单的比较器。实验结果显示,该简化电路依然保留了输入分类的能力,并与仿真结果高度一致。同时,电路对输入时机的变化具有鲁棒性,晚24小时加入输入仍能产生相同的分类结果。
可扩展的分类能力
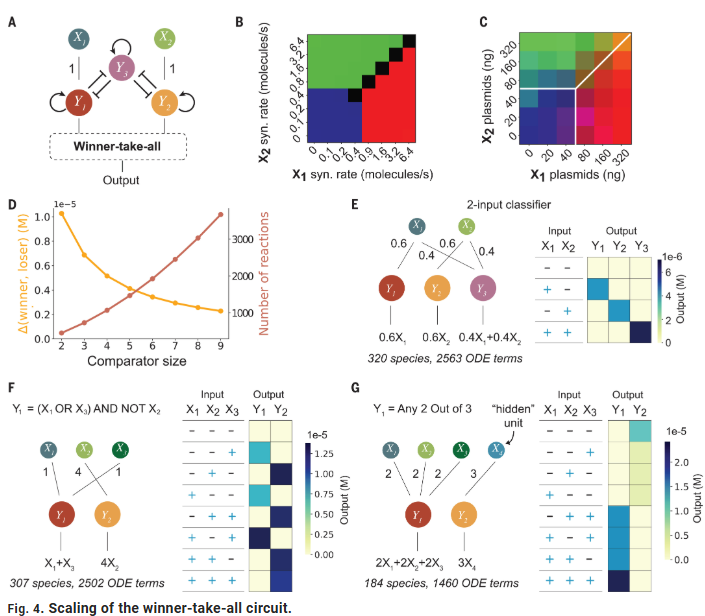
神经网络的关键特征在于其可扩展性。研究人员通过增加一个互相抑制的节点,将分类任务扩展为三种输出状态。实验中,引入了HRV3C蛋白酶作为第三节点,并在稳定细胞系中实现了三类输入分类,结果与模拟预测一致。这证明了随着正交蛋白酶的增加,Perceptein网络可以扩展其分类能力。
进一步的仿真研究表明,Perceptein系统可以扩展至更高维度,处理m个输入与m个节点的分类任务,且化学反应复杂度与网络规模线性增长。此外,该系统还能够执行更复杂的分类功能。例如,通过增加一个节点,系统可实现单层电路中需要多层逻辑门才能完成的复合函数(如**(X1 OR X3) AND NOT X2**)。通过添加“隐藏”单元,还可以实现更复杂的阈值计算,如“任意两个输入激活”功能。
讨论
早期的合成生物学主要将计算机科学中的数字逻辑范式引入生物系统。然而,自然界中的生化网络往往表现为密集的多对多相互作用,类似于神经网络。这引发了一个问题:是否能够通过合成方法构建受神经网络启发的生化电路?在本研究中,研究人员证明,通过全新设计的蛋白质域与工程化酶相结合,可以在活细胞中实现经典的胜者为王神经网络分类,并实现决策边界的可调控性。
在Perceptein设计中,系统运行过程中产生了大量不同的蛋白质种类与复合体(例如,两输入两输出电路产生158种分子),但这些复杂的分子被有效“压缩”成数量较少的起始蛋白种类(例如,两输入两输出电路中仅8种起始蛋白)。这一过程类似于某些蛋白家族通过可变剪接、蛋白质水解或组合组装多聚复合体等方式,产生丰富多样的蛋白质产物。
尽管本研究展示了基本的神经计算功能,但仍存在一些局限性。首先,分类结果未能达到完全数字化的输出,可能与瞬时转染方法导致的表达变异性有关。此外,网络规模目前受到可靠工程化蛋白酶数量的限制,以及大规模蛋白质表达控制的技术难题。最后,将完整电路(或比较器)扩展到更复杂的计算任务,组件数量将呈线性或倍增增长。
要在细胞内实现大规模的计算电路,未来需要具备以下能力:构建具有可编程特异性的大量酶变体、改进多基因表达系统的工程化方法,以及引入更高效的自然启发式架构。尽管如此,随着基因组工程技术的不断进步,神经电路架构有望进一步扩展活细胞的计算能力,实现更大规模、更复杂的合成生物系统。
整理 | WJM
参考资料
Chen, Zibo, James M. Linton, Shiyu Xia, Xinwen Fan, Dingchen Yu, Jinglin Wang, Ronghui Zhu, and Michael B. Elowitz. "A synthetic protein-level neural network in mammalian cells." Science 386, no. 6727 (2024): 1243-1250.
内容中包含的图片若涉及版权问题,请及时与我们联系删除

