DRUGAI
今天为大家介绍的是来自Wenfei Li团队的一篇论文。蛋白质通过运动来实现其生物功能。尽管利用基于深度学习的方法高通量预测蛋白质的三维静态结构已被证明是可行的,但预测其构象运动仍然是一个挑战。纯粹依赖数据驱动的机器学习方法难以解决此类运动问题,因为目前关于蛋白质构象运动的实验数据仍然有限。在这项研究中,作者开发了一种方法,将物理能量景观信息整合到基于深度学习的方法中,以生成蛋白质的变构运动。作者证明了局部能量挫折(即能量景观中的局部特征量化)可以用于增强AlphaFold2 (AF2) 在预测蛋白质构象运动方面的能力。从基态的静态结构出发,这种整合方法通过在输入的多序列比对中逐步增强能量挫折特征,生成蛋白质构象运动的替代结构和运动路径。以腺苷酸激酶为模型蛋白,作者展示了所生成的构象运动与现有的实验数据和分子动力学模拟数据相一致。将该方法应用于涉及大幅度构象变化的其他两种蛋白质KaiB和核糖结合蛋白时,也成功地生成了替代构象。作者还展示了如何提取AF2能量景观拓扑结构的整体特征,这被许多人视为一个黑箱。将物理知识纳入基于深度学习的结构预测算法,提供了一种解决变构蛋白动态结构预测挑战的有效策略。

AF2利用先进的transformer架构,从序列中预测蛋白质结构,其准确性接近实验结果。AF2最初设计用于提供单一预测结构,但它为更广泛的应用提供了基础。最近的研究开始深入探索AF2在预测蛋白质的其他构象、动态、功能和突变效应方面的能力,充分利用从多序列比对(MSA)中获得的丰富进化信息。Jussupow和Kaila的研究显示,AF2给出的统计预测分数与通过分子动力学(MD)模拟采样的蛋白质内在构象波动之间存在很强的相关性。Roney和Ovchinnikov也证明,AF2已经有效地学习了一种基于信息的能量函数,可以用于排序输出蛋白质预测的质量。这些研究突显了AF2在利用MSA的进化数据来揭示蛋白质能量景观方面的潜力。
此外,还成功地利用浅层MSA和其他管道来引导AF2预测替代结构。这些研究表明,通过限制MSA深度或屏蔽序列中某些位置的氨基酸身份来减少MSA信息,能够作为AF2预测结果的扰动,从而获得替代结构。最近,Wayment-Steele等人展示了通过聚类具有高度序列相似性的MSA,AF2可以用高置信度预测多种构象状态。
AF2常被视为一个复杂的“黑箱”,其内部过程大多未被深入研究。然而,过去几十年中,人们对控制蛋白质折叠和变构运动的物理原理已有了较为成熟的理解。最小挫折(minimal frustration)原理揭示了动力学和热力学约束是如何在可折叠蛋白质的序列中编码的。进化塑造了一个整体漏斗形的能量景观,但偏离完美漏斗的部分通常会编码功能。残基水平的局部挫折定位了蛋白质变构运动的关键节点。酶的催化路径也依赖于局部挫折的特定空间分布模式。
将AF2与蛋白质能量挫折分析结合的流程步骤
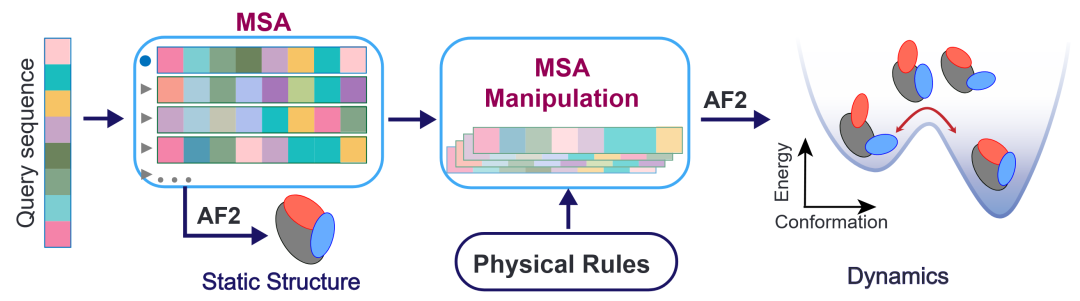
图 1
通过利用AF2在使用MSA进化数据方面的能力,本文介绍了一种方法,将AF2的结构预测能力与蛋白质能量挫折分析相结合,以预测蛋白质构象运动(见图1)。“挫折”概念强调了相互冲突的相互作用在蛋白质折叠、动力学和功能中的作用,是解锁AF2更深层能力的关键。通过利用基于既有生物物理工具提取的变构蛋白的能量挫折模式,作者旨在扩展AF2的能力,不仅限于静态结构预测,还包括变构蛋白的动态特性,从而加深对基因型与表型映射的理解。
该方法不仅成功预测了一些典型变构蛋白的替代构象,还提供了预测的构象运动的动态路径信息。结合AF2和生物物理规则的这一计算方法,将能够有效深入地研究蛋白质的动态特性,可能对治疗策略有重要意义。
蛋白质能量景观的变构特征
虽然为了实现稳健的蛋白质折叠,需要一个漏斗形的能量景观,使天然结构中只有较少的能量冲突,但变构蛋白通常表现出局部有较高能量挫折的显著区域。这些挫折区域对于功能相关的构象运动至关重要。局部挫折指数用于衡量在天然结构中,两个残基之间的特定接触与所有其他可能随机接触相比,在能量上是多么有利。蛋白质中具有最低挫折相互作用的区域通常相对刚性,而高度挫折的区域则可能采用不同的构象,因此更具灵活性。局部挫折指数可以通过Ferreiro等人开发的Frustratometer工具轻松计算。
腺苷酸激酶(AdK)是一种多结构域蛋白,由LID结构域、NMP结构域和核心结构域组成(图2A),已成为研究变构的典型蛋白。它被用于说明局部能量挫折、构象运动和催化功能之间的相互作用。AdK在所有生命领域中起着重要的功能作用,通过可逆地催化三磷酸腺苷(ATP)与一磷酸腺苷(AMP)之间的磷酸基团转移,生成两个二磷酸腺苷分子,从而维持细胞内ATP水平。在酶促反应周期中,这种激酶在开放和闭合构象之间进行大幅度的构象转换,涉及多个中间结构。研究表明,局部能量挫折通过促进特定的构象运动,在塑造催化路径中起到了关键作用。在本文中,作者也将使用这种酶作为模型,展示如何利用局部能量景观特征,通过AF2预测蛋白质的构象运动,这些特征是通过局部能量挫折评分量化的。
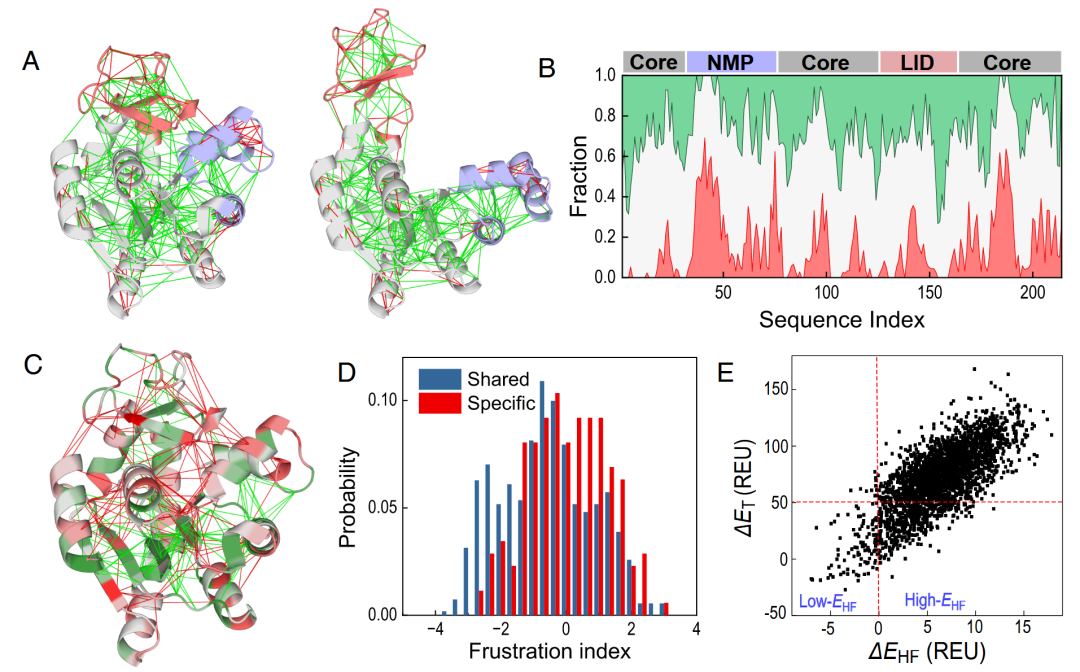
图 2
图2A展示了腺苷酸激酶(AdK)在闭合和开放构象中的局部挫折模式。使用Frustratometer计算的成对挫折指数。正如Ferreiro等人在之前的研究中观察到的那样,AdK结构域内部富含最小挫折的相互作用(绿色线),形成了密集的网络连接。相反,结构域界面和铰链区域的相互作用通常表现出较高的挫折(红色线)。沿AdK序列,各残基周围高挫折接触的密度呈现不连续的分布(图2B)。为了更清晰地展示挫折模式的关键特征,作者还计算了平滑挫折得分,其中和是由Frustratometer给出的残基挫折指数。因此,高的接触对应于高挫折位点的界面。可以看到,在AdK的闭合结构中,结构域界面富含高值的接触(图2C,红线),这似乎是促进结构域运动的重要能量特征。作者进一步计算了两类接触的平滑挫折得分分布,即共享接触和特定接触。如果接触在两种结构形式中均完全形成,则将其归类为“共享接触”;如果接触仅存在于一种结构中,则归类为“特定接触”。在构象变化过程中,这些特定接触需要在结构域运动时断开,通常仅位于闭合结构的结构域界面。对于Cα距离小于10埃且序列间隔大于8的残基对(i, j),考虑其作为共享和特定接触并计算。如图2D所示,特定接触比共享接触更容易产生挫折,这与先前关于挫折与变构之间联系的观察结果一致。对变构蛋白的调查显示,AdK观察到的这种蛋白能量景观特征对于其他变构蛋白也是普遍的。有趣的是,Raisinghani等人最近的研究显示,ABL激酶中低丰度的不活跃构象具有大量高挫折残基簇的特点,这往往会增加AF2结构预测的难度。
通过挫折过滤利用AF2预测变构蛋白的替代结构
对于大肠杆菌腺苷酸激酶(AdK),输入完整的MSA序列集后,AF2会返回一个闭合构象的预测结构,与相应的晶体结构非常相似。基于该预测结构计算的能量挫折也显示出与晶体结构几乎相同的模式。接下来,作者将展示如何利用这些局部挫折信息来迫使AF2预测蛋白质的构象运动。考虑到MSA序列包含的共同进化信息为结构预测提供了线索,作者使用特定参考结构上MSA序列的能量挫折特征来引导AF2生成替代结构。
如前所述,从闭合状态到开放状态的AdK构象转变涉及到特定接触的破坏,这些接触通常表现出高挫折特性。可以引导AF2模拟与这些高挫折位点配置相关的结构动态。首先,将MSA序列排列到AdK的闭合结构上,并使用Rosetta能量函数计算所有天然接触的总能量(ET)以及高挫折接触的能量(EHF)。然后,可以根据二维“进化”反应坐标和(见图2E)对MSA序列进行分类。这些坐标描述了给定序列相对于参考大肠杆菌AdK序列的整体稳定性和局部挫折程度。ΔEHF较高的序列往往在这些高挫折位点表现出更明显的局部挫折,这些位点通常涉及特定接触(见图2D)。
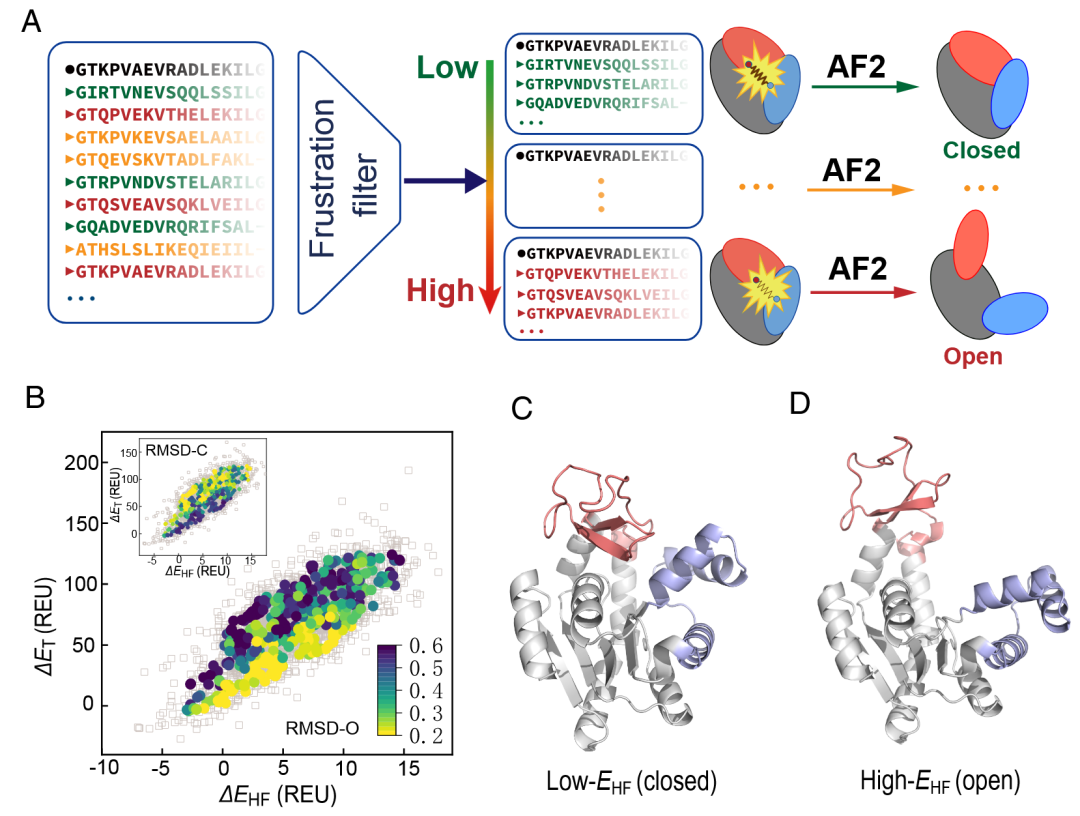
图 3
可以合理地假设,在AF2结构预测中,对于这些特定接触能量较低的序列,将更可能呈现出闭合构象(见图3A)。相反,对于特定接触能量较高的序列,更可能偏向于开放结构,因为这些接触变得不稳定。因此,通过利用局部能量挫折特征来调整MSA,作者能够使用AF2生成闭合和开放的结构。
基于这种挫折过滤的物理图景,作者引入了一种能量阈值方法,用于对MSA序列进行子采样,并预测变构蛋白的替代构象。作者分别从二维能量空间的不同区域抽取序列,并仅将这些序列与输入的查询(query)序列一起提供给AF2进行结构预测。从每个区域选取50个MSA序列用于结构预测。显著的是,AF2主要为低的MSA序列返回闭合结构,为高ΔEHF的序列返回开放结构,且在每种情况下的置信度都很高(图3B)。
特别是,当使用最低的子采样MSA序列(500个序列)时,AF2返回一个几乎完全闭合的结构,其与闭合状态的晶体结构相比,RMSD为0.80埃(PDB代码:4AKE)。同样,当仅使用高和低的MSA序列(50个序列)时,AF2生成的开放结构与开放状态的晶体结构相比,RMSD为1.63埃(图3C和D)。相应地,生成开放结构的MSA序列往往具有较高的值和降低的特定接触能量。由此可以看出,局部能量挫折的信息可以用来引导AF2预测变构蛋白的有意义的替代结构。作为对照,作者还进行了额外的测试计算,遵循相似的流程,但将挫折评分随机打乱。当挫折评分被打乱时,MSA的能量特征与预测结构的构象之间不再有对应关系。
编译 | 黄海涛
审稿 | 曾全晨
参考资料
Guan, X., Tang, Q. Y., Ren, W., Chen, M., Wang, W., Wolynes, P. G., & Li, W. (2024). Predicting protein conformational motions using energetic frustration analysis and AlphaFold2. Proceedings of the National Academy of Sciences, 121(35), e2410662121.

